introduktion
internationell bevarandepolitik erkänner biologisk mångfald på tre nivåer, ekosystem, arter och genetiska, och att förvaltningen bör syfta till att behålla alla tre (konvention om biologisk mångfald, 2007). Detta återspeglas tydligt i konventionen om biologisk mångfald, Aichi Biodiversity Targets, som antogs 2010 (https://www.cbd.int/sp/targets/), där det finns specifik hänvisning i mål och mål, inte bara till ekosystem och arter utan också till genetisk mångfald. Men nuvarande metoder för bevarande av biologisk mångfald bygger till stor del på geografiska områden, ekosystem, ekologiska samhällen och arter, med mindre uppmärksamhet på genetisk mångfald och Art-populationskontinuum. Faktum är att majoriteten av bevarandeutövare fokuserar på arter som mått för biologisk mångfald, med viss hänsyn till intraspecifika enheter inom intensivt hanterade hotade arter. I detta sammanhang finns det en naturlig önskan att använda enkla kategoriseringar—namngivna arter och andra namngivna taxa (dvs. underarter) och ibland bevarandeenheter inom arter. Särskilt arter ger ett gemensamt mått på biologisk mångfald som ligger till grund för mycket vetenskaplig och förvaltningsarbete (Mace, 2004; Wilson, 2017). Som en grundläggande enhet för bevarande används arter ofta för att kvantifiera värdet av biologisk mångfald genom listor över arter inom skyddade områden, identifiering av hotade arter inom jurisdiktioner och som grund för biosäkerhetsförfaranden. Dessutom är arter vanligtvis de enheter med vilka vi kommunicerar bevarandefrågor till allmänheten.
denna drivkraft för att helt enkelt identifiera diskreta enheter i naturen strider mot flera relaterade verkligheter. För det första är speciering inte ögonblicklig. Snarare är det vanligtvis en långvarig process (Dynesius och Jansson, 2014; Roux et al., 2016), som går från isolerade populationer, kanske utsatta för olika urvalstryck, till entydiga arter över långa tidsskalor, med frekventa omkastningar orsakade av miljöförändringar eller enkel genetisk sammanslagning när intervall återansluts (Figur 1; Seehausen et al., 2008; Rosenblum et al., 2012). För det andra är de flesta erkända arterna inte genetiskt enhetliga. Divergens (drift och långvarig isolering) och retikulering (geneflow, hybridisering, introgression och rekombination) formar effektivt geografisk och genomisk variation inom arter (Cutter and Payseur, 2013; Edwards et al., 2016) och vissa arter kan struktureras i långa isolerade populationer, varav några kan motivera erkännande som fulla men kanske morfologiskt kryptiska arter (Bickford et al., 2007; Jorger och Schr Auddl, 2013). För det tredje, så länge erkända, namngivna arter är inte likvärdiga när det gäller hur mycket evolutionärt arv de representerar—enkelt uttryckt kan en platypus (som representant för en gammal divergerad släkt) anses vara värd mer än en nyligen divergerad gnagare (Isaac et al., 2007). För det fjärde kastas inte artbeteckningar i sten, utan de är hypoteser som ska testas när nya bevis blir tillgängliga (Hej et al., 2003). Var och en av dessa frågor skapar komplexitet och tvetydighet i att avgränsa arter i allmänhet och tilldela bevarandeuppmärksamhet åt dem eller deras komponentpopulationer. Dessutom är avgränsningen av arter också föremål för olika artbegrepp och sätt att diagnostisera dem, vilket har orsakat stor bestörtning bland bevarandebiologer (Mace, 2004; Frankham et al., 2012; Garnett och Christidis, 2017).
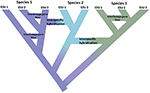
Figur 1. Speciering som ett kontinuum som utvecklas från isolerade populationer till entydiga arter som visas av färgskiftet med färgerna som representerar olika arter. Vissa linjer som uppstår på grund av befolkningsisolering utvecklas till Sanna arter med trädets inre noder som representerar initiering av specieringsprocessen. Andra släkter utvecklas inte till Sanna arter men skiljer sig över tiden vilket indikerar att arter inte är genetiskt enhetliga. Dessa linjer visas som evolutionärt signifikanta enheter (esu). Inter-lineage genflöde är indicerat för Art 1 och Art 3. I detta fall kan evolutionärt oberoende vara övergående eftersom två linjer som tillhör samma art inte är reproduktivt isolerade. Medan de förblir som distinkta linjer (arter) kan interspecifik hybridisering förekomma med begränsat genutbyte (färgskift) mellan arter.genomförandet av effektiva bevarandestrategier som syftar till att skydda både arter och genetisk mångfald skulle dra nytta av både en mer konsekvent syn på hur vi avgränsar arter och andra enheter över hela populationsartens kontinuum och hur dessa enheter tolkas i olika lagar globalt. Nya möjligheter från genomics ger både möjligheter och utmaningar för att uppnå dessa mål. I det följande anser vi: (i) hur genomskaliga analyser kan förenas med ett konservativt tillvägagångssätt för avgränsning och namngivning av arter, samtidigt som man identifierar viktiga komponenter i genetisk mångfald inom arter, (ii) hur befintlig bevarandelagstiftning över flera kontinenter, som definierar ”arter” för att inkludera ett brett spektrum av infraspecifika kategorier, kan användas med tanke på den höga upplösningen som erbjuds av genomskärmar, och (iii) hur insikterna om specieringsprocesser och strukturering av variation från genomik överensstämmer med lagstiftningen och sedan till förvaltningen. Vi föreslår sedan några sätt framåt i att anpassa bevarandeenheter med lagstiftningsmetoder för att skydda hela spektrumet av mångfald från populationer genom arter.
genomik, speciering och taxonomi
liksom många områden inom biologisk vetenskap, Systematik, praxis att namnge taxa och förstå deras relationer och hur de bildar, är betydligt avancerad av genomik och statistiska modeller. Vare sig genom hela genomsekvensering (t.ex. Nater et al., 2017) eller genomprovtagning (SNPs, Target capture-sekvensering; Jones och bra, 2016; Leache and Oaks, 2017), har vi nu oöverträffad upplösning av mönster av genetisk mångfald (Funk et al., 2012). Tillsammans med allt kraftfullare statistiska modeller för att dra historier om genomisk och arter divergens, dessa data ger nya insikter i de evolutionära processer som genererar arter och genetisk variation.
tillämpning av dessa nya verktyg genererar två motsatta insikter. Å ena sidan avslöjar många studier mycket divergerande genetiska populationer inom namngivna arter, så kallade kryptiska arter (Bickford et al., 2007; Jorger och Schr Auddl, 2013; Struck et al., 2017). Detta är inget nytt, eftersom det bygger på en lång historia av rumslig populationsgenetik och molekylär fylogeografi som har varit en produktiv strävan i tre decennier (Avise et al., 1987). Vad genomik ger till denna strävan är både ökad upplösning av befolkningsstrukturen och data som är tillräckliga för att statistiskt dra slutsatsen befolkningshistorier och testa alternativa modeller av divergens (Degnan och Rosenberg, 2009; Sukumaran och Knowles, 2017). Å andra sidan avslöjar genomskalanalyser också att (så länge erkänts för växter) är genetisk utbyte (introgression) bland djurarter vanligare än tidigare trott, både under och efter speciering, och kan till och med driva nya adaptiva strålningar (Mallet, 2007; Rieseberg och Willis, 2007; Arnold och Kunte, 2017) Figur 1. Tillsammans betonar dessa insikter vidare att speciering är långvarig (Roux et al., 2016) och avslöja de motsatta krafterna som arbetar genom fylogeografi-fylogenetik kontinuum (Edwards et al., 2016).
hur ansluter allt detta till taxonomi och bevarandemål? Det finns en stark och motiverad oro för att den ökade upplösningen som genomskärmarna ger kan leda till överdriven splittring av arter (Isaac et al., 2004), som potentiellt begränsar ledningsflexibiliteten och sänder små genetiskt divergerande populationer till inavel och eventuell utrotning (Frankham et al., 2012). Omvänt, Gippoliti et al. (2018) hävdar att taxonomisk tröghet, vilket resulterade i tidigare misslyckande med att känna igen många arter av afrikanska hovdjur, hade resulterat i avelsdepression efter blandning och misslyckandet med att skydda geografiskt begränsade taxa. Deras syn (och se även Morrison et al., 2009) är att det inte finns några bevis för att taxonomisk inflation har negativa effekter på hovskydd (Gippoliti et al., 2018). En relaterad oro är att instabil taxonomi fördröjer både utövandet av bevarande och offentliga uppfattningar (Garnett och Christidis, 2017). Dessa farhågor drivs delvis av den långvariga debatten kring artbegrepp och diagnostiska metoder inom Systematisk biologi, särskilt tillämpning av det fylogenetiska artbegreppet som hävdar att arter är minimalt diagnostiserbara enheter (Cracraft, 1983). Majoriteten av taxonomerna är emellertid i sig konservativa, bara namngivna arter när det finns överensstämmelse över flera bevislinjer (Dayrat, 2005; Padial et al., 2010; Schlick-Steiner et al., 2010: Yeates et al., 2011). Vår uppfattning är att arter ska representera robusta oberoende utvecklande linjer som förblir i stort sett intakta vid kontakt med nära släktingar—i enlighet med avsikten med Evolutionary Species Concept (Wiley, 1978) och Generalized Lineage Species Concept (de Queiroz, 1998, 2007). I praktiken kräver detta direkta bevis på reproduktiv isolering eller rimliga surrogater av samma, samtidigt som det möjliggör begränsad genetisk utbyte (Singhal et al., 2018).
genomiska data ger större kraft än tidigare för att identifiera differentiering och divergens inom och mellan arter, vilket kräver större hänsyn till hur detta påverkar avgränsningen av taxonomiska enheter. Där populationer är både starkt genetiskt och fenotypiskt divergerande i förhållande till redan namngivna arter, finns det ett starkt fall för att namnge dessa som separata arter. Om populationer är starkt divergerande fenotypiskt med liten genomisk separation, finns det en bedömning om huruvida dessa fenotypiska skillnader är (i) ärftliga och (ii) sannolikt att orsaka materiell reproduktiv isolering. Genomskanningar kan lösa sådana situationer genom att testa för begränsat genflöde. Den mer utmanande frågan är där morfologiskt liknande, men genetiskt divergerande populationer upptäcks. Sådana populationer kan lätt diagnostiseras som separata evolutionära linjer genom att tillämpa statistiska avgränsningsmetoder på multilocus-data (Carstens et al., 2013; Rannala, 2015), men är de flyktiga populationer eller hållbara arter (Sukumaran och Knowles, 2017)? Det är verkligen möjligt, inom nomenklaturens regler, att känna igen arter från DNA-sekvenser ensam, vilket ger sekvensbaserad diagnostik (Tautz et al., 2003; Cook et al., 2010). Till exempel Murphy et al. (2015) avgränsa och beskriv sex australiska ökenfjäder amphipodarter baserat på DNA-sekvensdata trots inga tydliga morfologiska skillnader. Detta är dock ett allmänt förbättringsområde där systematister måste tillämpa termen ”kryptiska arter” med större konsistens och rigor (Struck et al., 2017).
i situationer där DNA-sekvensdata ger en potentiell nyckeldefinierande funktion för erkännande av en art, föreslår vi att det är särskilt viktigt att tillämpa andra bevislinjer för att undvika överlappning (Oliver et al., 2015). Ett tillvägagångssätt är att testa för betydande reproduktiv isolering där sådana populationer bildar naturliga kontaktzoner. Till exempel, i en jämförande genomisk analys av kontaktzoner bland släktpar av Carlia ödlor som är fenotypiskt oskiljbara, var index för genetisk isolering starkt korrelerade med divergenstid (Singhal och Moritz, 2013; Singhal och Bi, 2017) (Figur 2). De mer divergerande av dessa härstamningspar har nu formellt erkänts som separata arter, medan närmare besläktade (men statistiskt diagnostiserbara) linjer med bevis på genomiskt omfattande genetisk utbyte över kontaktzoner inte var (Singhal et al., 2018). Med tanke på det starka empiriska förhållandet mellan divergens och reproduktiv isolering i dessa taxa separerade dessa författare ytterligare två allopatriska och kryptiska isolat (av en nära besläktad art) som hade ännu högre genomisk divergens än de avgränsade ovan. Utvidga denna reproduktiva isolering informerade metriska till andra kongeneriska arter med djupt divergerande men morfologiskt liknande fylogeografiska linjer (Potter et al., 2016, 2018; Afonso Silva et al., 2017a) föreslår att fler arter återstår att beskrivas, särskilt på öar, (Figur 2, Carlai amax och C. rufilatus), och faktiskt har vissa sådana arter beskrivits efter komplementär fenotypisk analys (Afonso Silva et al., 2017b). Andra linjer med nyare skillnader, men fortfarande statistiskt avgränsade som separata linjer med hjälp av en stor uppsättning kärngener, kan representeras som evolutionärt signifikanta enheter (ESUs) inom arter.
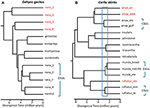
Figur 2. Fylogenomisk upplösning av artgränser i ödlor från de australiska Monsoonala tropikerna (AMT). I varje fall beräknas relationer och relativa divergenstider från 100 exoner med hjälp av multispecies coalescent implementerad i StarBEAST2, men tidsskalorna mellan de två exemplen är inte nödvändigtvis desamma. (A) resultat för nana-gruppen av Gehyra geckos (Moritz et al., 2018). Den fylogenetiska mångfalden av linjer inom G. nana, som tidigare erkänts, inkluderade fyra andra redan beskrivna och morfologiskt distinkta arter. Efterföljande analyser av intervallöverlappningar och morfologi har lett till erkännande av var och en av de mest fylogenetiskt divergerande linjerna (röda taxon-etiketter) som separata arter (Doughty et al., 2018). De (morfologiskt oskiljaktiga) linjer som finns kvar i G. nana som nu erkänns kan betraktas som ESUs. (B) pågående upplösning av artgränser i Carlia skinks från AMT. Vissa linjer har redan erkänts som arter baserade på en kombination av genetisk artavgränsning och post hoc-identifiering av diagnostiska morfologiska skillnader (C. insularis och C. isostriacantha; Afonso Silva et al., 2017b). För andra komplex (Potter et al., 2016, 2018), taxonomiska revisioner pågår. Den blå rektangeln indikerar intervallet av divergenstider där betydande reproduktiv isolering, korrelerad med divergens, har observerats i kontaktzoner mellan morfologiskt kryptiska linjer av besläktade arter (Singhal och Bi, 2017). Konservativt, och följande (Singhal et al., 2018), vi känner igen som kandidatart tre genetiska linjer med skillnader större än denna empiriska tröskel. Detta skulle lämna genetiskt distinkt ESUs inom var och en av Carlia amax, Carlia munda och Carlia rufilatus (blå parentes). Skalstängerna representerar uppskattningar av divergenstid på miljontals år.
i extrema fall avslöjar genomiska metoder enstaka taxonomiska arter för att inte bara vara genetiskt heterogena utan i fylogenetisk mening också innehålla andra namngivna arter. Sådan” paraphyly ” kan uppstå om den senaste arten från en enda, genetiskt uppdelad Art (Patton och Smith, 1994), men det genererar också hypotesen att det genetiskt heterogena taxonet kan representera flera arter. Återigen krävs mer bevis. Som ett typexempel avgränsade fylogenomisk analys av ett komplex av Gehyra gecko ödlor från norra Australien nio evolutionära linjer, med fyra redan namngivna arter fylogenetiskt inbäddade bland dem (Moritz et al., 2018; Figur 2). Efterföljande bevis på geografisk överlappning (samtidigt som man behåller fylogenetisk separation) och subtila morfologiska skillnader stödde namngivning av de fyra mest fylogenetiskt divergerande linjerna (Doughty et al., 2018). Omvänt hade de närmare besläktade (men statistiskt separerbara) linjerna intilliggande snarare än överlappande fördelningar, men var inte morfologiskt diagnoserbara och behölls följaktligen som en enda art (G. nana sensu stricto), om än med flera esu, i avvaktan på ytterligare undersökning (Figur 2). Efterföljande genomskanningar av prover över gränserna för dessa esu avslöjade att åtminstone vissa har betydande genetisk utbyte, validera den ursprungliga, konservativ strategi som vidtagits för att erkänna arter.
i andra fall kan växtgenomiska studier, även genom enkel ordination av SNP, avslöja starka och oförutsedda skillnader mellan populationsprover, vilket tyder på närvaron av kryptiska taxa som sedan kan analyseras för diagnostiska fenotypiska tecken. Till exempel visade SNP-baserade bevis för genomisk divergens mellan populationer och ekotypiska skillnader i Eucalyptus salubris två distinkta molekylära linjer som behöll sin särskiljningsförmåga inom ett område med geografisk överlappning, med ekotypisk anpassning anses vara en viktig faktor för att minimera genflödet mellan linjerna. (Steane et al., 2015; Figur 3). I ett annat fall, i australiensisk Pelargonium, genomisk analys, kombinerat med morfologiska och reproduktiva studier, har lett till betydande förändringar i taxonomi, både splittring och klumpning av taxa, särskilt där tidigare använda morfologiska egenskaper visar sig vara labila och disharmoniska med fylogeni (Nicotra et al., 2016).
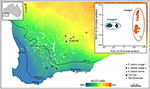
Figur 3. Genomisk analys med SNP-data identifierade kryptiska linjer i den utbredda Eucalyptus salubris. Placeringen av prover populationer av varje härstamning visas över en aridity index gradient i sydvästra västra Australien. PCoA baserat på binära genetiska avstånd härledda från 16,122 DArTseq-markörer, visar två distinkta linjer. Anpassad från Steane et al. (2015).
ovanstående fall illustrerar den progressiva hypotesprovningsmetoden som har använts i integrativ taxonomi (Padial et al., 2010; Yeates et al., 2011). Vidare använder dessa fall någon form av genomreduktionssekvensering för att erhålla högkvalitativa data från 1000-tals oberoende gener, vilket ger mycket större förtroende för resultaten än vad som är fallet med bara plastid loci eller några kärngener. Ett exempel på hur tillämpning av integrativ taxonomi dramatiskt kan förändra uppfattningar om mångfald kommer från en kombinerad genetisk, akustisk och morfologisk undersökning av grodor från Madagaskar som nästan fördubblade den kända artens mångfald (Vieites et al., 2009). Dessa författare föreslog en serie termer som återspeglar tillståndet för taxonomisk upplösning och kongruens: Bekräftade Kandidatarter, för genetiskt och fenotypiskt divergerande, men ännu inte namngivna taxa; obekräftade Kandidatarter för genetiskt divergerande taxa där morfologi och akustik ännu inte hade undersökts; och djupt specifika linjer för enheter som är kända för att vara genetiskt divergerande men fenotypiskt kryptiska. Genomiska analyser är ännu inte tillämpade på dessa taxa, men skulle säkert informera beslut om specifika linjer mot kandidatarter.
genomiska tillvägagångssätt, även när de tillämpas konservativt, kommer utan tvekan att leda till ytterligare splittring av morfologiskt konservativa taxa. Men omvänt finns det tydliga exempel där samma tillvägagångssätt avslöjar överdelade eller felaktigt diagnostiserade arter (Nikotra et al., 2016). Naturligtvis finns det redan många molekylära studier på växt-och djurartskomplex baserade på mindre datamängder (t. ex., microsatellite, mtDNA och cpDNA) som har identifierat var arter har feldiagnostiserats eller där flera arter har avgränsats taxonomiskt men nu anses vara en enda art. Till exempel Rabosky et al. (2014) använde mitokondriella och nukleära genfylogenier för att revidera scincid Ctenotus inornatus-komplexet, vilket resulterade i en nettominskning av arter och omjustering av artgränser; de morfologiska egenskaper som tillämpades tidigare, medan de var lämpliga för fältbiologer, var mycket labila och misslyckades till stor del med att matcha artgränser som diagnostiserade genetiskt. I växter finns några av de mest kända exemplen i orchids, såsom European orchid genus Ophrys och Australian orchid genus Caladenia, där molekylära studier indikerar betydligt färre enheter som skulle erkännas som arter än de som bygger på morfologiska egenskaper och pseudokopulatoriska pollineringssyndrom (se Devey et al., 2008; Swarts et al., 2014).
Genomskaladata, när de tolkas konservativt och idealiskt med parallella analyser av fenotyper, kommer att fortsätta att leda till taxonomiska förändringar, särskilt i morfologiskt konservativa taxa (Fi kuber et al., 2018). Men i motsats till oro som uttrycks av vissa (Garnett och Christidis, 2017) anser vi att detta är den rätta strävan efter evolutionär Systematik, även om resultaten är tillfälligt obekväma.
genomik och intraspecifika Bevarandeenheter
Även om mindre omtvistad än taxonomisk avgränsning och artnivådebatten, men på många sätt lika komplex, har varit den betydande diskussionen om definitionen och avgränsningen av bevarandeenheter som viktiga delar av intraspecifik mångfald som behöver beaktas i bevarandeåtgärder (Ryder, 1986; Waples, 1991; Dizon et al., 1992; Moritz, 1994; Vogler och de Salle, 1994; Crandall et al., 2000; Fraser och Bernatchez, 2001; Avise, 2004; Funk et al., 2012) (Figur 4). Med tanke på det försiktiga tillvägagångssättet att erkänna arter från genomiska bevis som vi förespråkar ovan (och se även Oliver et al., 2015; Singhal et al., 2018) är det oundvikligt att vissa taxonomiska arter fortfarande kommer att innehålla långa isolerade populationer inom dem. Begrepp som evolutionärt signifikanta enheter (esu) eller Designerbara enheter (du) har föreslagits för att komplettera befintlig taxonomi (Ryder, 1986; Waples, 1991; Moritz, 1994; Mee et al., 2015). Som med arter varierar tolkningar och kriterier, men den gemensamma avsikten är att erkänna viktiga delar av intraspecifik mångfald för bevarandeåtgärder (Funk et al., 2012). Moritz (1994, 2002) förespråkade ett förenklat men praktiskt molekylärt tillvägagångssätt för att identifiera oberoende utvecklande segment inom arter, den ”historiska” mångfaldens axel; medan andra lägger större vikt vid den ”adaptiva” axeln för att inkludera funktionell mångfald (Ryder, 1986; Crandall et al., 2000; Fraser och Bernatchez, 2001; Waples et al., 2001; Mee et al., 2015) (Figur 4). Oavsett de relativa fördelarna med dessa alternativ är det uppenbart att de tekniska och analytiska verktygen för befolkningsgenomik nu robust kan identifiera båda komponenterna i intraspecifik mångfald (Funk et al., 2012; Hoban et al., 2013; Whitlock, 2014; Yang och Rannala, 2014).
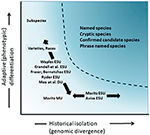
Figur 4. Separation av genetisk mångfald i två komponenter: adaptiv variation som uppstår genom naturligt urval och analyseras genom analys av fenotyper och historisk isolering eller neutral divergens bedömd genom genomisk divergens. Det mörkare skuggade området ovanför kurvan indikerar förhållanden där populationer sannolikt kommer att betraktas som separata arter under de flesta begrepp. Olika bevarandeenhetskoncept och infra-specifika taxonomiska kategorier visas i förhållande till de två axlarna för genetisk mångfald (efter Moritz, 2002).
detta leder till frågan: varför inte bara namnge ESUs som underarter? Underarter har traditionellt definierats och betraktats av både botaniker och zoologer som fenotypiskt distinkta, allopatriska populationer som kan intergradera in i varandra vid geografiska gränser (Mayr, 1942; Grant, 1981). Som sådan återspeglar de potentiellt adaptiv divergens utan att nödvändigtvis ha varit historiskt isolerade. Underarter beteckningar används ofta i växter, med geografiska och ekologiska skillnader det primära sättet genom vilket de skiljer sig (Hamilton och Reichard, 1992). I den mån underarter uppvisar lokal anpassning, med eller utan pågående genflöde, föreslår vi att fenotypiskt definierade underarter bör fortsätta att erkännas i bevarande och inte sammanfogas med ESUs, åtminstone inte ESUs som definieras för att representera historiskt isolerade populationer.
på samma sätt som populationsgenomik ger mycket större precision vid identifiering av ESUs genom att använda både neutrala och adaptiva loci ger det också ett sätt att använda ett stort antal neutrala loci för att lättare avgränsa förvaltningsenheter (MUs) som demografiskt oberoende enheter som kännetecknas av begränsat genflöde (Funk et al., 2012). Medan det mesta av vårt fokus här är på ESU: s och analoga enheter som DUs som kan erkännas som de viktigaste intra-specifika enheterna för bevarande och har accepterats för skydd under vissa jurisdiktioner, MUs har också identifierats som representerar demografiskt oberoende populationer (se Palsb Acill et al., 2007) som kan hanteras för att behålla den större ESU (Moritz, 1994; Funk et al., 2012). Förvaltningsenheter kan vara viktiga för att övervaka och hantera populationer av arter och har också kallats ”bestånd”, särskilt relevanta för fiskeriförvaltningen (Avise, 2004; Laikre et al., 2005). Definitionen av MUs av Moritz (1994) som ”populationer med signifikant divergens av allelfrekvenser vid kärn-eller mitokondriella loci, oberoende av allelernas fylogenetiska särskiljningsförmåga” är allmänt accepterad, även om, som påpekats av Palsb Acvisill et al. (2007), demografisk anslutning är nyckelfaktorn inte nivån på historiskt genflöde.
att avgränsa bevarandeenheter som viktiga delar av intraspecifik mångfald har också betydande konsekvenser för genetisk räddning av små inavlade populationer genom återställande av genflöde eller genom förstärkning och det blir allt tydligare att genetisk räddning måste övervägas bredare om ökad befolkningsutrotning ska avvärjas (Love Stowell et al., 2017; Ralls et al., 2018). Det föreslogs av Moritz (1999) att blandning av MUs men inte ESUs skulle kunna betraktas som en lämplig strategi för genetisk räddning. Som betonas av Frankham et al. (2012) blandning av taxonomiskt erkända arter skulle i allmänhet inte anses vara acceptabel av chefer, i vilket fall taxonomisk överlappning kan begränsa möjligheterna att rädda små inavlade populationer. Vi föreslår att även om ett mer rigoröst tillvägagångssätt för att avgränsa intraspecifika enheter som MUs kan hjälpa till att lindra detta problem, är det också uppenbart att genomisk uppskattning av migrationshastigheter och adaptiv divergens har potential att ge mycket mer robust vägledning när det gäller att inte bara definiera artgränser och intraspecifik variation, utan att utforma genetiska räddningsstrategier.
fylogenomik och bevarande
genomik kombinerat med fylogenetiska metoder kan lätt lösa relationer och evolutionärt avstånd mellan taxa, såväl som avgränsande arter. Detta uppnås bäst med hjälp av artträdmetoder, snarare än sammanfogning, eftersom den tidigare redogör för genträdets heterogenitet och undviker överinflation av spetslängder på grund av förfädernas polymorfism (Edwards et al., 2016; Ogilvie et al., 2016). Medan massiva antal loci ofta inte är nödvändiga, inklusive mer loci förbättrar ofta upplösningen av både relationer och grenlängder (t.ex. Blom et al., 2016). Vikten av bevarande är dubbelt. För det första gör fylogenetiska bedömningar det möjligt att överväga särdragen hos arter eller ESUs, inte bara om de beskrivs. Således EDGE-programmet (Isaac et al., 2007) förespråkar viktning av arter för bevarande baserat på hur mycket unik evolutionär historia de representerar, förutom vanliga IUCN-kriterier. För det andra kan grenlängderna själva representera biologiska mångfaldsvärden, oberoende av taxonomi (Faith, 1992), vilket har visat värde för att prioritera områden för bevarande (Laity et al., 2015; Rosauer et al., 2016, 2018). Genom att anta fylogenetiska mätvärden med hjälp av åtgärder som fylogenetisk mångfald och fylogenetisk endemism gör det möjligt att lyfta fram områden med hög evolutionär mångfald som kan riktas mot skydd och ges hög prioritet i alla planeringsprocesser för förbättrad bevarande av biologisk mångfald. Detta tillvägagångssätt kan förbättra skyddet av genetisk mångfald och evolutionära processer för olika grupper av organismer över en rad geografiska skalor (Laity et al., 2015) fatta beslut om områden för att prioritera mer robusta till vagaries av hur genomiska bevis översätts till erkännande av arter eller intra specifika enheter.
den verkliga världen—Lagstiftningsdefinitioner
politik och lagstiftningsramar för bevarande av biologisk mångfald sträcker sig från internationella konventioner och strategier, genom nationella strategier och lagstiftning, och statlig eller regional lagstiftning och strategier inom länder. De viktigaste internationella instrumenten för biologisk bevarande inkluderar konventionen om biologisk mångfald som genomförs genom parternas konvention (COP), världsarvskonventionen, den globala strategin för biologisk mångfald, Montrealprocessen för skogsvård, Ramsarkonventionen om våtmarker, konventionen om migrerande arter, konventionen om internationell handel med utrotningshotade arter (CITES), den internationella unionen för naturvård (IUCN) röda listan över hotade arter och FN: s ramkonvention om klimatförändringar. Alla dessa stadgar förkroppsligar principen att bevara arter och genetisk mångfald som ett grundläggande mål för bevarande av biologisk mångfald, med arter som den enhet som i stort sett definieras för skydd. Definitionerna av arter enligt lagstiftning och internationella direktiv är emellertid extremt varierande. De innehåller taxonomiskt erkännande av arter och intraspecifika taxa, men kan också innehålla genetiska/evolutionära begrepp som ofta omfattas av den juridiska definitionen av en ”art” (Tabell 1).

tabell 1. Världslagstiftning och direktiv som definierar ”arter” för notering och skydd.
Med tanke på det breda utbudet av infra-specifika nivåer som kan skyddas (Tabell 1) bör lagstiftningen och tillhörande noteringsprocesser i länder underlätta bevarandet av genetisk mångfald. Till exempel kan definitionen av en ”art”, när man överväger lagstiftning runt om i världen (Tabell 1), inkludera: arter, underarter, sorter och geografiskt och/eller genetiskt distinkta populationer, medan det enligt Sydafrikansk lagstiftning också kan omfatta sorter, geografiska raser, stammar och hybrider. Således, enligt de flesta lagstiftning globalt en ”art” omfattar enheter som återspeglar olika infraspecifika nivåer som alla är en del av en bredare artfördelning och återspeglar i varierande grad populationsartens kontinuum. Detta paralleller den oundvikliga svårigheten som taxonomer, evolutionsbiologer och bevarandegenetiker står inför för att hitta ett mer konsekvent och enhetligt sätt att specificera enheter inom detta kontinuum.
Vissa hävdar att farhågor om de politiska konsekvenserna av arter och infraspecifik osäkerhet är felplacerade (Hej et al., 2003), men vi anser fortfarande att även om lagstiftning kan ge en mekanism för att skydda genetisk mångfald på en rad nivåer, åligger det biologer att ge mer klarhet och konsekvens om hur dessa nivåer ska avgränsas från arter till enskilda populationer. För närvarande lämnas chefer och lagstiftare med ett förvirrande utbud av artkoncept och infraspecifika enheter som tillhandahålls av forskare när de avgränsar enheter över populationsartens kontinuum. Till exempel anser alla jurisdiktioner arter som erkänns av taxonomer, som viktiga enheter för bevarande (Tabell 1), men det finns fortfarande en bred debatt bland taxonomer kring artbegrepp, och hur arter diagnostiseras i praktiken varierar inom och mellan grupper. Detta har lett till en ny uppmaning till ett mer reglerat tillvägagångssätt för taxonomi (Garnett och Christidis, 2017), som andra har motverkat att reglering av taxonomi inte är svaret (Thomson et al., 2018). Även om vi instämmer med några av de punkter som Thomson et al. (2018) Vi föreslår att det är ganska naivt att föreslå att lagstiftningen själv behöver ändras för att uttryckligen referera till det specifika taxon-konceptet som antyds av ett namn. Dessa och tidigare sådana debatter belyser behovet av evolutionära biologer och taxonomer att ta ledningen för att säkerställa tydlighet (öppenhet) i hur de översätter genomiska och andra bevis för avgränsning av arter eller intraspecifika enheter, och hur dessa taxa relaterar till relevant lagstiftning för bevarande och skydd.
översättning till ledning
som diskuterats ovan erkänner all lagstiftning och direktiv artnivån som den grundläggande bevarandeenheten, men föreskriver också erkännande av olika infraspecifika enheter. Många har också ett underförstått krav på formell namngivning och taxonomisk beskrivning av arter när de beaktas för bevarandeprioritering och formellt skydd. Till exempel, enligt IUCN Red notering riktlinjer notering av icke namngivna arter avskräcks och det finns ett krav på att den nya artbeskrivningen kommer att publiceras inom fyra år efter det att arten tagits med på IUCN Red List eller det skulle tas bort. Detta belyser vikten av taxonomisk namngivning och beskrivning av enheter, på både arter och infraspecifika nivåer, den begränsade taxonomiska informationen för många växt-och djurgrupper (Mace, 2004; Oliver och Lee, 2010; Wilson, 2017) och behovet av att påskynda taxonomisk ansträngning, särskilt i rika områden med biologisk mångfald (Riedel et al., 2013; Wege et al., 2015; Wilson, 2017).
taxonomiskt namngivna arter och intraspecifika taxonomiska kategorier, såsom underarter och sorter, ger i allmänhet de grundläggande enheterna för formell notering, bevarandeprioritering och skydd, men i vissa jurisdiktioner erkänns andra kategorier av intraspecifika bevarandeenheter formellt. Dessa identifieras generellt med hjälp av kombinerade genomiska och fenotypiska data. Till exempel erkänner USA Endangered Species Act distinkta Befolkningssegment (DPS) som i stort sett är baserade på ESUs (Waples, 1991), medan den kanadensiska Species at Risk Act erkänner DUs. En operativ tolkning av ”distinkta populationer” enligt Canadian Species at Risk Act, syftar DUs till att identifiera diskreta populationer som representerar viktiga komponenter i artens evolutionära arv och som sannolikt inte kommer att ersättas genom naturlig spridning (Mee et al., 2015).
både DUs och DPS överlappar konceptuellt med ESUs (Waples et al., 2013) och har tillämpats mest på nordamerikanska fiskar, inklusive nordvästra USA Laxfiskar (Waples, 1995) och kanadensiska coregonids (Mee et al., 2015) intressant DPS i USA gäller endast ryggradsdjur medan i Kanada DUs kan appliceras på alla organismer som kan listas enligt dess lag (se Waples et al., 2013), med DUs för närvarande listad för leddjur, blötdjur och växter. I Australien, som Kanada, kan distinkta populationer listas för alla organismer som erkänns enligt lagen om miljöskydd och biologisk mångfald bevarande 1999 (EPBC Act) även om de inte är en specifikt namngiven kategori, såsom DUSs och DPS. Men hittills har de bara erkänts för ryggradsdjur. I USA finns det 127 ryggradsdjur DPS listade för bevarande (https://ecos.fws.gov/ecp/); I Kanada har 70 DUs listats som täcker alla organismer (http://www.registrelep-sararegistry.gc.ca/species/schedules_e.cfm?id=1); och i Australien har populationer av 7 ryggradsarter som täcker däggdjur, fisk och fåglar erkänts ha specifik bevarandestatus enligt EPBC-lagen (http://www.environment.gov.au/cgi-bin/sprat/public/publicthreatenedlist.pl). Medan bedömningar av dessa populationer i USA och Kanada i allmänhet använder genetisk information (Waples et al., 2013), i Australien är de vanligtvis baserade på geografisk och jurisdiktionsidentifiering, även om de i vissa av dessa fall har fått stöd av analys av genetisk differentiering för däggdjur (Firestone et al., 1999; Armstrong, 2006) och fisk (Colgan och Paxton, 1997; Ahonen et al., 2009).
noteringen av infraspecifika bevarandeenheter för växter och ryggradslösa djur i Kanada (DUs) men inte i USA eller Australien belyser en annan fråga som inte är specifikt relaterad till avgränsningen av bevarandeenheter utan mer allmänt till betydande inkonsekvenser i prioritering av biologisk mångfald.ikoniska ryggradsdjur som fåglar, däggdjur och fisk är ett fokus i många jurisdiktioner medan andra organismer inte är det. Denna förmånliga lista över populationer av vissa organismer återspeglar en rad frågor som är mindre att göra med vetenskapen om att avgränsa bevarandeenheter och mer att göra med noteringen av populationer baserade på allmänhetens medvetenhet, politisk lämplighet och den karismatiska karaktären hos organismen som ska listas (se Waples et al., 2013).
medan vissa populationer av välkända däggdjur, fåglar och fiskar i Australien skyddas enligt EPBC-lagen, finns det nu en betydande mängd information baserad på molekylära studier på reptiler, ryggradslösa djur och växter där populationer kan erkännas som ESUs eller MUs, och många av dessa är av bevarandebetydelse med tanke på deras smala geografiska intervall, liten befolkningsstorlek, nedgång och mottaglighet för hotande processer. Till exempel kan slående exempel på mycket strukturerade populationer och djupt divergerande linjer inom arter som lätt skulle passa kravet på en ESU hittas i en rad ryggradsdjur med låg spridning från tropikerna (Figur 2) och på andra håll. På samma sätt i Australiska ryggradslösa djur, såsom amfipoder och tusenfotar, betydande genetisk struktur och fylogeografiska mönster stöder avgränsningen av diskreta befolkningskluster som ESUs som är geografiskt olika, med smala geografiska intervall, begränsade till specifika livsmiljöer och representeras av endast ett fåtal populationer (Finston et al., 2007; Murphy et al., 2013; Nistelberger et al., 2014a). Ofta är dessa kortdistans endemiska taxa av bevarandeproblem (Harvey et al., 2011) och samlokaliseras i endemism hotspots (t.ex. Oliver et al., 2017), som själva är höga prioriteringar för multi-taxon bevarande uppmärksamhet (Rosauer et al., 2018), såsom Kimberley-regionen och Stirling Range i sydvästra västra Australien.
i Australiska växter finns det också ett betydande antal populationsgenetiska och fylogeografiska studier som identifierar distinkta befolkningsgrupper inom en rad arter från olika släkter som återspeglar disjunct och historiskt isolerade populationssystem, geologiska och edafiska komplexiteter och upptar kontrasterande livsmiljöer när det gäller vegetation och klimat (se Broadhurst et al., 2017). Genetiskt distinkta populationer är särskilt tydliga i arter från sydvästra västra Australien (Coates, 2000; Byrne et al., 2014) med ett antal studier som lyfter fram betydande genetisk struktur och mycket divergerande linjer i en rad växtsläkter och familjer inklusive: Acacia (Mimosaceae; Coates, 1988), Atriplex (Amaranthaceae; Shepherd et al., 2015), Banksia (Proteaceae; Coates et al., 2015), Lambertia (Proteaceae; Byrne et al., 2002), Hakea (Proteaceae; Sampson et al., 2014), Eremophila (Scrophulariaceae; Llorens et al., 2015), Pultanaea (Fabaceae; Millar och Byrne, 2013) och Calothamnus (Myrtaceae; Nistelberger et al., 2014b). I många fall erkänns dessa populationer i bevarandeplanering av statliga myndigheter och beaktas i miljökonsekvensbedömningar (se Shepherd et al., 2015) även om de inte är listade för skydd enligt lagstiftning. Vi erkänner emellertid att formellt erkännande av dessa enheter skulle förbättra deras bevarande på lång sikt.
dessa australiska exempel belyser det ökande behovet av att känna igen detta lager av genetisk mångfald under artnivån. Utmaningen är att se till att mycket strukturerade populationer och djupt divergerande linjer som är av bevarandehänsyn skyddas inför konkurrerande krav på bevarandeuppmärksamhet. Utöver ikoniska arter är denna utmaning också uppenbar globalt med undantag för Kanada. Underlåtenhet att skydda tydligt avgränsade bevarandeenheter kommer sannolikt att öka risken för kryptisk utrotning och förlust av betydande genetisk mångfald, vilket i många fall kan innebära linjer och historiskt isolerade populationer som har kvarstått inom arter i miljontals år (Moritz, 2002).
slutsatser och rekommendationer
noteringen av arter och olika infraspecifika kategorier enligt hotad artlagstiftning eller enligt internationella organisationer som IUCN, indikerar ett åtagande att skydda genetisk mångfald över hela artens populationskontinuum. Men nuvarande tillvägagångssätt för bevarande av biologisk mångfald fokuserar till stor del på artens slut på detta kontinuum med mindre uppmärksamhet åt genetisk mångfald och infraspecifik variation. Detta mer begränsade fokus på genetisk variation inom artnivå tyder på att medan bevarandechefer i allt högre grad känner igen värdet av att uttryckligen överväga genetisk information i förvaltningsåtgärder och prioritering, saknar de ofta handlingsbar information från biodiversitetsforskare (Waples et al., 2008; Laikre et al., 2010; kock och Sgrcr, 2017; Taylor et al., 2017). Denna klyfta mellan vetenskap och bevarandepraxis hindrar en mer effektiv global strategi för att bevara och hantera genetisk mångfald.
Vi föreslår att bristen på klarhet från biodiversitetsforskare över bevarandeenheter i stor utsträckning bygger på inkonsekvenser på två nivåer. Den första handlar om flera arter begrepp och de olika diagnostiska metoder som används för att avgränsa arter som skapar inkonsekvens mellan taxonomiska grupper. Den andra avser brist på konsistens och överenskommelse av taxonomer, evolutionsbiologer och bevarandebiologer om hur man behandlar infra-specifika taxa och andra enheter som ESUs, DUs och MUs.
även om vi anser att lagstiftningen ofta redan är tillräckligt flexibel när det gäller hur taxa (eller ”arter”) definieras, hävdar vi att det finns ett behov av att systematiskt ta itu med populationsartens kontinuum och tydligare definiera enheter för bevarande med större erkännande av begrepp som ESU: s och hur dessa relaterar till relevant lagstiftning. Vi betonar att denna fråga utan tvekan kommer att bli mer kritisk med tanke på genomikens kraft att upptäcka differentiering i progressivt finare skalor och dess ökande användning för att avgränsa arter och infraspecifika enheter.
från föregående analys erbjuder vi följande rekommendationer:
(I) genomiska data bör tolkas konservativt när man avgränsar arter, med hjälp av ett integrativt taxonomiskt tillvägagångssätt som vanligtvis innehåller bevis utöver de genetiska uppgifterna ensam.
(ii) där genetiker identifierar flera släkter eller distinkta populationer inom en Art, och dessa inte motiverar höjning till artnivå och inte taxonomiskt erkänns, att det finns starkare drivkraft och en mer enhetlig metod för att identifiera tydligt motiverade intraspecifika enheter för praktisk bedömning av bevarandestatus eller konsekvensbedömningar, och att dessa uttryckligen erkänns som enheter för bevarande inom ramen för lagstiftning som är relevant för den jurisdiktionen.
(iii) övervägande ges av IUCN, genom sin Conservation Genetics Specialist Group och dess regionala undergrupper, för att utveckla riktlinjer för erkännande och märkning av intraspecifika enheter som överensstämmer med bästa vetenskapliga praxis.
(iv) den mer nyanserade uppfattningen som kommer från evolutionär genomik av arter och kontinuum från populationer till arter bör kommuniceras mer effektivt av forskare till bevarandeförvaltare och beslutsfattare.
Mer Allmänt upprepar vi rekommendationer som nyligen gjorts i ett antal recensioner (Frankham, 2010; Santamar Czona och M Ubigndez, 2012; Hoban et al., 2013; Moritz och Potter, 2013; Laity et al., 2015; Pierson et al., 2016; kock och Sgrcr, 2017; Taylor et al., 2017) som belyser behovet av forskare att förbättra kommunikationen och öka medvetenheten med beslutsfattare, myndigheter, icke-statliga organisationer och utövare som resultat från molekylär Systematik, molekylär taxonomi, fylogeografi och populationsgenetisk forskning kan ge kritisk information om att bevara genetisk mångfald och hantera evolutionära processer.
Författarbidrag
DC, MB och CM bidrog alla till förberedelserna och revideringen av manuskriptet.
intressekonflikt uttalande
författarna förklarar att forskningen genomfördes i avsaknad av kommersiella eller finansiella relationer som kan tolkas som en potentiell intressekonflikt.
bekräftelser
detta projekt stöddes av Australian Research Council (ARC Linkage Grant No. Lp120200063), Australian National University och Western Australian Department of Biodiversity, Conservation och attraktioner.
Afonso Silva, A. C., Bragg, J. G., Potter, S., Fernandes, C., Coelho, M. M. och Moritz, C. (2017a). Tropisk specialist kontra klimatgeneralist: diversifiering och demografisk historia av systerarter av Carlia skinks från nordvästra Australien. Mol. Ecol. 26:4045–4058. doi: 10.1111 / mec.14185
CrossRef fulltext / Google Scholar
Afonso Silva, AC, Santos, N., Ogilvie, ha och Moritz, C. (2017b). Validering och beskrivning av två nya nordvästra australiska Regnbågsskinn med multispecies koalescerande metoder och morfologi. PeerJ 5: e3724. doi: 10.7717/peerj.3724
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Ahonen, H., Harcourt, R. G. och Stow, A. J. (2009). Nukleärt och mitokondriellt DNA avslöjar isolering av imperillerad grå Sjuksköterska hajpopulationer (Carcharias taurus). Mol. Ecol. 18, 4409–4421. doi: 10.1111 / j. 1365-294x.2009.04377.X
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Armstrong, K. N. (2006). Fylogeografisk struktur i Rhinonicteris aurantia (Chiroptera: Hipposideridae): konsekvenser för bevarande. Acta Chiropterologica 8, 63-81. doi: 10.3161/1733-5329(2006)82.0.CO
CrossRef fulltext / Google Scholar
Arnold, M. L., och Kunte, K. (2017). Adaptiv genetisk utbyte: en trassig historia av blandning och evolutionär innovation. Trender Ecol. Evol. 32, 601–611. doi: 10.1016/j.träd.2017.05.007
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Avise, J. C. (2004). Molekylära markörer, naturhistoria och Evolution. 2: a Edn. Sunderland, MA; Oxford: Oxford University Press.
Google Scholar
Avise, J. C., Arnold, J., boll, R. M., Bermingham, E., lamm, T., Neigel, J. E., et al. (1987). Intraspecifik fylogeografi: den mitokondriella DNA-bron mellan populationsgenetik och systematik. Ann. Rev. Ecol. Sys. 18, 489–522. doi: 10.1146 / annurev. es. 18. 110187.002421
CrossRef fulltext | Google Scholar
Bickford, D., Lohman, D. J., Sodhi, ns, Ng, P. K., Meier, R., Winker, K., et al. (2007). Kryptiska arter som ett fönster mot mångfald och bevarande. Trender Ecol. Evol. 22, 148–155. doi: 10.1016/j.träd.2006.11.004
PubMed Abstrakt | CrossRef fulltext/Google Scholar
Blom, M. P. K., Bragg, J. G., Potter, S. och Moritz, C. (2016). Redovisning av osäkerhet i genträd uppskattning: sammanfattning-koalescerande arter träd inferens i en utmanande strålning av australiska ödlor. Syst. Biol. 66, 352–366. doi: 10.1093 / sysbio / syw089
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Broadhurst, L., ras, M., Lowe, A., Bragg, J., Catullo, R., Coates, D. J., et al. (2017). Genetisk mångfald och struktur av den australiensiska floran. Dykare. Distrib. 23, 41–52. doi: 10.1111/ddi.12505
CrossRef fulltext / Google Scholar
Byrne, M., Coates, D. J., skog, F., Hopper, S. D., Krauss, S. L., Sniderman, J. M. K., et al. (2014). ”En mångsidig flora-Art och genetiska relationer”, i växtlivet på Sandlätterna i sydvästra Australien, en global Hotspot för biologisk mångfald, redaktörer H. Lambers (Crawley, WA: University of Western Australia Publishing), 81-99.
PubMed Abstrakt
Byrne, M., Macdonald, B. Och Coates, D. J. (2002). Fylogeografiska mönster och hög cpdna-mångfald inom Acacia acuminata (Leguminosae:Mimosoideae). komplex i västra Australien. J. Evol. Biol. 15, 576–587. doi: 10.1046 / j. 1420-9101. 2002.00429.X
CrossRef fulltext / Google Scholar
Carstens, BC, Pelletier, ta, Reid, Nm och Satler, jd (2013). Hur man misslyckas vid artavgränsning. Mol. Ecol. 22, 4369–4383. doi: 10.1111 / mec.12413
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Coates, D. J. (1988). Genetisk mångfald och populationsgenstruktur i den sällsynta Chittering gräs wattle, Acacia anomala domstol. Aust. J. Bot. 36:273–286. doi: 10.1071 / BT9880273
CrossRef fulltext / Google Scholar
Coates, D. J. (2000). Definiera bevarandeenheter i en rik och fragmenterad flora: konsekvenser för hantering av genetiska resurser och evolutionära processer i sydvästra Australien växter. Aust. J. Bot. 48, 329–339. doi: 10.1071 / BT99018
CrossRef fulltext / Google Scholar
Coates, D. J., McArthur, S. L. och Byrne, M. (2015). Betydande förlust av genetisk mångfald efter patogendriven befolkningsutrotning i den sällsynta endemiska Banksia brownii (Proteaceae). Biol. Conserv. 192, 353–360. doi: 10.1016 / j. biocon.2015.10.013
CrossRef fulltext / Google Scholar
Colgan, DJ och Paxton, Jr (1997). Biokemisk genetik och erkännande av ett västerländskt bestånd av den gemensamma gemfishen, Rexea solandri (Scombroidea: Gempylidae)., i Australien. Mar. Freshw. Res. 48, 103-118. doi: 10.1071 / MF96048
CrossRef fulltext / Google Scholar
konventionen om biologisk mångfald (2007). Tillgänglig online på: https://www.cbd.int/doc/meetings/cop-bureau/cop-bur-2007/cop-bur-2007-10-14-en.pdf
Cook, C. N. och Sgrcr, C. M. (2017). Anpassning av vetenskap och politik för att uppnå evolutionärt upplyst bevarande. Conserv. Biol. 31, 501–512. doi: 10.1111 / cobi.12863
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Cook, LG, Edwards, Rd, Crisp, MD och Hardy, Nb (2010). Behöver morfologi alltid krävas för nya artbeskrivningar? Invertera. Sys. 24, 322–332. doi: 10.1071 / IS10011
CrossRef fulltext / Google Scholar
Cracraft, J. (1983). Artbegrepp och specieringsanalys. Curr. Ornitol. 1, 159–187. doi: 10.1007/978-1-4615-6781-3_6
CrossRef fulltext / Google Scholar
Crandall, ka, Bininda-Emonds, or, Mace, GM och Wayne, rk (2000). Med tanke på evolutionära processer inom bevarandebiologi. Trender Ecol. Evol. 15, 290–295. doi: 10.1016 / S0169-5347(00) 01876-0
PubMed Abstrakt | CrossRef fulltext | Google Scholar
Cutter, A. D., och Payseur, B. A. (2013). Genomiska signaturer för urval på länkade platser: förena skillnaden mellan arter. Nat. Rev.Genet. 14, 262–274. doi: 10.1038/nrg3425
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Dayrat, B. (2005). Mot integrativ taxonomi. Biol. J. Linn. Soc. 85, 407–415. doi: 10.1111 / j. 1095-8312. 2005.00503.X5
CrossRef fulltext / Google Scholar
de Queiroz, K. (1998). ”Det allmänna begreppet Art, artkriterier och specieringsprocessen”, i former: Art och speciering, D. J. Howard,R S. H. Berlocher (New York, NY: Oxford University Press), 57-75.
Google Scholar
de Queiroz, K. (2007). Artbegrepp och artavgränsning. Syst. Biol. 56, 879–886. doi: 10.1080 / 10635150701701083
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Degnan, J. H. och Rosenberg, N. A. (2009). Genträd discordance, fylogenetisk inferens och multispecies coalescent. Trender Ecol. Evol. 24, 332–340. doi: 10.1016/j.träd.2009.01.009
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Devey, D. S., Bateman, R. M., Fay, M. F. och Hawkins, J. A. (2008). Vänner eller släktingar? Fylogenetik och artavgränsning i det kontroversiella Europeiska orchid-släktet Ophrys. Ann. Bot. 101, 385–402. doi: 10.1093 / AOB / mcm299
PubMed Abstrakt | CrossRef fulltext/Google Scholar
Dizon, A. E., Lockyer, C, Perrin, W. F., Demaster, D. P. och Sisson, J. (1992). Ompröva aktiekonceptet-ett fylogeografiskt tillvägagångssätt. Conserv. Biol. 6, 24–36. doi: 10.1046 / j. 1523-1739. 1992.610024.X
CrossRef fulltext / Google Scholar
Doughty, P., Bourke, G., Tedeschi, L., Pratt, R., Oliver, P., Palmer, R., et al. (2018). Artavgränsning i Gehyra nana (Squamata: Gekkonidae). komplex: kryptisk och divergerande morfologisk utveckling i de australiska Monsoonala tropikerna, med beskrivningen av fyra nya arter. Zootaxa, 4403: 201-244. doi: 10.11646/zootaxa.4403.2.1
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Dynesius, M. och Jansson, R. (2014). Persistens av linjer inom arter: en försummad kontroll av specieringshastigheter. Evolution 68, 923-934. doi: 10.1111 / evo.12316
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Edwards, S. V., Potter, S., Schmitt, C. J., Bragg, J. G. och Moritz, C. (2016). Retikulering, divergens och fylogeografi–fylogenetik kontinuumc. Proc. Natl. Acad. Sci. USA 113, 8025-8032. doi: 10.1073 / pnas.1601066113
CrossRef fulltext / Google Scholar
tro, D. P. (1992). Bevarande utvärdering och fylogenetisk mångfald. Biol. Nackdelar. 61, 1–10. doi: 10.1016/0006-3207(92)91201-3
CrossRef fulltext / Google Scholar
Finston, T. L., Johnson, ms, Humphreys, W. F., Eberhard, S. M. och Halse, S. A. (2007). Kryptisk speciering i två utbredda underjordiska amfipodsläkten återspeglar historiska dräneringsmönster i ett gammalt landskap. Mol. Ecol. 16, 355–365. doi: 10.1111 / j. 1365-294x.2006.03123.X
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Firestone, K. B., Elphinstone, ms, Sherwin, W. B. Och Houlden, B. A. (1999). Fylogeografisk befolkningsstruktur av tiger quolls dasyurus maculatus (Dasyuridae: Marsupalia), en hotad köttätande pungdjur. Mol. Ecol. 8, 1613–1625. doi: 10.1046 / j. 1365-294x.1999.00745.X
CrossRef fulltext / Google Scholar
Fi C., Robinson, C. T. och Malard, F. (2018). Kryptiska arter som ett fönster in i paradigmskiftet av artbegreppet. Mol. Ecol. 27, 613–635. doi: 10.1111 / mec.14486
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Frankham, R. (2010). Utmaningar och möjligheter med genetiska metoder för biologisk bevarande. Biol. Conserv. 143,1919–1927. doi: 10.1016 / j. biocon.2010.05.011
CrossRef fulltext / Google Scholar
Frankham, R., Ballou, J. D., Dudash, M. R. Eldridge, M. D., Fenster, C. B., Lacy, R. C., et al. (2012). Konsekvenser av olika artbegrepp för att bevara biologisk mångfald. Biol. Conserv. 153, 25–31. doi: 10.1016 / j. biocon.2012.04.034
CrossRef fulltext / Google Scholar
Fraser, D. J. och Bernatchez, L. (2001). Adaptiv evolutionär bevarande: mot ett enhetligt koncept för att definiera bevarandeenheter. Mol. Ecol. 10, 2741–2752. doi: 10.1046 / j. 1365-294X.2001.t01-1-01411.X
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Funk, W. C., Mckay, J. K., Hohenlohe, P. A., och Allendorf, F. W. (2012). Utnyttja genomik för att avgränsa bevarandeenheter. Trender Ecol. Evol. 27, 489–496. doi: 10.1016/j.träd.2012.05.012
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Garnett, S. T. och Christidis, L. (2017). Taxonomianarki hindrar bevarande. Natur 546, 25-27. doi: 10.1038 / 546025a
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Gippoliti, S., Cotterill, F. P. D., Zinner, D. och Groves, C. P. (2018). Effekter av taxonomisk tröghet för bevarande av afrikansk hovdjur mångfald: översikt. Biol. Rev. 93, 115-130. doi: 10.1111 / brv.12335
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Grant, V. (1981). Växt Speciering. New York, NY: Columbia University Press.
PubMed Abstract / Google Scholar
Hamilton, C. W. och Reichard, S. H. (1992). Nuvarande praxis vid användning av underarter, sorter och forma vid klassificering av vilda växter. Taxon 41, 485-498. doi: 10.2307 / 1222819
CrossRef fulltext / Google Scholar
Harvey, M. S., Rix, M. G., Framenau, V. W., Hamilton, Z. R., Johnson, M. S., Teale, R. J., et al. (2011). Skydda oskyldiga: att studera kortdistans endemiska taxa förbättrar bevaranderesultat. Invertera. Syst. 25, 1–10. doi: 10.1071/IS11011
CrossRef fulltext/Google Scholar
Hej, J., Waples, rs, Arnold, ml, Butlin, rk och Harrison, rg (2003). Förstå och konfrontera artens osäkerhet i biologi och bevarande. Trender Ecol. Evol. 18, 597–603. doi: 10.1016/j.träd.2003.08.014
CrossRef fulltext / Google Scholar
Hoban, S. M., Hauffe, H. C., Perez-Espona, S., Arntzen, J. W., Bertorelle, G., Bryja, J., et al. (2013). Att föra genetisk mångfald i framkant av bevarandepolitik och förvaltning. Conserv. Genet. Resour. 5, 593–598. doi: 10.1007 / s12686-013-9859-y
CrossRef fulltext / Google Scholar
Isaac, nj, Mallet, J. och Mace, GM (2004). Taxonomisk inflation: dess inflytande på makroekologi och bevarande. Trender Ecol. Evol. 19, 464–469. doi: 10.1016/j.träd.2004.06.004
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Isaac, N. J., Turvey, S. T., Collen, B., Waterman, C. och Baillie, J. E. (2007). Däggdjur på kanten: bevarandeprioriteringar baserade på hot och fylogeni. PLoS EN 2: e296. doi: 10.1371 / tidskrift.pone.0000296
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Jones, M. R., och bra, J. M. (2016). Målinriktad fångst i evolutionär och ekologisk genomik. Mol. Ecol. 25, 185–202. doi: 10.1111 / mec.13304
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Jorger, K. M., och Schr Occuldl, M. (2013). Hur beskriver man en kryptisk Art? Praktiska utmaningar för molekylär taxonomi. Front. Zool. 10:59. doi: 10.1186/1742-9994-10-59
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Laikre, L., Allendorf, F., Aroner, L. C., Baker, S., Gregovich, D. P., Hansen, M. M., et al. (2010). Försummelse av genetisk mångfald vid genomförandet av konventionen om biologisk mångfald. Conserv. Biol. 24, 86–88. doi: 10.1111 / j. 1523-1739. 2009.01425.X
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Laikre, L., Palm, S. och Ryman, N. (2005). Fiskens genetiska befolkningsstruktur: konsekvenser för kustområdets förvaltning. Ambio 34,111-119. doi: 10.1579/0044-7447-34.2.111
PubMed Abstract / CrossRef fulltext / Google Scholar
Laity, T., Laffan, S. W., Gonz Ubiclez-Orozco, ce, tro, D. P., Rosauer, D. F., Byrne, M., et al. (2015). Phylodiversity för att informera bevarandepolitiken: ett australiskt exempel. Sci. Total Miljö. 534, 131–143. doi: 10.1016/j.scitotenv.2015.04.113
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Leache, AD och Oaks, Jr (2017). Användbarheten av enkel nukleotidpolymorfism (SNP). data i fylogenetik. Ann. Rev. Ecol. Syst. 48, 69–84. doi: 10.1146/annurev-ecolsys-110316-022645
CrossRef Full Text | Google Scholar
Llorens, T. M., Macdonald, B., McArthur, S., Coates, D. J., and Byrne, M. (2015). Disjunct, highly divergent genetic lineages within two rare Eremophila (Scrophulariaceae: Myoporeae). species in a biodiversity hotspot: implications for taxonomy and conservation. Biol. J. Linn. Soc. 177, 96–111. doi: 10.1111/boj.12228
CrossRef Full Text | Google Scholar
Love Stowell, S. M., Pinzone, C. A., and Martin, A. P. (2017). Att övervinna hinder för aktiva insatser för genetisk mångfald. Biologisk mångfald. Conserv. 26, 1753–1765. doi: 10.1007 / s10531-017-1330-z
CrossRef fulltext / Google Scholar
Mace, G. M. (2004). Taxonomins roll i artbevarande. Philos. Trans. R. Soc. B: Biol. Sci. 359, 711–719. doi: 10.1098 / rstb.2003.1454
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Mallet, J. (2007). Hybrid speciering. Natur 446, 279-283. doi: 10.1038/nature05706
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Mayr, E. (1942). Systematik och artens ursprung. New York, NY: Columbia University Press.
Google Scholar
Mee, J. A., Bernatchez, L., Reist, J. D., Rogers, S. M. och Taylor, E. B. (2015). Identifiera designerbara enheter för intraspecifik bevarandeprioritering: ett hierarkiskt tillvägagångssätt som tillämpas på sjöfiskartskomplexet (Coregonus spp.). Evol. Appl. 8, 423–441. doi: 10.1111 / eva.12247
PubMed Abstract | CrossRef Full Text | Google Scholar
Millar, M. A., and Byrne, M. (2013). Cryptic divergent lineages of Pultenaea pauciflora M.B. Scott (Fabaceae: Mirbelieae) exhibit different evolutionary history. Biol. J. Linn. Soc. 108, 871–881. doi: 10.1111/bij.12002
CrossRef Full Text | Google Scholar
Moritz, C. (1994). Defining evolutionarily significant units for conservation. Trends Ecol. Evol. 9, 373–375. doi: 10.1016/0169-5347(94)90057-4
CrossRef Full Text | PubMed Abstract
Moritz, C. (1999). Bevarandeenheter och translokationer: strategier för att bevara evolutionära processer. Hereditas 130, 217-228. doi: 10.1111 / j. 1601-5223. 1999.00217.X
CrossRef fulltext / Google Scholar
Moritz, C. (2002). Strategier för att skydda biologisk mångfald och de evolutionära processer som upprätthåller den. Syst. Biol. 51, 238–254. doi: 10.1080 / 10635150252899752
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Moritz, C., Och Potter, S. (2013). Vikten av ett evolutionärt perspektiv i planeringen av bevarandepolitiken. Mol. Ecol. 22, 5969–5971. doi: 10.1111 / mec.12565
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Moritz, C., Pratt, R. C., Bank, S., Bourke, G., Bragg, J. G., Doughty, P., et al. (2018). Kryptisk härstamning mångfald, kroppsstorlek divergens, och sympatri i en art komplex av australiska ödlor (Gehyra). Evolution 27, 54-66. doi: 10.1111 / evo.13380
CrossRef fulltext / Google Scholar
Morrison, W. R., Lohr, J. L., Duchen, P., Wilches, R., Trujillo, D., Mair, M., et al. (2009). Effekterna av taxonomisk förändring på bevarande: Dödar det, kan det spara, eller är det bara irrelevant? Biol. Nackdelar. 142, 3201–3206. doi: 10.1016 / j. biocon.2009.07.019
CrossRef fulltext / Google Scholar
Murphy, NP, Adams, M., Guzik, M. T. och Austin, A. D. (2013). Extraordinär mikroendemism i Australiska ökenfjäder amfipoder. Mol. Fylogenet. Evol. 66, 645–653. doi: 10.1016 / j. ympev.2012.10.013
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Murphy, NP, Kung, ra och Delean, S. (2015). Arter, ESUs eller populationer? Avgränsar och beskriver morfologiskt kryptisk mångfald i Australiska ökenfjäder amfipoder. Invertera. Syst. 29, 457–467. doi: 10.1071/IS14036
CrossRef fulltext | Google Scholar
Nater, A., Mattle-Greminger, M. P., Nurcahyo, A., Nowak, M. G., De Manual, M., Desai, T., et al. (2017). Morfometriska, beteendemässiga och genomiska bevis för en ny orangutangart. Curr. Cell Biol. 27, 3487–3498. doi: 10.1016 / j. unge.2017.09.047
CrossRef fulltext / Google Scholar
Nicotra, A. B., Chong, C., Bragg, J. G., Ong, C. R., Aitken, N. C., Chuah, A., et al. (2016). Befolkning och fylogenomisk sönderdelning via genotypning-genom-sekvensering i australiskt Pelargonium. Mol. Ecol. 25, 2000–2014. doi: 10.1111 / mec.13584
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Nistelberger, H., Byrne, M., Coates, D. och Roberts, J. D. (2014a). Stark fylogeografisk struktur i en millipede indikerar pleistocen vicariance mellan populationer på bandade järnformationer i halvtorra Australien. PLoS en 9: e0093038. doi: 10.1371 / tidskrift.pone.0093038
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Nistelberger, H., Gibson, N., Macdonald, B., Tapper, S.-L. och Byrne, M. (2014b). Fylogeografiska bevis för två mesic refugia i en hotspot för biologisk mångfald. Ärftlighet 113, 454-463. doi: 10.1038/hdy.2014.46
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Ogilvie, H. A., Heled, J., Xie, D. och Drummond, A. J. (2016). Beräkningsprestanda och statistisk noggrannhet hos djur och jämförelser med andra metoder. Syst. Biol. 65, 381–396. doi: 10.1093/sysbio/syv118
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Oliver, P. M., Keogh, J. S. och Moritx, C. (2015). Nya tillvägagångssätt för katalogisering och förståelse av evolutionär mångfald: ett perspektiv från Australisk herpetologi. Aust. J. Zool. 62, 417–430. doi: 10.1071/ZO14091
CrossRef fulltext/Google Scholar
Oliver, P. M., Laver, R. J., Demello Martins, F., Pratt, R. C., Hunjan, S. och Moritz, C. (2017). En ny hotspot av ryggradsdjur endemism och en evolutionär refugium i tropiska Australien. Dykare. Distrib. 23, 53–66. doi: 10.1111/ddi.12506
CrossRef fulltext / Google Scholar
Oliver, P. M. och Lee, M. S. Y. (2010). De botaniska och zoologiska koderna hindrar forskning om biologisk mångfald genom att avskräcka publicering av namnlösa nya arter. Taxon 59, 1201-1205. doi: 10.2307 / 20773990
CrossRef fulltext / Google Scholar
Padal, J., Miralles, A., De la Riva, I. och Vences, M. (2010). Den integrerade framtiden för taxonomi. Front. Zool. 7:16. doi: 10.1186/1742-9994-7-16
PubMed Abstract / CrossRef fulltext / Google Scholar
Palsb Ubill, PJ, B Ubitrub Ubic, M. och Allendorf, FW (2007). Identifiering av förvaltningsenheter med hjälp av populationsgenetiska data. Trender Ecol. Evol. 22, 11–16. doi: 10.1016/j.träd.2006.09.003
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Patton, J. L. och Smith, M. F. (1994). Paraphyly, polyphyly, och arten av artens gränser i Pocket Gophers (släktet Thomomys). Syst. Biol. 43, 11–26. doi: 10.1093 / sysbio / 43.1.11
CrossRef fulltext / Google Scholar
Pierson, J. C., Coates, D. J., Oostermeijer, J. G. B., Beissinger, S. R., Bragg, J. G., Sunnucks, P., et al. (2016). Genetiska faktorer i hotade arter återhämtningsplaner på tre kontinenter. Front. Ecol. Miljö. 14, 433–440. doi: 10.1002 / avgift.1323
CrossRef fulltext / Google Scholar
Potter, S., Bragg, J. G., Peter, B. M., Bi, K. och Moritz, C. (2016). Fylogenomics på tips: inferring linjer och deras demografiska historia i en tropisk ödla, Carlia amax. Mol. Ecol. 25, 1367–1380. doi: 10.1111 / mec.13546
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Potter, S., Xue, A. T., Bragg, J. G., Rosauer, D. F., Roycroft, E. J. och Moritz, C. (2018). Pleistocene klimatförändringar Driver diversifiering över en tropisk savanna. Mol. Ecol. 27, 520–532. doi: 10.1111 / mec.14441/full
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Rabosky, D. L., Hutchinson, M. N., Donnellan, S. C., Talaba, A. L. och Lovette, I. J. (2014). Fylogenetisk demontering av artgränser i en utbredd grupp australiska skinks (Scincidae: Ctenotus). Mol. Fylogenet. Evol. 77, 71–82. doi: 10.1016 / j. ympev.2014.03.026
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Ralls, K., Ballou, J. D., Dudash, M. R., Eldridge, M. D. B., Fenster, C. B., Lacy, R. C., et al. (2018). Kräver ett paradigmskifte i den genetiska hanteringen av fragmenterade populationer. Conserv. Lett. 11, 1–6. doi: 10.1111 / conl.124126
CrossRef fulltext / Google Scholar
Rannala, B. (2015). Konsten och vetenskapen om artavgränsning. Curr. Zool. 61, 846–853. doi: 10.1093 / czoolo / 61.5.846
CrossRef fulltext / Google Scholar
Riedel, A., Sagata, K., Suhardjono, Y. R., T Acignzler, R. och Balke, M. (2013). Integrativ taxonomi på snabbspåret – mot mer hållbarhet inom forskning om biologisk mångfald. Front. Zool. 10:15. doi: 10.1186 / 1742-9994-10-15
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Rieseberg, L. H. och Willis, J. H. (2007). Växt speciering. Vetenskap 317, 910-914. doi: 10.1126 / vetenskap.1137729
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Rosauer, D. F., Blom, M. P. K., Bourke, G., Catalano, S., Donnellan, S., Mulder, E., et al. (2016). Fylogeografi, hotspots och bevarandeprioriteringar: ett exempel från Australiens övre ände. Biol. Nackdelar. 204, 83–93. doi: 10.1016 / j. biocon.2016.002
CrossRef fulltext / Google Scholar
Rosauer, D. F., Byrne, M., Blom, M. P. K., Coates, D. J., Donnellan, S., Doughty, P., et al. (2018). Verklig bevarandeplanering för evolutionär mångfald i Kimberley, Australien, sidesteps osäker taxonomi Conserv. Lett. 11: e12438. doi: 10.1111 / conl.12438
CrossRef fulltext / Google Scholar
Rosenblum, E. B., Sarver, B. A. J., Brown, J. W., Des Roches, S., Hardwick, K. M., Hether, T. D., et al. (2012). Goldilocks möter Santa Rosalia: en kortvarig specieringsmodell förklarar mönster för diversifiering över tidsskalor. Evol. Biol. 39, 255–261. doi: 10.1007 / s11692-012-9171-X
PubMed Abstract / CrossRef fulltext / Google Scholar
Roux, C., fra Jacobsse, C., Romiguier, J., Anciaux, Y., Galtier, N. och Bierne, N. (2016). Kasta ljus över den grå zonen av speciering längs ett kontinuum av genomisk divergens. PLoS Biol. 14: e2000234. doi: 10.1371 / tidskrift.pbio.2000234
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Ryder, O. A. (1986). Artbevarande och systematik-underartens dilemma. Trender Ecol. Evol. 1, 9–10. doi: 10.1016/0169-5347 (86)90059-5
CrossRef fulltext | Google Scholar
Sampson, J. F., Byrne, M., Yates, C. J., Gibson, N., Thavornkanlapachai, R., Stankowski, S., et al. (2014). Samtida pollenmedierad geninvandring återspeglar den historiska isoleringen av en sällsynt buske i ett fragmenterat landskap. Ärftlighet 112, 172-181. doi: 10.1038/hdy.2013.89
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Santamar Cuba, L. och M Ubigndez, P. F. (2012). Utvecklingen i politiken för biologisk mångfald: nuvarande luckor och framtida behov. Evol. Appl. 5, 202–218. doi: 10.1111 / j. 1752-4571. 2011.00229.X
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Schlick-Steiner, BC, Steiner, FM, Seifert, B., Stauffer, C., Christian, E. och Crozier, Rh (2010). Integrativ taxonomi: en multisource-strategi för att utforska biologisk mångfald. Annu. Rev Entomol. 55, 421–438. doi: 10.1146 / annurev-ento-112408-085432
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Seehausen, O, Takimoto, G., Roy, D. och Jokela, J. (2008). Specieringsomvandling och biologisk mångfaldsdynamik med hybridisering i förändrade miljöer. Mol Ecol. 17, 30–44. doi: 10.1111 / j. 1365-294x.2007.03529.X
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Shepherd, K. A., Thiele, K. R., Sampson, J., Coates, D. J. och Byrne, M. (2015). En sällsynt, ny art av Atriplex (Chenopodiaceae) som omfattar två genetiskt distinkta men morfologiskt kryptiska populationer i torra Västra Australien: konsekvenser för taxonomi och bevarande. Aust. Syst. Bot. 28, 234–245. doi: 10.1071/SB15029
CrossRef fulltext / Google Scholar
Singhal, S. och Bi, K. (2017). Historien rensar upp messes: tidens inverkan vid körning av divergens och introgression i en tropisk suturzon. Evolution 71, 1888-1899. doi: 10.1111 / evo.13278
PubMed Abstrakt | CrossRef fulltext/Google Scholar
Singhal, S., Hoskin, C., Couper, P., Potter, S. och Moritz, C. (2018). Ram för att lösa kryptiska arter: en fallstudie från ödlorna i de australiska våta tropikerna. Syst. Biol. . doi: 10.1093/sysbio/syy
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Singhal, S. och Moritz, C. (2013). Reproduktiv isolering mellan fylogeografiska linjer vågar med divergens. Proc. Roy. Soc B 280: 20132246. doi: 10.1098 / rspb.2013.2246026
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Steane, Da, Potts, B. M., McLean, E., Collins, L., Prober, S. M., lager, W. D., et al. (2015). Genomomfattande skanningar avslöjar kryptisk befolkningsstruktur i en torranpassad eukalypt. Träd Genet. Genom 11:33. doi: 10.1007 / s11295-015-0864-z
CrossRef fulltext / Google Scholar
Struck, T. H., Feder, J. L., Bendiksby, M., Birkeland, S., Cerca, J., Gusarov, V. I., et al. (2017). Hitta evolutionära processer dolda i kryptiska arter. Trender Ecol. Evol. 33, 153–163. doi: 10.1016/j.träd.2017.11.007
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Sukumaran, J., Och Knowles, Ll. L. (2017). Multispecies koalescerande avgränsar strukturen inte arter. Proc. Nat. Aca. Sci. USA 114, 1607-1612. doi: 10.1073 / pnas.1607921114
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Swarts, nd, Clements, ma, Bower, CC och Miller, jt (2014). Definiera bevarandeenheter i ett komplex av morfologiskt liknande, sexuellt bedrägliga, mycket hotade orkider. Biol. Nackdelar. 174, 55–64. doi: 10.1016 / j. biocon.2014.03.017
CrossRef fulltext / Google Scholar
Tautz, D., Arctander, P., Minelli, A., Thomas, Rh och Vogler, A. P. (2003). En grund för DNA-taxonomi. Trender Ecol. Evol. 18, 70–74. doi: 10.1016 / S0169-5347 (02)00041-1
CrossRef fulltext | Google Scholar
Taylor, HR, Dussex, N. och van Heezik, Y. (2017). Överbrygga bevarandegenetikgapet genom att identifiera hinder för genomförande för bevarandeutövare. Glob. Ecol. Conserv. 10, 231–242. doi: 10.1016/j.gecco.2017.04.001
CrossRef fulltext/Google Scholar
Thomson, S. A., Pyle, R. L., Ahyong, S. T., Alonso-Zarazaga, M., Ammirati, J., Araya, J. F., et al. (2018). Taxonomi baserad på vetenskap är nödvändig för global bevarande. PLoS Biol. 16: e2005075. doi: 10.1371 / tidskrift.pbio.2005075
PubMed Abstract / CrossRef fulltext / Google Scholar
Vieites, D. R., Wollenberg, K. C., Andreonec, F., K Xiahler, J., Glaw, F. och Vences, M. (2009). Omfattande underskattning av Madagaskars biologiska mångfald framgår av en integrativ amfibisk inventering. Proc. Natl. Acad. Sci. USA 106, 8267-8272. doi: 10.1073 / pnas.0810821106
CrossRef fulltext
Vogler, A. P. och de Salle, R. (1994). Diagnostisera enheter för bevarande förvaltning. Nackdelar. Biol. 8, 354–363. doi: 10.1046 / j. 1523-1739. 1994.08020354.X
CrossRef fulltext / Google Scholar
Waples, R. S. (1991). Stillahavslax, Oncorhynchus spp., och definitionen av” arter ” enligt lagen om hotade arter. Mar. Fisk. Rev. 53, 11-22.
Google Scholar
Waples, R. S. (1995). ”Evolutionärt betydande enheter och bevarande av biologisk mångfald enligt lagen om utrotningshotade arter”, i evolutionen och det akvatiska ekosystemet: definiera unika enheter i befolkningsskydd, eds Jl Nielsen (Bethesda, MD:American Fisheries Society), 8-27.
Google Scholar
Waples, R. S., Gustafson, R. G., Weitkamp, La, Myers, J. M., Jjonson, O. W., Busby, P. J., et al. (2001). Karaktäriserar mångfald i lax från nordvästra Stillahavsområdet. J. Fisk Biol. 59, 1–41. doi: 10.1111 / j. 1095-8649. 2001.tb01376.X
CrossRef fulltext / Google Scholar
Waples, rs, Nammack, M., Cochrane, JF och Hutchings, Ja (2013). En berättelse om två handlingar: utrotningshotade arter notering praxis i Kanada och USA. BioScience 63, 723-734. doi: 10.1093 / bioscience / 63.9.723
CrossRef fulltext / Google Scholar
Waples, rs, Punt, AE och Cope, JM (2008). Integrera genetiska data i hanteringen av marina resurser: hur kan vi göra det bättre? Fisk 9, 423-449. doi: 10.1111 / j. 1467-2979. 2008.00303.X
CrossRef fulltext / Google Scholar
Wege, J. A., Thiele, K. R., Shepherd, K. A., Butcher, R., Macfarlane, T. D. och Coates, D. J. (2015). Strategisk taxonomi i ett biodiverse landskap: ett nytt tillvägagångssätt för att maximera bevaranderesultat för sällsynt och dåligt känd flora. Biologisk mångfald. Conserv. 24, 17–32. doi: 10.1007 / s10531-014-0785-4
CrossRef fulltext / Google Scholar
Whitlock, R. (2014). Förhållanden mellan adaptiv och neutral genetisk mångfald och ekologisk struktur och funktion: en metaanalys. J. Ecol. 102, 857–872. doi: 10.1111/1365-2745. 12240
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Wiley, E. O. (1978). Det evolutionära artbegreppet omprövas. Syst. Biol. 27, 17–26. doi: 10.2307 / 2412809
CrossRef fulltext / Google Scholar
Wilson, E. O. (2017). Forskning om biologisk mångfald kräver fler stövlar på marken. Nat. Ecol. Evol. 1, 1590–1591. doi: 10.1038 / s41559-017-0360-y
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Yang, zh och Rannala, B. (2014). Unguided Art avgränsning med hjälp av DNA-sekvensdata från flera loci. Mol. Biol. Evol. 31, 3125–3135. doi: 10.1093 / molbev / msu279
PubMed Abstrakt / CrossRef fulltext / Google Scholar
Yeates, DK, Seago, A., Nelson, L., Cameron, sl, Joseph, L. och Trueman, Wh (2011). Integrativ taxonomi eller iterativ taxonomi? Sys. Ent. 36, 209–217. doi: 10.1111/j.1365-3113.2010.00558.x
CrossRef Full Text | Google Scholar