- Introduction
- Génomique, Spéciation et taxonomie
- Génomique et Unités de conservation intraspécifiques
- Phylogénomique et conservation
- Le monde réel — Définitions législatives
- Traduction vers la gestion
- Conclusions et recommandations
- Contributions de l’auteur
- Déclaration sur les conflits d’intérêts
- Remerciements
Introduction
La politique internationale de conservation reconnaît la biodiversité à trois niveaux, écosystème, espèce et génétique, et que la gestion devrait viser à conserver les trois (Convention sur la diversité biologique, 2007). Cela est clairement reflété dans la Convention sur la diversité biologique, Objectifs d’Aichi pour la biodiversité, convenus en 2010 (https://www.cbd.int/sp/targets/), où les objectifs et cibles font spécifiquement référence non seulement aux écosystèmes et aux espèces, mais également à la diversité génétique. Pourtant, les approches actuelles de conservation de la biodiversité sont largement basées sur les zones géographiques, les écosystèmes, les communautés écologiques et les espèces, avec moins d’attention sur la diversité génétique et le continuum espèce-population. En effet, la majorité des praticiens de la conservation se concentrent sur les espèces en tant que mesure de la biodiversité, avec une certaine considération des unités intraspécifiques au sein d’espèces menacées gérées de manière intensive. Dans ce contexte, il existe un désir naturel d’employer des catégorisations simples — espèces nommées et autres taxons nommés (c’est-à-dire sous-espèces), et parfois des unités de conservation au sein des espèces. Les espèces en particulier fournissent une mesure commune de la biodiversité qui sous-tend de nombreux efforts scientifiques et de gestion (Mace, 2004; Wilson, 2017). En tant qu’unité fondamentale de conservation, les espèces sont souvent utilisées pour quantifier la valeur de la biodiversité par le biais de listes d’espèces dans les aires protégées, de l’identification des espèces menacées dans les juridictions et comme base pour les procédures de biosécurité. De plus, les espèces sont généralement les entités avec lesquelles nous communiquons les questions de conservation au public.
Cette impulsion à identifier simplement des unités discrètes dans la nature va à l’encontre de plusieurs réalités connexes. Premièrement, la spéciation n’est pas instantanée. Il s’agit plutôt d’un processus généralement prolongé (Dynesius et Jansson, 2014; Roux et al., 2016), passant de populations isolées, peut-être soumises à différentes pressions de sélection, à des espèces non ambiguës sur de longues échelles de temps, avec des inversions fréquentes causées par des changements environnementaux ou une simple fusion génétique à mesure que les aires de répartition se reconnectent (Figure 1; Seehausen et al., 2008; Rosenblum et coll., 2012). Deuxièmement, la plupart des espèces reconnues ne sont pas génétiquement uniformes. La divergence (dérive et isolement prolongé) et la réticulation (flux génétique, hybridation, introgression et recombinaison) façonnent efficacement la variation géographique et génomique au sein des espèces (Cutter et Payseur, 2013; Edwards et al., 2016) et certaines espèces peuvent être structurées en populations longtemps isolées, dont certaines pourraient mériter d’être reconnues comme des espèces complètes, mais peut-être morphologiquement cryptiques (Bickford et al., 2007; Jorger et Schrödl, 2013). Troisièmement, tant qu’elles sont reconnues depuis longtemps, les espèces nommées ne sont pas équivalentes en termes d’héritage évolutif qu’elles représentent — en termes simples, un ornithorynque (en tant que représentant d’une lignée divergente ancienne) peut être considéré comme valant plus qu’un rongeur récemment divergé (Isaac et al., 2007). Quatrièmement, les désignations d’espèces ne sont pas coulées dans la pierre, mais plutôt des hypothèses à tester à mesure que de nouvelles preuves deviennent disponibles (Hey et al., 2003). Chacune de ces questions crée de la complexité et de l’ambiguïté dans la délimitation des espèces en général et l’attribution d’une attention à leur conservation ou à leurs populations composantes. De plus, la délimitation des espèces est également soumise à différents concepts d’espèces et façons de les diagnostiquer, ce qui a suscité une grande consternation chez les biologistes de la conservation (Mace, 2004; Frankham et al., 2012; Garnett et Christidis, 2017).
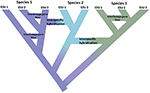
Figure 1. La spéciation en tant que continuum progressant des populations isolées vers des espèces non ambiguës, comme le montre le changement de couleur avec les couleurs représentant différentes espèces. Certaines lignées qui apparaissent en raison de l’isolement de la population deviennent de véritables espèces, les nœuds internes de l’arbre représentant le début du processus de spéciation. D’autres lignées ne deviennent pas de véritables espèces mais divergent au fil du temps, ce qui indique que les espèces ne sont pas génétiquement uniformes. Ces lignées sont présentées comme des Unités évolutivement significatives (USE). Le flux génétique inter-lignage est indiqué pour les espèces 1 et 3. Dans ce cas, l’indépendance évolutive peut être transitoire car deux lignées appartenant à la même espèce ne sont pas isolées de manière reproductive. Bien qu’elles restent des lignées distinctes (espèces), l’hybridation interspécifique peut se produire avec un échange génétique limité (changement de couleur) entre les espèces.
La mise en œuvre de stratégies de conservation efficaces visant à protéger à la fois les espèces et la diversité génétique bénéficierait à la fois d’une vision plus cohérente de la façon dont nous délimitons les espèces et les autres unités dans le continuum population-espèces, et de la façon dont ces unités sont interprétées dans différentes législations à l’échelle mondiale. Les nouvelles capacités de la génomique offrent à la fois des opportunités et des défis pour atteindre ces objectifs. Dans ce qui suit, nous considérons: (i) comment les analyses à l’échelle du génome peuvent être conciliées avec une approche conservatrice de la délimitation et de la dénomination des espèces, tout en identifiant les principales composantes de la diversité génétique au sein des espèces, (ii) comment la législation de conservation existante sur plusieurs continents, qui définit les « espèces” pour inclure un large éventail de catégories infra-spécifiques, peut être utilisée compte tenu de la haute résolution offerte par les écrans génomiques, et (iii) comment les connaissances sur les processus de spéciation et la structuration de la variation de la génomique s’alignent sur la législation, puis sur la gestion. Nous suggérons ensuite des moyens d’aligner les unités de conservation sur les approches législatives afin de protéger l’ensemble de la diversité des populations à travers les espèces.
Génomique, Spéciation et taxonomie
Comme dans de nombreux domaines de la science biologique, la systématique, la pratique de nommer les taxons et de comprendre leurs relations et comment ils se forment, est considérablement avancée par la génomique et les modèles statistiques. Que ce soit par séquençage du génome entier (par exemple, Nater et al., 2017) ou l’échantillonnage du génome (SNPs, capture-séquençage de cibles; Jones et Good, 2016; Leache et Oaks, 2017), nous avons maintenant une résolution sans précédent des modèles de diversité génétique (Funk et al., 2012). Couplées à des modèles statistiques de plus en plus puissants pour inférer des histoires de divergence génomique et d’espèces, ces données fournissent de nouvelles informations sur les processus évolutifs qui génèrent des espèces et des variations génétiques.
L’application de ces nouveaux outils génère deux idées opposées. D’une part, de nombreuses études révèlent des populations génétiques très divergentes au sein d’espèces nommées, dites espèces cryptiques (Bickford et al., 2007; Jorger et Schrödl, 2013; Struck et coll., 2017). Ce n’est pas nouveau, car il s’appuie sur une longue histoire de la génétique spatiale des populations et de la phylogéographie moléculaire qui a été une entreprise productive pendant trois décennies (Avise et al., 1987). Ce que la génomique apporte à cet effort, c’est à la fois une résolution accrue de la structure des populations et des données suffisantes pour déduire statistiquement l’historique des populations et tester d’autres modèles de divergence (Degnan et Rosenberg, 2009; Sukumaran et Knowles, 2017). D’autre part, les analyses à l’échelle du génome révèlent également que (comme cela a été reconnu depuis longtemps pour les plantes), les échanges génétiques (introgression) entre espèces animales sont plus fréquents qu’on ne le pensait auparavant, à la fois pendant et après la spéciation, et peuvent même entraîner de nouveaux rayonnements adaptatifs (Mallet, 2007; Rieseberg et Willis, 2007; Arnold et Kunte, 2017) Figure 1. Mises bout à bout, ces idées soulignent en outre que la spéciation est prolongée (Roux et al., 2016) et révèlent les forces opposées opérant à travers le continuum phylogéographie-phylogénétique (Edwards et al., 2016).
Comment tout cela se connecte-t-il avec les objectifs de taxonomie et de conservation? Il existe une préoccupation forte et justifiable que la résolution accrue offerte par les écrans génomiques puisse conduire à un fractionnement excessif des espèces (Isaac et al., 2004), restreignant potentiellement la flexibilité de gestion et reléguant les petites populations génétiquement divergentes à la consanguinité et à l’extinction éventuelle (Frankham et al., 2012). Inversement, Gippoliti et al. (2018) soutiennent que l’inertie taxonomique, qui avait entraîné une incapacité antérieure à reconnaître de nombreuses espèces d’ongulés africains, avait entraîné une dépression de reproduction consécutive au mélange et l’incapacité de protéger des taxons géographiquement restreints. Leur point de vue (et voir aussi Morrison et al., 2009) est qu’il n’y a aucune preuve que l’inflation taxonomique ait des effets négatifs sur la conservation des ongulés (Gippoliti et al., 2018). Une préoccupation connexe est que la taxonomie instable retarde à la fois la pratique de la conservation et les perceptions du public (Garnett et Christidis, 2017). Ces préoccupations sont en partie motivées par le long débat autour des concepts d’espèces et des méthodes de diagnostic en biologie systématique, en particulier l’application du Concept d’espèces phylogénétiques qui soutient que les espèces sont des unités peu diagnostiquables (Cracraft, 1983). Cependant, la majorité des taxonomistes sont intrinsèquement conservateurs, ne nommant les espèces que lorsqu’il y a concordance entre plusieurs sources de données (Dayrat, 2005; Padial et al., 2010; Schlick-Steiner et coll., 2010 : Yeates et coll., 2011). Notre point de vue est que les espèces devraient représenter des lignées robustes en évolution indépendante qui restent en grande partie intactes lorsqu’elles sont en contact avec des parents proches — conformément à l’intention du Concept d’espèce Évolutive (Wiley, 1978) et du Concept d’Espèce de Lignée Généralisée (de Queiroz, 1998, 2007). En pratique, cela nécessite une preuve directe d’isolement reproductif ou de substituts raisonnables de celui-ci, tout en permettant un échange génétique limité (Singhal et al., 2018).
Les données génomiques fournissent une plus grande puissance qu’auparavant pour identifier la différenciation et la divergence au sein et entre les espèces, ce qui nécessite une plus grande considération de la façon dont cela affecte la délimitation des unités taxonomiques. Lorsque les populations sont à la fois fortement divergentes génétiquement et phénotypiquement par rapport aux espèces déjà nommées, il existe de solides arguments pour les nommer comme espèces distinctes. Si les populations sont fortement divergentes sur le plan phénotypique avec peu de séparation génomique, on peut alors juger si ces différences phénotypiques sont (i) héréditaires et (ii) susceptibles de provoquer un isolement reproductif important. Les analyses génomiques peuvent résoudre de telles situations en testant le flux génétique restreint. Le problème le plus difficile est celui de la détection de populations morphologiquement similaires, mais génétiquement divergentes. De telles populations peuvent facilement être diagnostiquées comme des lignées évolutives distinctes en appliquant des méthodes de délimitation statistique à des données multilocales (Carstens et al., 2013; Rannala, 2015), mais s’agit-il de populations éphémères ou d’espèces durables (Sukumaran et Knowles, 2017) ? Il est certainement possible, dans les règles de la nomenclature, de reconnaître des espèces à partir de séquences d’ADN seules, en fournissant des diagnostics basés sur les séquences (Tautz et al., 2003; Cook et coll., 2010). Par exemple, Murphy et coll. (2015) délimitent et décrivent six espèces d’amphipodes de printemps du désert australien sur la base de données de séquence d’ADN malgré l’absence de différences morphologiques claires. Cependant, il s’agit d’un domaine général à améliorer où les systématiciens doivent appliquer le terme « espèces cryptiques” avec plus de cohérence et de rigueur (Struck et al., 2017).
Dans les situations où les données sur les séquences d’ADN constituent une caractéristique déterminante potentielle pour la reconnaissance d’une espèce, nous suggérons qu’il est particulièrement important d’appliquer d’autres sources de données pour éviter une sur-division (Oliver et al., 2015). Une approche consiste à tester un isolement reproductif important lorsque ces populations forment des zones de contact naturelles. Par exemple, dans une analyse génomique comparative des zones de contact entre des paires de lignées de lézards Carlia qui sont phénotypiquement indiscernables, les indices d’isolement génétique étaient fortement corrélés au temps de divergence (Singhal et Moritz, 2013; Singhal et Bi, 2017) (Figure 2). Les plus divergentes de ces paires de lignées ont maintenant été officiellement reconnues comme des espèces distinctes, alors que les lignées plus étroitement apparentées (mais pouvant être diagnostiquées statistiquement) avec des preuves d’échanges génétiques étendus sur le plan génomique entre les zones de contact ne l’étaient pas (Singhal et al., 2018). Compte tenu de la forte relation empirique entre la divergence et l’isolement reproductif chez ces taxons, ces auteurs ont en outre séparé deux isolats allopatriques et cryptiques (d’une espèce étroitement apparentée) qui présentaient une divergence génomique encore plus élevée que ceux délimités ci-dessus. L’extension de cet isolement reproducteur à d’autres espèces congénères ayant des lignées phylogéographiques profondément divergentes mais morphologiquement similaires (Potter et al., 2016, 2018; Afonso Silva et coll., 2017a) suggère qu’il reste encore plus d’espèces à décrire, en particulier sur les îles (Figure 2, Carlai amax et C. rufilatus), et en effet, certaines de ces espèces ont été décrites suite à une analyse phénotypique complémentaire (Afonso Silva et al., 2017b). D’autres lignées avec des divergences plus récentes, mais toujours délimitées statistiquement en tant que lignées distinctes utilisant un grand ensemble de gènes nucléaires, peuvent être représentées comme des Unités évolutivement significatives (USE) au sein des espèces.
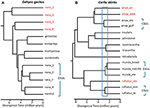
Figure 2. Phylogenomic resolution of species boundaries in lizards from the Australian Monsoonal Tropics (AMT). Dans chaque cas, les relations et les temps de divergence relative sont estimés à partir de 100 exons en utilisant la coalescence multi-espèces mise en œuvre dans StarBEAST2, mais les échelles de temps entre les deux exemples ne sont pas nécessairement les mêmes. (A) Résultats pour le groupe nana des geckos Gehyra (Moritz et al., 2018). La diversité phylogénétique des lignées au sein de G. nana, telle que précédemment reconnue, comprenait quatre autres espèces déjà décrites et morphologiquement distinctes. Des analyses ultérieures des chevauchements d’aires de répartition et de la morphologie ont conduit à la reconnaissance de chacune des lignées les plus divergentes phylogénétiquement (étiquettes de taxons rouges) en tant qu’espèces distinctes (Doughty et al., 2018). Les lignées (morphologiquement inséparables) restantes chez G. nana telles que maintenant reconnues peuvent être considérées comme des ESUs. (B) Résolution continue des limites des espèces chez les scinques de Carlia à partir de l’AMT. Certaines lignées ont déjà été reconnues comme espèces sur la base d’une combinaison de délimitation d’espèces génétiques et d’identification post hoc de différences morphologiques diagnostiques (C. insularis et C. isostriacantha; Afonso Silva et coll., 2017b). Pour d’autres complexes (Potter et al., 2016, 2018), des révisions taxonomiques sont en cours. Le rectangle bleu indique la plage de temps de divergence où un isolement reproductif important, corrélé à la divergence, a été observé dans les zones de contact entre des lignées morphologiquement cryptiques d’espèces apparentées (Singhal et Bi, 2017). Conservativement, et suivant (Singhal et al., 2018), nous reconnaissons comme espèces candidates trois lignées génétiques avec des divergences supérieures à ce seuil empirique. Cela laisserait des ESUs génétiquement distincts dans chacune des Carlia amax, Carlia munda et Carlia rufilatus (parenthèses bleues). Les barres d’échelle représentent des estimations du temps de divergence en millions d’années.
Dans les cas extrêmes, les méthodes génomiques révèlent que les espèces taxonomiques uniques sont non seulement génétiquement hétérogènes, mais, au sens phylogénétique, contiennent également d’autres espèces nommées. Une telle « paraphylie » peut survenir par une spéciation récente d’une seule espèce génétiquement subdivisée (Patton et Smith, 1994), mais elle génère également l’hypothèse que le taxon génétiquement hétérogène peut représenter plusieurs espèces. Encore une fois, plus de preuves sont nécessaires. À titre d’exemple, l’analyse phylogénomique d’un complexe de lézards gecko de Gehyra du nord de l’Australie a délimité neuf lignées évolutives, avec quatre espèces déjà nommées intégrées phylogénétiquement parmi elles (Moritz et al., 2018; Figure 2). Des preuves ultérieures de chevauchement géographique (tout en conservant la séparation phylogénétique) et de différences morphologiques subtiles ont soutenu la dénomination des quatre lignées les plus divergentes phylogénétiquement (Doughty et al., 2018). Inversement, les lignées les plus étroitement apparentées (mais statistiquement séparables) avaient des distributions adjacentes plutôt que chevauchantes, mais n’étaient pas diagnostiquables morphologiquement et, par conséquent, ont été conservées comme une seule espèce (G. nana sensu stricto), bien qu’avec de multiples USE, en attendant des recherches plus approfondies (figure 2). Des analyses du génome ultérieures d’échantillons à travers les limites de ces UDE ont révélé qu’au moins certains ont des échanges génétiques substantiels, validant l’approche originale et conservatrice adoptée pour reconnaître les espèces.
Dans d’autres cas, les études de génomique végétale, même par simple ordination de SNP, peuvent révéler des divergences marquées et imprévues entre les échantillons de population, suggérant la présence de taxons cryptiques qui peuvent ensuite être analysés pour des caractères phénotypiques diagnostiques. Par exemple, des preuves basées sur le SNP pour la divergence génomique entre les populations et les différences écotypiques chez Eucalyptus salubris ont révélé deux lignées moléculaires distinctes qui ont conservé leur caractère distinctif dans une zone de chevauchement géographique, l’adaptation écotypique étant considérée comme un facteur important pour minimiser le flux de gènes entre les lignées. (Steane et coll., 2015; Figure 3). Dans un autre cas, dans le pélargonium australien, l’analyse génomique, combinée à des études morphologiques et reproductives, a conduit à des changements significatifs dans la taxonomie, à la fois la division et le regroupement des taxons, en particulier lorsque les traits morphologiques précédemment utilisés se révèlent labiles et discordants avec la phylogénie (Nicotra et al., 2016).
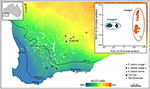
Figure 3. L’analyse génomique à l’aide de données SNP a permis d’identifier des lignées cryptiques dans l’Eucalyptus salubris répandu. L’emplacement des populations d’échantillons de chaque lignée est indiqué à travers un gradient d’indice d’aridité dans le sud-ouest de l’Australie-occidentale. Le PCoA basé sur des distances génétiques binaires dérivées de 16 122 marqueurs DArTseq, montre deux lignées distinctes. Adapté de Steane et al. (2015).
Les cas ci-dessus illustrent l’approche progressive de test d’hypothèses qui a été utilisée en taxonomie intégrative (Padial et al., 2010; Yeates et coll., 2011). De plus, ces cas utilisent une certaine forme de séquençage de réduction du génome pour obtenir des données de haute qualité à partir de 1000 de gènes indépendants, ce qui donne une confiance beaucoup plus grande dans les résultats que ce n’est le cas en utilisant uniquement des locus plastidiens ou quelques gènes nucléaires. Un exemple de la façon dont l’application de la taxonomie intégrative peut modifier considérablement les perceptions de la diversité provient d’une étude génétique, acoustique et morphologique combinée de grenouilles de Madagascar qui a presque doublé la diversité des espèces connues (Vieites et al., 2009). Ces auteurs ont proposé une série de termes reflétant l’état de résolution taxonomique et de congruence: Espèces candidates confirmées, pour des taxons génétiquement et phénotypiquement divergents, mais non encore nommés; Espèces candidates non confirmées pour des taxons génétiquement divergents dont la morphologie et l’acoustique n’avaient pas encore été examinées; et Lignées Profondément Conspécifiques pour des entités connues pour être génétiquement divergentes mais phénotypiquement cryptiques. Les analyses génomiques n’ont pas encore été appliquées à ces taxons, mais elles pourraient certainement éclairer les décisions concernant les lignées conspécifiques par rapport aux espèces candidates.
Les approches génomiques, même appliquées de manière conservatrice, conduiront sans aucun doute à une nouvelle division des taxons morphologiquement conservateurs. Mais, inversement, il existe des exemples clairs où les mêmes approches révèlent des espèces sur-divisées ou mal diagnostiquées (Nicotra et al., 2016). Bien sûr, il existe déjà de nombreuses études moléculaires sur les complexes d’espèces végétales et animales basées sur des ensembles de données à plus petite échelle (par ex., microsatellite, ADNmt et adNPC) qui ont identifié des espèces mal diagnostiquées ou des espèces multiples qui ont été circonscrites taxonomiquement mais qui sont maintenant considérées comme une seule espèce. Par exemple, Rabosky et al. (2014) ont utilisé les phylogénies des gènes mitochondriaux et nucléaires pour réviser le complexe scincide Ctenotus inornatus, ce qui a entraîné une réduction nette des espèces et un réalignement des limites des espèces; les traits morphologiques appliqués précédemment, bien que pratiques pour les biologistes de terrain, étaient très labiles et ne correspondaient en grande partie pas aux limites des espèces telles que diagnostiquées génétiquement. Chez les plantes, certains des exemples les plus connus se trouvent dans les orchidées, comme le genre d’orchidées européennes Ophrys et le genre d’orchidées australiennes Caladenia, où les études moléculaires indiquent nettement moins d’entités qui seraient reconnues comme espèces, que celles basées sur des traits morphologiques et des syndromes de pollinisation pseudocopulatoires (voir Devey et al., 2008; Swarts et coll., 2014).
Les données à l’échelle du génome, lorsqu’elles sont interprétées de manière prudente et idéalement avec des analyses parallèles de phénotypes, continueront de conduire à des changements taxonomiques, en particulier dans les taxons morphologiquement conservateurs (Fišer et al., 2018). Mais, contrairement aux préoccupations exprimées par certains (Garnett et Christidis, 2017), nous considérons qu’il s’agit de la poursuite appropriée de la systématique évolutive, même si les résultats sont temporairement gênants.
Génomique et Unités de conservation intraspécifiques
Bien que moins controversée que la délimitation taxonomique et le débat au niveau des espèces, mais à bien des égards tout aussi complexe, la discussion importante sur la définition et la délimitation des unités de conservation en tant qu’éléments importants de la diversité intraspécifique qui doivent être pris en compte dans les actions de conservation (Ryder, 1986; Waples, 1991; Dizon et al., 1992; Moritz, 1994; Vogler et de Salle, 1994; Crandall et coll., 2000; Fraser et Bernatchez, 2001; Avise, 2004; Funk et coll., 2012) (Figure 4). Compte tenu de l’approche prudente de reconnaissance des espèces à partir de preuves génomiques que nous préconisons ci-dessus (et voir aussi Oliver et al., 2015; Singhal et coll., 2018), il est inévitable que certaines espèces taxonomiques contiennent encore des populations longtemps isolées en leur sein. Des concepts tels que les Unités Évolutivement significatives (UDE) ou les Unités Désignables (UDE) ont été proposés pour compléter la taxonomie existante (Ryder, 1986; Waples, 1991; Moritz, 1994; Mee et al., 2015). Comme pour les espèces, les interprétations et les critères varient, mais l’intention commune est de reconnaître les principaux éléments de la diversité intraspécifique pour les actions de conservation (Funk et al., 2012). Moritz (1994, 2002) a préconisé une approche moléculaire simpliste mais pratique pour identifier des segments évoluant indépendamment au sein des espèces, l’axe « historique” de la diversité; tandis que d’autres ont mis davantage l’accent sur l’axe « adaptatif” pour inclure la diversité fonctionnelle (Ryder, 1986; Crandall et al., 2000; Fraser et Bernatchez, 2001; Waples et coll., 2001; Mee et coll., 2015) (Figure 4). Quels que soient les mérites relatifs de ces alternatives, il est clair que les outils techniques et analytiques de la génomique des populations peuvent maintenant identifier de manière robuste les deux composantes de la diversité intraspécifique (Funk et al., 2012; Hoban et coll., 2013; Whitlock, 2014; Yang et Rannala, 2014).
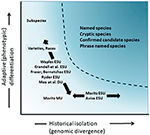
Figure 4. Séparation de la diversité génétique en deux composantes: variation adaptative résultant de la sélection naturelle, et mesurée par l’analyse des phénotypes et de l’isolement historique ou de la divergence neutre évaluée par la divergence génomique. La zone ombrée plus foncée au-dessus de la courbe indique les conditions dans lesquelles les populations sont susceptibles d’être considérées comme des espèces distinctes selon la plupart des concepts. Divers concepts d’unités de conservation et catégories taxonomiques infra-spécifiques sont présentés par rapport aux deux axes de la diversité génétique (d’après Moritz, 2002).
Cela conduit à la question: pourquoi ne pas simplement nommer ESUs comme sous-espèce? Les sous-espèces sont traditionnellement définies et considérées par les botanistes et les zoologistes comme des ensembles allopatriques phénotypiquement distincts de populations qui peuvent s’intercaler les unes dans les autres aux frontières géographiques (Mayr, 1942; Grant, 1981). En tant que tels, ils reflètent potentiellement une divergence adaptative sans nécessairement avoir été historiquement isolés. Les désignations de sous-espèces sont largement utilisées chez les plantes, les différences géographiques et écologiques étant le principal moyen de les distinguer (Hamilton et Reichard, 1992). Dans la mesure où les sous-espèces manifestent une adaptation locale, avec ou sans flux génétique continu, nous suggérons que les sous-espèces définies phénotypiquement devraient continuer à être reconnues dans la conservation, et non confondues avec les USE, du moins pas les USE telles que définies pour représenter des populations historiquement isolées.
De la même manière que la génomique des populations fournit une précision beaucoup plus grande dans l’identification des UDE en utilisant à la fois des locus neutres et adaptatifs, elle fournit également un moyen d’utiliser un grand nombre de locus neutres pour délimiter plus facilement les Unités de gestion (UDE) en tant qu’unités démographiquement indépendantes caractérisées par un flux génétique restreint (Funk et al., 2012). Alors que nous nous concentrons principalement sur les UDE et les unités analogues telles que les UD qui peuvent être reconnues comme les principales unités intraspécifiques de conservation et qui ont été acceptées pour la protection dans certaines juridictions, des UD ont également été identifiées représentant des ensembles de populations démographiquement indépendants (voir Palsbøll et al., 2007) qui peuvent être gérés pour conserver l’UDE plus importante (Moritz, 1994; Funk et al., 2012). Les unités de gestion peuvent jouer un rôle important dans la surveillance et la gestion des populations d’espèces et ont également été appelées » stocks « , particulièrement pertinentes pour la gestion des pêches (Avise, 2004; Laikre et al., 2005). La définition de MUs par Moritz (1994) comme » populations présentant une divergence significative de fréquences d’allèles aux loci nucléaires ou mitochondriaux, indépendamment du caractère distinctif phylogénétique des allèles ” est généralement largement acceptée, bien que, comme l’ont souligné Palsbøll et al. (2007), la connectivité démographique est le facteur clé et non le niveau de flux génétique historique.
La délimitation des unités de conservation en tant qu’éléments importants de la diversité intraspécifique a également des implications importantes pour le sauvetage génétique de petites populations consanguines par la restauration du flux génétique ou par augmentation et il devient de plus en plus clair que le sauvetage génétique doit être envisagé plus largement si l’on veut éviter une extinction accrue de la population (Love Stowell et al., 2017; Ralls et coll., 2018). Il a été proposé par Moritz (1999) que le mélange d’UDE mais pas d’UDE pourrait être considéré comme une stratégie appropriée pour le sauvetage génétique. Comme l’ont souligné Frankham et coll. (2012) le mélange d’espèces reconnues sur le plan taxonomique ne serait généralement pas considéré comme acceptable par les gestionnaires, auquel cas une sur-répartition taxonomique pourrait restreindre les possibilités de sauver de petites populations consanguines. Nous suggérons qu’une approche plus rigoureuse de la délimitation des unités intraspécifiques telles que les UM pourrait aider à atténuer ce problème, mais il est également clair que l’estimation génomique des taux de migration et de la divergence adaptative a le potentiel de fournir des conseils beaucoup plus solides non seulement pour définir les limites des espèces et la variation intraspécifique, mais aussi pour concevoir des stratégies de sauvetage génétique.
Phylogénomique et conservation
La génomique combinée à des méthodes phylogénétiques peut facilement résoudre les relations et la distance évolutive entre les taxons, ainsi que la délimitation des espèces. Il est préférable d’y parvenir en utilisant des méthodes d’arbres d’espèces, plutôt que la concaténation, car les premières tiennent compte de l’hétérogénéité des arbres géniques et évitent le gonflage excessif des longueurs des pointes en raison du polymorphisme ancestral (Edwards et al., 2016; Ogilvie et coll., 2016). Bien qu’un nombre massif de loci ne soit souvent pas essentiel, le fait d’inclure plus de loci améliore souvent la résolution des relations et de la longueur des branches (par exemple, Blom et al., 2016). L’importance pour la conservation est double. Premièrement, les évaluations phylogénétiques permettent de prendre en compte le caractère distinctif des espèces ou des UDE, et pas seulement de savoir si elles sont décrites. Ainsi, le programme EDGE (Isaac et al., 2007) préconise la pondération des espèces pour la conservation en fonction de l’histoire évolutive unique qu’elles représentent, en plus des critères habituels de l’UICN. Deuxièmement, les longueurs de branches elles-mêmes peuvent représenter des valeurs de biodiversité, indépendamment de la taxonomie (Faith, 1992), ce qui a démontré une valeur dans la hiérarchisation des zones de conservation (Laity et al., 2015; Rosauer et coll., 2016, 2018). En ce sens, l’adoption de métriques phylogénétiques utilisant des mesures telles que la diversité phylogénétique et l’endémisme phylogénétique permet de mettre en évidence des zones de grande diversité évolutive qui peuvent être ciblées pour la protection, et être prioritaires dans tout processus de planification pour une meilleure conservation de la biodiversité. Cette approche peut améliorer la protection de la diversité génétique et des processus évolutifs pour divers groupes d’organismes à différentes échelles géographiques (Laity et al., 2015) prenant des décisions sur des domaines à prioriser plus robustes aux aléas de la façon dont les preuves génomiques sont traduites en reconnaissance d’espèces ou d’unités intra-spécifiques.
Le monde réel — Définitions législatives
Les cadres politiques et législatifs pour la conservation de la biodiversité vont des conventions et stratégies internationales aux stratégies et législations nationales, en passant par les législations et stratégies nationales ou régionales au sein des pays. Les principaux instruments internationaux sur la conservation biologique comprennent la Convention sur la Diversité Biologique mise en œuvre par le biais de la Convention des Parties (COP), la Convention du Patrimoine Mondial, la Stratégie Mondiale pour la Biodiversité, le Processus de Montréal pour la Conservation des forêts, la Convention de Ramsar sur les Zones humides, la Convention sur les Espèces migratrices, la Convention sur le Commerce International des Espèces Menacées (CITES), la Liste rouge de l’Union Internationale pour la Conservation de la Nature (UICN) sur les espèces menacées et la Convention-Cadre des Nations Unies sur les changements climatiques. Toutes ces chartes incarnent le principe de la conservation des espèces et de la diversité génétique comme objectif fondamental de la conservation de la biodiversité, les espèces étant l’unité largement définie pour la protection. Cependant, les définitions des espèces dans la législation et les directives internationales sont extrêmement variables. Ils intègrent la reconnaissance taxonomique des espèces et des taxons intraspécifiques, mais peuvent également intégrer des concepts génétiques/évolutifs qui sont souvent couverts par la définition juridique d’une « espèce” (tableau 1).

Tableau 1. Législation et directives mondiales définissant les » espèces » à inscrire et à protéger.
Compte tenu de la large gamme de niveaux infra-spécifiques pouvant être protégés (tableau 1), la législation et les processus d’inscription associés des pays devraient faciliter la conservation de la diversité génétique. Par exemple, la définition d’une » espèce ”, lorsqu’on considère la législation dans le monde (tableau 1), peut inclure: les espèces, sous-espèces, variétés et populations géographiquement et / ou génétiquement distinctes, tandis qu’en vertu de la législation sud-africaine, elles peuvent également inclure des cultivars, des races géographiques, des souches et des hybrides. Ainsi, dans la plupart des lois à l’échelle mondiale, une « espèce” comprend des entités qui reflètent divers niveaux infra-spécifiques qui font tous partie d’une distribution plus large des espèces et reflètent à des degrés divers le continuum population-espèce. Cela est parallèle à la difficulté inévitable que rencontrent les taxonomistes, les biologistes de l’évolution et les généticiens de la conservation pour trouver une manière plus cohérente et uniforme de spécifier des unités dans ce continuum.
Certains soutiennent que les préoccupations concernant les implications politiques des espèces et l’incertitude infra-spécifique sont mal placées (Hey et al., 2003), mais nous sommes toujours d’avis que même si la législation peut fournir un mécanisme de protection de la diversité génétique à divers niveaux, il incombe aux biologistes de fournir plus de clarté et de cohérence sur la façon dont ces niveaux devraient être délimités des espèces aux populations individuelles. À l’heure actuelle, les gestionnaires et les législateurs se retrouvent avec un éventail déconcertant de concepts d’espèces et d’unités infra-spécifiques fournis par les scientifiques lors de la délimitation des unités à travers le continuum population-espèce. Par exemple, toutes les administrations considèrent les espèces reconnues par les taxonomistes comme des unités clés pour la conservation (tableau 1), mais il y a encore un large débat entre les taxonomistes autour des concepts d’espèces, et la façon dont les espèces sont diagnostiquées dans la pratique varie au sein des groupes et d’un groupe à l’autre. Cela a conduit à un appel récent à une approche plus réglementée de la taxonomie (Garnett et Christidis, 2017), auquel d’autres ont répliqué que la réglementation de la taxonomie n’était pas la réponse (Thomson et al., 2018). Bien que nous soyons d’accord avec certains des points soulevés par Thomson et coll. (2018) nous suggérons qu’il est plutôt naïf de proposer que la législation elle-même doive changer pour faire explicitement référence au concept de taxon spécifique impliqué par un nom. Ces débats et les précédents de ce type soulignent la nécessité pour les biologistes évolutionnistes et les taxonomistes de prendre l’initiative d’assurer la clarté (transparence) de la manière dont ils traduisent les preuves génomiques et autres en délimitation d’espèces ou d’unités intraspécifiques, et comment ces taxons se rapportent à la législation pertinente pour la conservation et la protection.
Traduction vers la gestion
Comme nous l’avons vu plus haut, toutes les lois et directives reconnaissent le niveau des espèces comme l’unité fondamentale de conservation, mais prévoient également la reconnaissance de diverses unités infra-spécifiques. Beaucoup ont également une exigence implicite pour la désignation officielle et la description taxonomique des espèces lorsqu’elles sont considérées pour la priorisation de la conservation et la protection formelle. Par exemple, en vertu des Lignes directrices de l’UICN sur la liste rouge, l’inscription d’espèces non nommées est déconseillée et il est nécessaire que la nouvelle description de l’espèce soit publiée dans les quatre ans suivant l’inscription de l’espèce sur la Liste rouge de l’UICN ou qu’elle soit supprimée. Cela met en évidence l’importance de la dénomination taxonomique et de la description des entités, aux niveaux des espèces et infra-spécifiques, les informations taxonomiques limitées pour de nombreux groupes de plantes et d’animaux (Mace, 2004; Oliver et Lee, 2010; Wilson, 2017), et la nécessité d’accélérer l’effort taxonomique, en particulier dans les zones riches en biodiversité (Riedel et al., 2013; Wege et coll., 2015; Wilson, 2017).
Les espèces nommées taxonomiquement et les catégories taxonomiques intraspécifiques, telles que les sous-espèces et les variétés, constituent généralement les unités de base pour l’inscription formelle, la hiérarchisation de la conservation et la protection, mais dans certaines juridictions, d’autres catégories d’unités de conservation intraspécifiques sont officiellement reconnues. Ceux-ci sont généralement identifiés à l’aide de données génomiques et phénotypiques combinées. Par exemple, la Loi sur les espèces en péril des États-Unis reconnaît des segments de population distincts qui sont largement fondés sur les UDE (Waples, 1991), tandis que la Loi sur les espèces en péril du Canada reconnaît les UDE. Interprétation opérationnelle des » populations distinctes » en vertu de la Loi canadienne sur les espèces en péril, les UD visent à identifier des ensembles distincts de populations qui représentent des composantes importantes de l’héritage évolutif des espèces et qui sont peu susceptibles d’être remplacées par la dispersion naturelle (Mee et al., 2015).
Les unités DH et DPSS se chevauchent conceptuellement avec les unités ESU (Waples et al., 2013) et ont été appliquées le plus largement aux poissons d’Amérique du Nord, notamment aux salmonidés du nord-ouest des États-Unis (Waples, 1995) et aux corégonidés canadiens (Mee et al., 2015) Il est intéressant de noter que les DPSS aux États-Unis ne s’appliquent qu’aux vertébrés, tandis qu’au Canada, les DHs peuvent s’appliquer à tout organisme pouvant être répertorié en vertu de sa Loi (voir Waples et al., 2013), avec des unités DH actuellement répertoriées pour les arthropodes, les mollusques et les plantes. En Australie, comme au Canada, des populations distinctes peuvent être répertoriées pour tout organisme reconnu en vertu de la Loi de 1999 sur la protection de l’environnement et la conservation de la biodiversité (Loi EPBC), bien qu’il ne s’agisse pas d’une catégorie spécifiquement nommée, telle que DUSs et DPSs. Cependant, à ce jour, ils n’ont été reconnus que pour les vertébrés. Aux États-Unis, il y a 127 DPS de vertébrés répertoriés pour la conservation (https://ecos.fws.gov/ecp/); au Canada, 70 DHs ont été répertoriés pour tous les organismes (http://www.registrelep-sararegistry.gc.ca/species/schedules_e.cfm?id=1); et en Australie, les populations de 7 espèces de vertébrés couvrant les mammifères, les poissons et les oiseaux ont été reconnues comme ayant un statut de conservation spécifique en vertu de la loi EPBC (http://www.environment.gov.au/cgi-bin/sprat/public/publicthreatenedlist.pl). Alors que les évaluations de ces populations aux États-Unis et au Canada utilisent généralement de l’information génétique (Waples et al., 2013), en Australie, ils sont généralement basés sur l’identification géographique et juridictionnelle, bien que dans certains de ces cas, ils aient été étayés par une analyse de la différenciation génétique des mammifères (Firestone et al., 1999; Armstrong, 2006) et fish (Colgan et Paxton, 1997; Ahonen et al., 2009).
La liste des unités de conservation infra-spécifiques pour les plantes et les invertébrés au Canada (DHs), mais pas aux États-Unis ou en Australie, met en évidence une autre question qui n’est pas spécifiquement liée à la délimitation des unités de conservation, mais plus largement à des incohérences importantes dans la priorisation de la conservation de la biodiversité; les vertébrés emblématiques tels que les oiseaux, les mammifères et les poissons sont au centre de nombreuses juridictions, alors que d’autres organismes ne le sont pas. Cette inscription préférentielle des populations de certains organismes reflète une série de questions qui relèvent moins de la science de la délimitation des unités de conservation que de l’inscription des populations basée sur la sensibilisation du public, l’opportunité politique et la nature charismatique de l’organisme à inscrire (voir Waples et al., 2013).
Alors qu’en Australie, certaines populations de mammifères, d’oiseaux et de poissons bien connus sont protégées en vertu de la loi EPBC, il existe maintenant un corpus important d’informations basées sur des études moléculaires chez les reptiles, les invertébrés et les plantes où les populations peuvent être reconnues comme des UDE ou des UDE, et bon nombre d’entre elles sont importantes pour la conservation en raison de leur aire géographique étroite, de leur petite taille, de leur taux de déclin et de leur sensibilité aux processus menaçants. Par exemple, des exemples frappants de populations très structurées et de lignées profondément divergentes au sein d’espèces qui répondraient facilement aux exigences d’une UDE peuvent être trouvés dans une gamme de vertébrés à faible dispersion des tropiques (figure 2) et ailleurs. De même chez les invertébrés australiens, comme les amphipodes et les mille-pattes, une structure génétique importante et des modèles phylogéographiques soutiennent la délimitation de groupes de populations discrets sous forme d’USE géographiquement disparates, avec des aires de répartition géographiques étroites, limitées à des habitats spécifiques et représentées par seulement quelques populations (Finston et al., 2007; Murphy et coll., 2013; Nistelberger et coll., 2014a). Souvent, ces espèces endémiques à courte distance sont des taxons préoccupants pour leur conservation (Harvey et al., 2011) et sont situés dans des points chauds de l’endémisme (par exemple, Oliver et al., 2017), qui sont elles-mêmes des priorités élevées pour la conservation de plusieurs taxons (Rosauer et al., 2018), comme la région de Kimberley et la chaîne de Stirling dans le sud-ouest de l’Australie-Occidentale.
Chez les plantes australiennes, il existe également un nombre important d’études génétiques et phylogéographiques de population identifiant des groupes de population distincts au sein d’une gamme d’espèces de différents genres reflétant des systèmes de population disjoints et historiquement isolés, des complexités géologiques et édaphiques, et occupant des habitats contrastés en termes de végétation et de climat (voir Broadhurst et al., 2017). Des populations génétiquement distinctes sont particulièrement évidentes chez les espèces du sud-ouest de l’Australie-occidentale (Coates, 2000; Byrne et al., 2014) avec un certain nombre d’études mettant en évidence une structure génétique significative et des lignées très divergentes dans une gamme de genres et de familles de plantes, notamment: Acacia (Mimosaceae; Coates, 1988), Atriplex (Amaranthaceae; Shepherd et al., 2015), Banksia (Proteaceae; Coates et al., 2015), Lambertia (Proteaceae; Byrne et al., 2002), Hakea (Proteaceae; Sampson et al., 2014), Eremophila (Scrophulariaceae; Llorens et al., 2015), Pultanaea (Fabaceae; Millar et Byrne, 2013), et Calothamnus (Myrtaceae; Nistelberger et al., 2014b). Dans de nombreux cas, ces populations sont reconnues dans la planification de la conservation par les agences gouvernementales de l’État et prises en compte dans les évaluations d’impact environnemental (voir Shepherd et al., 2015) même s’ils ne sont pas inscrits sur la liste des produits protégés par la législation. Cependant, nous reconnaissons que la reconnaissance officielle de ces unités améliorerait leur conservation à long terme.
Ces exemples australiens soulignent le besoin croissant de reconnaître cette couche de diversité génétique en dessous du niveau de l’espèce. Le défi consiste à faire en sorte que les populations hautement structurées et les lignées profondément divergentes qui sont préoccupantes pour la conservation soient protégées face à des demandes concurrentes d’attention à la conservation. Au-delà des espèces emblématiques, ce défi est également évident à l’échelle mondiale, à l’exception du Canada. Le fait de ne pas protéger des unités de conservation clairement délimitées augmentera probablement le risque d’extinction cryptique et de perte d’une diversité génétique importante, ce qui, dans de nombreux cas, peut impliquer des lignées et des populations historiquement isolées qui persistent au sein des espèces depuis des millions d’années (Moritz, 2002).
Conclusions et recommandations
L’inscription d’espèces et de diverses catégories infra-spécifiques dans la législation sur les espèces menacées ou dans les organisations internationales telles que l’UICN, indique un engagement à protéger la diversité génétique dans le continuum des populations d’espèces. Pourtant, les approches actuelles de conservation de la biodiversité se concentrent en grande partie sur la fin des espèces de ce continuum avec moins d’attention accordée à la diversité génétique et à la variation infra-spécifique. Cette focalisation plus limitée sur la variation génétique au niveau intra-espèce suggère que, bien que les gestionnaires de la conservation reconnaissent de plus en plus la valeur de la prise en compte explicite de l’information génétique dans les actions de gestion et la priorisation, ils manquent souvent d’informations exploitables de la part des scientifiques de la biodiversité (Waples et al., 2008; Laikre et coll., 2010; Cook et Sgrò, 2017; Taylor et coll., 2017). Cet écart entre la science et les pratiques de conservation empêche une approche globale plus efficace de la conservation et de la gestion de la diversité génétique.
Nous suggérons que le manque de clarté des scientifiques de la biodiversité sur les unités de conservation repose en grande partie sur des incohérences à deux niveaux. La première concerne les concepts d’espèces multiples et les diverses méthodes de diagnostic utilisées pour délimiter les espèces, ce qui crée des incohérences entre les groupes taxonomiques. La seconde concerne un manque de cohérence et d’accord des taxonomistes, des biologistes de l’évolution et des biologistes de la conservation sur la façon de traiter les taxons infra-spécifiques et d’autres entités telles que les USE, les DUS et les MUs.
Bien que nous considérions que la législation est souvent déjà suffisamment flexible en termes de définition des taxons (ou « espèces”), nous soutenons qu’il est nécessaire de traiter systématiquement le continuum des espèces de population et de définir plus clairement les unités de conservation en tenant davantage compte de concepts tels que les UDE et de leur lien avec la législation pertinente. Nous soulignons que cette question deviendra sans doute plus critique étant donné le pouvoir de la génomique de détecter la différenciation à des échelles progressivement plus fines et son utilisation croissante pour délimiter des espèces et des entités infra-spécifiques.
De l’analyse précédente, nous formulons les recommandations suivantes:
(i)Les données génomiques doivent être interprétées de manière prudente lors de la délimitation des espèces, en utilisant une approche taxonomique intégrative qui inclut généralement des preuves au-delà des seules données génétiques.
(ii) Lorsque les généticiens identifient plusieurs lignées ou populations distinctes au sein d’une espèce, et que celles-ci ne justifient pas une élévation au niveau de l’espèce et ne sont pas reconnues taxonomiquement, qu’il y a une impulsion plus forte et une approche plus uniforme pour identifier des unités intraspécifiques clairement justifiées pour l’évaluation pratique de l’état de conservation ou des évaluations d’impact, et que celles-ci soient explicitement reconnues comme unités de conservation dans le contexte de la législation pertinente pour cette juridiction.
(iii) L’UICN, par l’intermédiaire de son Groupe de spécialistes de la génétique de la conservation et de ses sous-groupes régionaux, envisage d’élaborer des lignes directrices pour la reconnaissance et l’étiquetage des unités intraspécifiques conformes aux meilleures pratiques scientifiques.
(iv) La vision plus nuancée émergeant de la génomique évolutive des espèces et du continuum des populations aux espèces devrait être communiquée plus efficacement par les scientifiques aux gestionnaires de la conservation et aux décideurs politiques.
Plus largement, nous faisons écho aux recommandations formulées récemment dans un certain nombre de revues (Frankham, 2010; Santamaría et Méndez, 2012; Hoban et al., 2013; Moritz et Potter, 2013; Laity et al., 2015; Pierson et coll., 2016; Cook et Sgrò, 2017; Taylor et coll., 2017) qui soulignent la nécessité pour les scientifiques d’améliorer la communication et de sensibiliser les décideurs, les agences gouvernementales, les organisations non gouvernementales et les praticiens que les résultats de la systématique moléculaire, de la taxonomie moléculaire, de la phylogéographie et de la recherche sur la génétique des populations peuvent fournir des informations essentielles sur la conservation de la diversité génétique et la gestion des processus évolutifs.
Contributions de l’auteur
DC, MB et CM ont tous contribué à la préparation et à la révision du manuscrit.
Déclaration sur les conflits d’intérêts
Les auteurs déclarent que la recherche a été menée en l’absence de relations commerciales ou financières pouvant être interprétées comme un conflit d’intérêts potentiel.
Remerciements
Ce projet a été soutenu par l’Australian Research Council (subvention ARC Linkage No. LP120200063), l’Université Nationale Australienne et le Département de la Biodiversité, de la Conservation et des Attractions de l’Australie Occidentale.
Afonso Silva, A. C., Bragg, J. G., Potter, S., Fernandes, C., Coelho, M. M. et Moritz, C. (2017a). Spécialiste tropical contre généraliste du climat: diversification et histoire démographique des espèces sœurs de scinques Carlia du nord-ouest de l’Australie. Mol. Ecol. 26:4045–4058. doi: 10.1111/mec.14185
CrossRef Texte intégral / Google Scholar
Afonso Silva, A. C., Santos, N., Ogilvie, H. A., et Moritz, C. (2017b). Validation et description de deux nouveaux scinques arc-en-ciel du nord-ouest de l’Australie avec des méthodes et une morphologie coalescentes multiespèces. PeerJ 5: e3724. doi: 10.7717 / peerj.3724
Résumé PubMed / Texte intégral croisé /Google Scholar
Ahonen, H., Harcourt, R. G., et Stow, A. J. (2009). L’ADN nucléaire et mitochondrial révèle l’isolement des populations de requins nourrices gris en péril (Carcharias taurus). Mol. Ecol. 18, 4409–4421. doi: 10.1111/j.1365-294X.2009.04377.x
Résumé publié par PubMed / Texte intégral croisé / Google Scholar
Armstrong, K. N. (2006). Structure phylogéographique chez Rhinonicteris aurantia (Chiroptera: Hipposideridae): implications pour la conservation. Acta Chiropterologica 8, 63-81. doi: 10.3161/1733-5329(2006)82.0.CO
CrossRef Texte intégral |Google Scholar
Arnold, M. L., et Kunte, K. (2017). Échange génétique adaptatif: une histoire enchevêtrée de mélange et d’innovation évolutive. Tendances Ecol. Evol. 32, 601–611. doi: 10.1016/j. arbre.2017.05.007
Résumé PubMed / Texte intégral croisé / Google Scholar
Avise, J.C. (2004). Marqueurs Moléculaires, Histoire naturelle et Évolution. 2e Éd. Sunderland, MA; Oxford: Oxford University Press.
Google Scholar
Avise, J. C., Arnold, J., Ball, R. M., Bermingham, E., Lamb, T., Neigel, J. E., et al. (1987). Phylogéographie intraspécifique: le pont de l’ADN mitochondrial entre la génétique des populations et la systématique. Ann. Rév. Ecol. Sys. 18, 489–522. doi: 10.1146/annurev.es.18.110187.002421
CrossRef Texte intégral|Google Scholar
Bickford, D., Lohman, D. J., Sodhi, N. S, Ng, P. K., Meier, R., Winker, K., et al. (2007). Les espèces cryptiques comme fenêtre sur la diversité et la conservation. Tendances Ecol. Evol. 22, 148–155. doi: 10.1016/j. arbre.2006.11.004
Résumé PubMed | Texte intégral CrossRef /Google Scholar
Blom, M. P. K., Bragg, J. G., Potter, S. et Moritz, C. (2016). Accounting for uncertainty in gene tree estimation: summary – coalescent species tree inference in a challenging radiation of Australian lizards. Syst. Biol. 66, 352–366. doi: 10.1093/sysbio/syw089
Résumé publié/ Texte intégral croisé / Google Scholar
Broadhurst, L., Breed, M., Lowe, A., Bragg, J., Catullo, R., Coates, D. J., et al. (2017). Diversité génétique et structure de la flore australienne. Plongeur. Distrib. 23, 41–52. doi: 10.1111/ddi.12505
CrossRef Texte intégral /Google Scholar
Byrne, M., Coates, D. J., Forest, F., Hopper, S. D., Krauss, S. L., Sniderman, J. M. K., et coll. (2014). « A diverse flora-species and genetic relationships », dans Plant Life on the Sandplains in Southwest Australia, a Global Biodiversity Hotspot, éditeurs H. Lambers (Crawley, WA: University of Western Australia Publishing), 81-99.
Résumé PubMed
Byrne, M., Macdonald, B., et Coates, D. J. (2002). Modèles phylogéographiques et grande diversité d’adNPC au sein de l’Acacia acuminata (Leguminosae: Mimosoideae). complexe en Australie occidentale. J. Evol. Biol. 15, 576–587. doi: 10.1046 /j.1420-9101.2002.00429.x
CrossRef Texte intégral /Google Scholar
Carstens, C.-B., Pelletier, T.A., Reid, N.M. et Satler, J.D. (2013). Comment échouer à la délimitation des espèces. Mol. Ecol. 22, 4369–4383. doi: 10.1111/mec.12413
Résumé publié par PubMed / Texte intégral croisé / Google Scholar
Coates, D. J. (1988). Genetic diversity and population gene structure in the rare Chittering grass wattle, Acacia anomala Court. Aust. J. Bot. 36:273–286. doi: 10.1071/BT9880273
CrossRef Texte intégral/Google Scholar
Coates, D. J. (2000). Définir des unités de conservation dans une flore riche et fragmentée: implications pour la gestion des ressources génétiques et des processus évolutifs dans les plantes du sud-ouest de l’Australie. Aust. J. Bot. 48, 329–339. doi: 10.1071/BT99018
CrossRef Texte intégral/Google Scholar
Coates, D. J., McArthur, S. L., et Byrne, M. (2015). Perte significative de diversité génétique à la suite de l’extinction de la population causée par les agents pathogènes chez la rare Banksia brownii endémique (Proteaceae). Biol. Conservez. 192, 353–360. doi: 10.1016/j. biocon.2015.10.013
CrossRef Texte intégral /Google Scholar
Colgan, D. J., et Paxton, J.R. (1997). Génétique biochimique et reconnaissance d’un stock occidental de poissons gemmes communs, Rexea solandri (Scombroidea: Gempylidae)., en Australie. Mar. Freshw. Rés. 48, 103 à 118. doi: 10.1071/MF96048
CrossRef Full Text/Google Scholar
Convention sur la diversité biologique (2007). Disponible en ligne à l’adresse suivante : https://www.cbd.int/doc/meetings/cop-bureau/cop-bur-2007/cop-bur-2007-10-14-en.pdf
Cook, C. N., et Sgrò, C. M. (2017). Aligner la science et la politique pour parvenir à une conservation éclairée sur le plan évolutif. Conservez. Biol. 31, 501–512. doi: 10.1111/ cobi.12863
Résumé PubMed | Texte intégral CrossRef / Google Scholar
Cook, L. G., Edwards, R. D., Crisp, MD et Hardy, N.-B. (2010). La morphologie doit-elle toujours être requise pour les descriptions de nouvelles espèces? Inverser. Sys. 24, 322–332. doi: 10.1071/IS10011
CrossRef Texte intégral/Google Scholar
Cracraft, J. (1983). Concepts d’espèces et analyse de spéciation. Curr. Ornithol. 1, 159–187. doi: 10.1007/978-1-4615-6781-3_6
CrossRef Full Text |Google Scholar
Crandall, K. A., Bininda-Emonds, O. R., Mace, G. M. et Wayne, R. K. (2000). Considérant les processus évolutifs en biologie de la conservation. Tendances Ecol. Evol. 15, 290–295. doi: 10.1016/S0169-5347 (00) 01876-0
Résumé PubMed / Texte intégral CrossRef /Google Scholar
Cutter, A.D., et Paysan, B.A. (2013). Signatures génomiques de sélection sur des sites liés : unifier la disparité entre les espèces. NAT. Rév. Genet. 14, 262–274. doi: 10.1038/nrg3425
Résumé PubMed / Texte intégral CrossRef /Google Scholar
Dayrat, B. (2005). Vers une taxonomie intégrative. Biol. J. Linn. Soc. 85, 407–415. doi: 10.1111/j.1095-8312.2005.00503.x5
CrossRef Texte intégral /Google Scholar
de Queiroz, K. (1998). « The general lineage concept of species, species criteria, and the process of speciation « , in Forms: Species and Speciation, D. J. Howard, r S. H. Berlocher (New York, NY: Oxford University Press), 57-75.
Google Scholar
de Queiroz, K. (2007). Concepts d’espèces et délimitation d’espèces. Syst. Biol. 56, 879–886. doi: 10.1080 / 10635150701701083
Résumé PubMed / Texte intégral croisé / Google Scholar
Degnan, J. H., et Rosenberg, N.A. (2009). Discordance de l’arbre génétique, inférence phylogénétique et coalescence multispécifique. Tendances Ecol. Evol. 24, 332–340. doi: 10.1016/j. arbre.2009.01.009
Résumé PubMed | Texte intégral CrossRef /Google Scholar
Devey, D. S., Bateman, R. M., Fay, M. F., et Hawkins, J.A. (2008). Amis ou parents? Phylogénétique et délimitation des espèces dans le genre controversé d’orchidées européennes Ophrys. Ann. Bot. 101, 385–402. doi: 10.1093/aob/mcm299
Résumé publié par PubMed / Texte intégral croisé/ Google Scholar
Dizon, A. E., Lockyer, C, Perrin, W. F., Demaster, D. P. et Sisson, J. (1992). Repenser le concept de stock – une approche phylogéographique. Conservez. Biol. 6, 24–36. doi: 10.1046 /j.1523-1739.1992.610024.x
CrossRef Texte intégral /Google Scholar
Doughty, P., Bourke, G., Tedeschi, L., Pratt, R., Oliver, P., Palmer, R., et al. (2018). Délimitation des espèces dans le Gehyra nana (Squamata: Gekkonidae). complexe : évolution morphologique cryptique et divergente dans les Tropiques de Mousson australiens, avec la description de quatre nouvelles espèces. Zootaxa, 4403: 201-244. doi: 10.11646 / zootaxa.4403.2.1
Résumé PubMed | Texte intégral CrossRef /Google Scholar
Dynesius, M., et Jansson, R. (2014). Persistance des lignées intra-espèces : un contrôle négligé des taux de spéciation. Évolution 68, 923-934. doi: 10.1111/evo.12316
Résumé PubMed / Texte intégral CrossRef /Google Scholar
Edwards, S. V., Potter, S., Schmitt, C. J., Bragg, J. G. et Moritz, C. (2016). Réticulation, divergence et continuum phylogéographie–phylogénétique. Proc. Natl. Acad. Sci. États-Unis 113, 8025-8032. doi: 10.1073/pnas.1601066113
CrossRef Texte intégral /Google Scholar
Faith, D.P. (1992). Conservation evaluation and phylogenetic diversity. Biol. Inconvénient. 61, 1–10. doi: 10.1016/0006-3207(92)91201-3
CrossRef Texte intégral | Google Scholar
Finston, T. L., Johnson, M. S., Humphreys, W. F., Eberhard, S. M. et Halse, S.A. (2007). La spéciation cryptique dans deux genres d’amphipodes souterrains répandus reflète les schémas de drainage historiques dans un paysage ancien. Mol. Ecol. 16, 355–365. doi: 10.1111/j.1365-294X.2006.03123.x
Résumé publié par PubMed / Texte intégral croisé / Google Scholar
Firestone, K. B., Elphinstone, M. S., Sherwin, W. B. et Houlden, B.A. (1999). Structure phylogéographique de la population de quolls tigres Dasyurus maculatus (Dasyuridae: Marsupalia), un marsupial carnivore en voie de disparition. Mol. Ecol. 8, 1613–1625. doi: 10.1046/j.1365-294x.1999.00745.x
CrossRef Texte intégral /Google Scholar
Fišer, C., Robinson, C. T. et Malard, F. (2018). Les espèces cryptiques comme fenêtre sur le changement de paradigme du concept d’espèce. Mol. Ecol. 27, 613–635. doi: 10.1111/mec.14486
Résumé PubMed | Texte intégral CrossRef /Google Scholar
Frankham, R. (2010). Défis et opportunités des approches génétiques de la conservation biologique. Biol. Conservez. 143,1919–1927. doi: 10.1016/j. biocon.2010.05.011
CrossRef Texte intégral /Google Scholar
Frankham, R., Ballou, J. D., Dudash, M. R., Eldridge, M. D., Fenster, C. B., Lacy, R. C., et al. (2012). Implications des différents concepts d’espèces pour la conservation de la biodiversité. Biol. Conservez. 153, 25–31. doi: 10.1016/j. biocon.2012.04.034
CrossRef Texte intégral /Google Scholar
Fraser, D. J., et Bernatchez, L. (2001). Conservation évolutive adaptative: vers un concept unifié pour la définition des unités de conservation. Mol. Ecol. 10, 2741–2752. doi: 10.1046/j.1365-294X.2001.t01-1-01411.x
Résumé publié / Texte intégral croisé / Google Scholar
Funk, W. C., Mckay, J. K., Hohenlohe, P.A., et Allendorf, F.W. (2012). Exploiter la génomique pour délimiter les unités de conservation. Tendances Ecol. Evol. 27, 489–496. doi: 10.1016/j. arbre.2012.05.012
Résumé PubMed | Texte intégral CrossRef /Google Scholar
Garnett, S. T., et Christidis, L. (2017). L’anarchie de la taxonomie entrave la conservation. Nature 546, 25-27. doi: 10.1038/ 546025a
Résumé PubMed / Texte intégral CrossRef /Google Scholar
Gippoliti, S., Cotterill, F. P. D., Zinner, D., et Groves, C. P. (2018). Impacts de l’inertie taxonomique pour la conservation de la diversité des ongulés africains: aperçu. Biol. Rév. 93, 115-130. doi: 10.1111/ brv.12335
Résumé PubMed / Texte intégral croisé / Google Scholar
Grant, V. (1981). Spéciation des plantes. New York, NY: Columbia University Press.
Résumé PubMed /Google Scholar
Hamilton, C. W., et Reichard, S. H. (1992). Pratique actuelle dans l’utilisation de sous-espèces, de variétés et de formes dans la classification des plantes sauvages. Taxon 41, 485-498. doi: 10.2307/1222819
CrossRef Texte intégral/Google Scholar
Harvey, M. S., Rix, M. G., Framenau, V. W., Hamilton, Z. R., Johnson, M. S., Teale, R. J., et coll. (2011). Protéger les innocents: l’étude des taxons endémiques à courte distance améliore les résultats de conservation. Inverser. Syst. 25, 1–10. doi: 10.1071/IS11011
CrossRef Texte intégral/Google Scholar
Hey, J., Waples, R. S., Arnold, M. L., Butlin, R. K. et Harrison, R. G. (2003). Comprendre et affronter l’incertitude des espèces en biologie et en conservation. Tendances Ecol. Evol. 18, 597–603. doi: 10.1016/j. arbre.2003.08.014
CrossRef Texte intégral /Google Scholar
Hoban, S. M., Hauffe, H. C., Perez-Espona, S., Arntzen, J. W., Bertorelle, G., Bryja, J., et al. (2013). Mettre la diversité génétique au premier plan de la politique et de la gestion de la conservation. Conservez. Genet. Ressources. 5, 593–598. doi: 10.1007/s12686-013-9859- y
CrossRef Texte intégral /Google Scholar
Isaac, N. J., Mallet, J. et Mace, G. M. (2004). Inflation taxonomique: son influence sur la macroécologie et la conservation. Tendances Ecol. Evol. 19, 464–469. doi: 10.1016/j. arbre.2004.06.004
Résumé PubMed | Texte intégral croisé / Google Scholar
Isaac, N. J., Turvey, S. T., Collen, B., Waterman, C. et Baillie, J. E. (2007). Mammifères à la LISIÈRE : priorités de conservation basées sur la menace et la phylogénie. PLoS ONE 2: e296. doi: 10.1371 / journal.pone.0000296
Résumé PubMed | Texte intégral CrossRef /Google Scholar
Jones, M. R., et Good, J. M. (2016). Capture ciblée en génomique évolutive et écologique. Mol. Ecol. 25, 185–202. doi: 10.1111/mec.13304
Résumé PubMed | Texte intégral croisé / Google Scholar
Jorger, K. M., et Schrödl, M. (2013). Comment décrire une espèce cryptique? Défis pratiques de la taxonomie moléculaire. Devant. Zou. 10:59. doi: 10.1186/1742-9994-10-59
Résumé PubMed | Texte intégral CrossRef / Google Scholar
Laikre, L., Allendorf, F., Aroner, L. C., Baker, S., Gregovich, D. P., Hansen, M. M., et al. (2010). Négligence de la diversité génétique dans la mise en œuvre de la convention sur la diversité biologique. Conservez. Biol. 24, 86–88. doi: 10.1111/j.1523-1739.2009.01425.x
Résumé PubMed / Texte intégral CrossRef /Google Scholar
Laikre, L., Palm, S. et Ryman, N. (2005). Structure génétique des populations de poissons : implications pour la gestion des zones côtières. Ambio 34,111-119. doi: 10.1579/0044-7447-34.2.111
Résumé PubMed | Texte intégral CrossRef / Google Scholar
Laïcs, T., Laffan, S. W., González-Orozco, C. E., Faith, D. P., Rosauer, D. F., Byrne, M., et al. (2015). La phylodiversité pour éclairer la politique de conservation: Un exemple australien. Sci. Total Environ. 534, 131–143. doi: 10.1016 / j. scitotenv.2015.04.113
Résumé PubMed / Texte intégral CrossRef /Google Scholar
Leache, A. D., et Oaks, J. R. (2017). L’utilité du polymorphisme nucléotidique unique (SNP). données en phylogénétique. Ann. Rév. Ecol. Syst. 48, 69–84. doi: 10.1146/annurev-ecolsys-110316-022645
CrossRef Full Text | Google Scholar
Llorens, T. M., Macdonald, B., McArthur, S., Coates, D. J., and Byrne, M. (2015). Disjunct, highly divergent genetic lineages within two rare Eremophila (Scrophulariaceae: Myoporeae). species in a biodiversity hotspot: implications for taxonomy and conservation. Biol. J. Linn. Soc. 177, 96–111. doi: 10.1111/boj.12228
CrossRef Full Text | Google Scholar
Love Stowell, S. M., Pinzone, C. A., and Martin, A. P. (2017). Surmonter les obstacles aux interventions actives pour la diversité génétique. Biodiversités. Conservez. 26, 1753–1765. doi: 10.1007/s10531-017-1330- z
CrossRef Texte intégral /Google Scholar
Mace, G. M. (2004). Le rôle de la taxonomie dans la conservation des espèces. Philo. Trans. R. Soc. D : Biol. Sci. 359, 711–719. doi: 10.1098/rstb.2003.1454
Résumé PubMed | Texte intégral CrossRef /Google Scholar
Mallet, J. (2007). Spéciation hybride. Nature 446, 279-283. doi: 10.1038/nature05706
Résumé publié | Texte intégral croisé / Google Scholar
Mayr, E. (1942). Systématique et Origine des espèces. New York, NY: Columbia University Press.
Google Scholar
Mee, J. A., Bernatchez, L., Reist, J. D., Rogers, S. M. et Taylor, E. B. (2015). Identification des unités désignables pour l’établissement de priorités de conservation intraspécifiques : une approche hiérarchique appliquée au complexe d’espèces de corégone de lac (Coregonus spp.). Evol. Appl. 8, 423–441. doi: 10.1111 / eva.12247
PubMed Abstract | CrossRef Full Text | Google Scholar
Millar, M. A., and Byrne, M. (2013). Cryptic divergent lineages of Pultenaea pauciflora M.B. Scott (Fabaceae: Mirbelieae) exhibit different evolutionary history. Biol. J. Linn. Soc. 108, 871–881. doi: 10.1111/bij.12002
CrossRef Full Text | Google Scholar
Moritz, C. (1994). Defining evolutionarily significant units for conservation. Trends Ecol. Evol. 9, 373–375. doi: 10.1016/0169-5347(94)90057-4
CrossRef Full Text | PubMed Abstract
Moritz, C. (1999). Unités de conservation et translocations: stratégies de conservation des processus évolutifs. Hérédité 130, 217-228. doi: 10.1111/j.1601-5223.1999.00217.x
CrossRef Texte intégral /Google Scholar
Moritz, C. (2002). Stratégies de protection de la diversité biologique et des processus évolutifs qui la soutiennent. Syst. Biol. 51, 238–254. doi: 10.1080 | 10635150252899752
Résumé PubMed / Texte intégral CrossRef /Google Scholar
Moritz, C., et Potter, S. (2013). L’importance d’une perspective évolutive dans la planification des politiques de conservation. Mol. Ecol. 22, 5969–5971. doi: 10.1111/mec.12565
Résumé publié / Texte intégral croisé / Google Scholar
Moritz, C., Pratt, R. C., Bank, S., Bourke, G., Bragg, J. G., Doughty, P., et al. (2018). Diversité cryptique des lignées, divergence de taille corporelle et sympatrie dans un complexe d’espèces de lézards australiens (Gehyra). Évolution 27, 54-66. doi: 10.1111/evo.13380
CrossRef Texte intégral / Google Scholar
Morrison, W. R., Lohr, J. L., Duchen, P., Wilches, R., Trujillo, D., Mair, M., et al. (2009). L’impact du changement taxonomique sur la conservation: Est-ce que ça tue, peut-il sauver, ou est-ce juste hors de propos? Biol. Inconvénient. 142, 3201–3206. doi: 10.1016/j. biocon.2009.07.019
CrossRef Texte intégral /Google Scholar
Murphy, N. P., Adams, M., Guzik, M. T. et Austin, A.D. (2013). Micro-endémisme extraordinaire chez les amphipodes de printemps du désert australien. Mol. Phylogenet. Evol. 66, 645–653. doi: 10.1016/ j. ympev.2012.10.013
Résumé PubMed | Texte intégral CrossRef /Google Scholar
Murphy, N. P., King, R.A., et Delean, S. (2015). Espèces, USE ou populations ? Délimitation et description de la diversité morphologiquement cryptique chez les amphipodes printaniers du désert australien. Inverser. Syst. 29, 457–467. doi: 10.1071/IS14036
CrossRef Texte intégral/Google Scholar
Nater, A., Mattle-Greminger, M. P., Nurcahyo, A., Nowak, M. G., de Manual, M., Desai, T., et al. (2017). Données morphométriques, comportementales et génomiques pour une nouvelle espèce d’orang-outan. Curr. Biol cellulaire. 27, 3487–3498. doi: 10.1016 / j. cub.2017.09.047
CrossRef Texte intégral / Google Scholar
Nicotra, A. B., Chong, C., Bragg, J. G., Ong, C. R., Aitken, N. C., Chuah, A., et coll. (2016). Population and phylogenomic decomposition via genotyping-by-sequencing in Australian Pelargonium. Mol. Ecol. 25, 2000–2014. doi: 10.1111/mec.13584
Résumé PubMed / Texte intégral croisé / Google Scholar
Nistelberger, H., Byrne, M., Coates, D. et Roberts, J. D. (2014a). Une forte structure phylogéographique chez un mille-pattes indique une vicariance du Pléistocène entre les populations des formations de fer rubanées en Australie semi-aride. PLoS ONE 9: e0093038. doi: 10.1371 / journal.pone.0093038
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Nistelberger, H., Gibson, N., Macdonald, B., Tapper, S.-L. et Byrne, M. (2014b). Preuve phylogéographique de deux refuges mésiques dans un point chaud de biodiversité. Hérédité 113, 454-463. doi: 10.1038/hdy.2014.46
Résumé PubMed | Texte intégral CrossRef / Google Scholar
Ogilvie, H. A., Heled, J., Xie, D. et Drummond, A. J. (2016). Performances de calcul et précision statistique de beast et comparaisons avec d’autres méthodes. Syst. Biol. 65, 381–396. doi: 10.1093/sysbio/syv118
Résumé publié par PubMed | Texte intégral croisé / Google Scholar
Oliver, P. M., Keogh, J. S. et Moritx, C. (2015). New approaches to cataloguing and understanding evolutionary diversity: a perspective from Australian herpetology. Aust. J. Zool. 62, 417–430. doi: 10.1071/ZO14091
CrossRef Texte intégral/Google Scholar
Oliver, P. M., Laver, R. J., Demello Martins, F., Pratt, R. C., Hunjan, S. et Moritz, C. (2017). Un nouveau point névralgique de l’endémisme des vertébrés et un refuge évolutif en Australie tropicale. Plongeur. Distrib. 23, 53–66. doi: 10.1111/ddi.12506
CrossRef Texte intégral /Google Scholar
Oliver, P. M., et Lee, M. S. Y. (2010). Les codes botaniques et zoologiques entravent la recherche sur la biodiversité en décourageant la publication de nouvelles espèces non nommées. Taxon 59, 1201-1205. doi: 10.2307/ 20773990
CrossRef Texte intégral/Google Scholar
Padial, J., Miralles, A., De la Riva, I., et Vences, M. (2010). L’avenir intégratif de la taxonomie. Devant. Zou. 7:16. doi: 10.1186/1742-9994-7-16
Résumé publié par PubMed | Texte intégral croisé / Google Scholar
Palsbøll, P. J., Bérubé, M. et Allendorf, F. W. (2007). Identification des unités de gestion à l’aide de données génétiques de population. Tendances Ecol. Evol. 22, 11–16. doi: 10.1016/j. arbre.2006.09.003
Résumé publié par PubMed | Texte intégral croisé / Google Scholar
Patton, J. L., et Smith, M. F. (1994). La paraphylie, la polyphylie et la nature des limites des espèces chez les Gophers de poche (genre Thomomys). Syst. Biol. 43, 11–26. doi: 10.1093/sysbio/43.1.11
CrossRef Texte intégral / Google Scholar
Pierson, J.C., Coates, D. J., Oostermeijer, J. G. B., Beissinger, S. R., Bragg, J. G., Sunnucks, P., et al. (2016). Genetic factors in threatened species recovery plans on three continents. Devant. Ecol. Environ. 14, 433–440. doi: 10.1002 / frais.1323
CrossRef Texte intégral / Google Scholar
Potter, S., Bragg, J. G., Peter, B. M., Bi, K., et Moritz, C. (2016). La phylogénomique aux conseils: inférer les lignées et leur histoire démographique chez un lézard tropical, Carlia amax. Mol. Ecol. 25, 1367–1380. doi: 10.1111/mec.13546
Résumé PubMed | Texte intégral CrossRef / Google Scholar
Potter, S., Xue, A. T., Bragg, J. G., Rosauer, D. F., Roycroft, E. J. et Moritz, C. (2018). Les changements climatiques du Pléistocène favorisent la diversification dans une savane tropicale. Mol. Ecol. 27, 520–532. doi: 10.1111/mec.14441/complet
Résumé publié par PubMed / Texte intégral croisé / Google Scholar
Rabosky, D. L., Hutchinson, M. N., Donnellan, S. C., Talaba, A. L. et Lovette, I. J. (2014). Désassemblage phylogénétique des limites des espèces dans un groupe répandu de scinques australiens (Scincidae: Ctenotus). Mol. Phylogenet. Evol. 77, 71–82. doi: 10.1016/ j. ympev.2014.03.026
Résumé PubMed | CrossRef Texte intégral / Google Scholar
Ralls, K., Ballou, J. D., Dudash, M. R., Eldridge, M. D. B., Fenster, C. B., Lacy, R. C., et al. (2018). Appel à un changement de paradigme dans la gestion génétique des populations fragmentées. Conservez. Lett. 11, 1–6. doi: 10.1111/conl.124126
CrossRef Texte intégral /Google Scholar
Rannala, B. (2015). L’art et la science de la délimitation des espèces. Curr. Zou. 61, 846–853. doi: 10.1093/czoolo/61.5.846
CrossRef Texte Intégral | Google Scholar
Riedel, A., Sagata, K., Suhardjono, Y. R., Tänzler, R., et Balke, M. (2013). Taxonomie intégrative sur la voie rapide – vers plus de durabilité dans la recherche sur la biodiversité. Devant. Zou. 10:15. doi: 10.1186/1742-9994-10-15
Résumé PubMed | Texte intégral croisé /Google Scholar
Rieseberg, L. H., et Willis, J. H. (2007). Spéciation des plantes. Science 317, 910-914. doi: 10.1126 / science.1137729
Résumé publié | Texte intégral croisé / Google Scholar
Rosauer, D. F., Blom, M. P. K., Bourke, G., Catalano, S., Donnellan, S., Mulder, E., et al. (2016). Phylogéographie, points chauds et priorités de conservation: un exemple de l’extrémité supérieure de l’Australie. Biol. Inconvénient. 204, 83–93. doi: 10.1016/j. biocon.2016.002
CrossRef Texte intégral / Google Scholar
Rosauer, D. F., Byrne, M., Blom, M. P. K., Coates, D. J., Donnellan, S., Doughty, P., et al. (2018). La planification de la conservation dans le monde réel pour la diversité évolutive dans le Kimberley, en Australie, évite les conservations incertaines de la taxonomie. Lett. 11:e12438. doi: 10.1111/conl.12438
CrossRef Texte intégral / Google Scholar
Rosenblum, E. B., Sarver, B. A. J., Brown, J. W., Des Roches, S., Hardwick, K. M., Hether, T. D., et al. (2012). Boucle d’Or rencontre Santa Rosalia: un modèle de spéciation éphémère explique les modèles de diversification à travers les échelles de temps. Evol. Biol. 39, 255–261. doi: 10.1007 / l11692-012-9171- x
Résumé PubMed | CrossRef Texte intégral |Google Scholar
Roux, C., Fraïsse, C., Romiguier, J., Anciens, Y., Galtier, N., et Bierne, N. (2016). Jeter la lumière sur la zone grise de spéciation le long d’un continuum de divergence génomique. PLoS Biol. 14: e2000234. doi: 10.1371 / journal.pbio.2000234
Résumé PubMed / Texte intégral CrossRef /Google Scholar
Ryder, O.A. (1986). Conservation des espèces et systématique – le dilemme des sous-espèces. Tendances Ecol. Evol. 1, 9–10. doi: 10.1016/0169-5347 (86) 90059-5
CrossRef Texte intégral |Google Scholar
Sampson, J. F., Byrne, M., Yates, C. J., Gibson, N., Thavornkanlapachai, R., Stankowski, S., et al. (2014). L’immigration génétique contemporaine médiée par le pollen reflète l’isolement historique d’un arbuste rare dans un paysage fragmenté. Hérédité 112, 172-181. doi: 10.1038/hdy.2013.89
Résumé PubMed | Texte intégral CrossRef /Google Scholar
Santamaría, L., et Méndez, P. F. (2012). Évolution de la politique de biodiversité : lacunes actuelles et besoins futurs. Evol. Appl. 5, 202–218. doi: 10.1111 / j.1752-4571.2011.00229.x
Résumé PubMed / CrossRef Texte intégral /Google Scholar
Schlick-Steiner, B. C., Steiner, F. M., Seifert, B., Stauffer, C., Christian, E. et Crozier, R. H. (2010). Taxonomie intégrative: une approche multisource pour explorer la biodiversité. Annu. Rév. Entomol. 55, 421–438. doi: 10.1146/annurev-ento-112408-085432
Résumé PubMed / Texte intégral croisé /Google Scholar
Seehausen, O, Takimoto, G., Roy, D. et Jokela, J. (2008). Inversion de spéciation et dynamique de la biodiversité avec hybridation dans des environnements changeants. Mol Ecol. 17, 30–44. doi: 10.1111/j.1365-294X.2007.03529.x
Résumé PubMed / Texte intégral CrossRef /Google Scholar
Shepherd, K. A., Thiele, K. R., Sampson, J., Coates, D. J. et Byrne, M. (2015). Une espèce rare et nouvelle d’Atriplex (Chenopodiaceae) comprenant deux populations génétiquement distinctes mais morphologiquement cryptiques en Australie occidentale aride: implications pour la taxonomie et la conservation. Aust. Syst. Bot. 28, 234–245. doi: 10.1071/SB15029
CrossRef Texte intégral /Google Scholar
Singhal, S., et Bi, K. (2017). L’histoire nettoie les dégâts: L’impact du temps sur la divergence et l’introgression dans une zone de suture tropicale. Évolution 71, 1888-1899. doi: 10.1111/evo.13278
Résumé PubMed | Texte intégral croisé / Google Scholar
Singhal, S., Hoskin, C., Couper, P., Potter, S. et Moritz, C. (2018). Cadre pour la résolution des espèces cryptiques: étude de cas des lézards des tropiques humides australiens. Syst. Biol. . doi: 10.1093/sysbio/syy
Résumé publié par PubMed | Texte intégral croisé / Google Scholar
Singhal, S., et Moritz, C. (2013). Isolement reproductif entre les lignées phylogéographiques échelles avec divergence. Proc. Roy. Soc B 280: 20132246. doi: 10.1098/ rspb.2013.2246026
Résumé PubMed | Texte intégral croisé / Google Scholar
Steane, D.A., Potts, Bm, McLean, E., Collins, L., Prober, S. M., Stock, W. D., et al. (2015). Les analyses à l’échelle du génome révèlent une structure de population cryptique dans un eucalyptus adapté à la sécheresse. Genet d’arbre. Génomes 11:33. doi: 10.1007 / l11295-015-0864- z
CrossRef Texte intégral / Google Scholar
Struck, T. H., Feder, J. L., Bendiksby, M., Birkeland, S., Cerca, J., Gusarov, V. I., et al. (2017). Trouver des processus évolutifs cachés dans des espèces cryptiques. Tendances Ecol. Evol. 33, 153–163. doi: 10.1016/j. arbre.2017.11.007
Résumé PubMed | Texte intégral CrossRef /Google Scholar
Sukumaran, J., et Knowles, L. L. (2017). La coalescence multispécifique délimite la structure et non les espèces. Proc. NAT. Aca. Sci. États-Unis 114, 1607-1612. doi: 10.1073/pnas.1607921114
Résumé PubMed | Texte intégral CrossRef / Google Scholar
Swarts, N.d., Clements, M.A., Bower, C.C. et Miller, J.T. (2014). Définir des unités de conservation dans un complexe d’orchidées morphologiquement similaires, sexuellement trompeuses et hautement menacées. Biol. Inconvénient. 174, 55–64. doi: 10.1016/j. biocon.2014.03.017
CrossRef Texte intégral /Google Scholar
Tautz, D., Arctander, P., Minelli, A., Thomas, R. H., et Vogler, A. P. (2003). Un plaidoyer pour la taxonomie de l’ADN. Tendances Ecol. Evol. 18, 70–74. doi: 10.1016/S0169-5347 (02) 00041-1
CrossRef Texte intégral/Google Scholar
Taylor, H. R., Dussex, N., et van Heezik, Y. (2017). Combler le fossé génétique de la conservation en identifiant les obstacles à la mise en œuvre pour les praticiens de la conservation. Glob. Ecol. Conservez. 10, 231–242. doi: 10.1016 / j. gecco.2017.04.001
CrossRef Texte intégral / Google Scholar
Thomson, S.a., Pyle, R. L., Ahyong, S. T., Alonso-Zarazaga, M., Ammirati, J., Araya, J. F., et al. (2018). La taxonomie basée sur la science est nécessaire à la conservation mondiale. PLoS Biol. 16: e2005075. doi: 10.1371 / journal.pbio.2005075
Résumé PubMed / CrossRef Texte intégral / Google Scholar
Vieites, D. R., Wollenberg, K. C., Andreonec, F., Köhler, J., Glaw, F., et Vences, M. (2009). Vaste sous-estimation de la biodiversité de Madagascar mise en évidence par un inventaire intégratif des amphibiens. Proc. Natl. Acad. Sci. États-Unis 106, 8267-8272. doi: 10.1073/pnas.0810821106
CrossRef Texte intégral
Vogler, A. P., et de Salle, R. (1994). Diagnostic des Unités de Gestion de la Conservation. Inconvénient. Biol. 8, 354–363. doi: 10.1046 /j.1523-1739.1994.08020354.x
CrossRef Texte intégral /Google Scholar
Waples, R.S. (1991). Saumon du Pacifique, Oncorhynchus spp., et la définition d' » espèce ” en vertu de la Loi sur les espèces en voie de disparition. Mar. Poisson. Rév. 53, 11-22.
Google Scholar
Waples, R.S. (1995). « Evolutionarily significant units and the conservation of biological diversity under the Endangered Species Act « , in Evolution and the Aquatic Ecosystem: Defining Unique Units in Population Conservation, éd. J. L. Nielsen (Bethesda, MD: American Fisheries Society), 8-27.
Google Scholar
Waples, R. S., Gustafson, R. G., Weitkamp, L.A., Myers, J. M., Jjonson, O. W., Busby, P. J., et al. (2001). Characterizing diversity in salmon from the Pacific Northwest. J. Poisson Biol. 59, 1–41. doi: 10.1111/j.1095-8649.2001.tb01376.x
CrossRef Texte intégral /Google Scholar
Waples, R. S., Nammack, M., Cochrane, J. F., et Hutchings, J.A. (2013). Un récit de deux actes : les pratiques d’inscription des espèces en voie de disparition au Canada et aux États-Unis. BioScience 63, 723-734. doi: 10.1093/bioscience/63.9.723
CrossRef Texte intégral /Google Scholar
Waples, R. S., Punt, A. E., et Cope, J. M. (2008). Intégrer les données génétiques dans la gestion des ressources marines : comment faire mieux ? Poisson 9, 423-449. doi: 10.1111/j.1467-2979.2008.00303.x
CrossRef Texte intégral /Google Scholar
Wege, J. A., Thiele, K. R., Shepherd, K. A., Butcher, R., Macfarlane, T. D., et Coates, D. J. (2015). Taxonomie stratégique dans un paysage riche en biodiversité: une nouvelle approche pour maximiser les résultats de conservation de la flore rare et mal connue. Biodiversités. Conservez. 24, 17–32. doi: 10.1007/s10531-014-0785-4
CrossRef Texte intégral /Google Scholar
Whitlock, R. (2014). Relations entre la diversité génétique adaptative et neutre et la structure et le fonctionnement écologiques: une méta-analyse. J. Ecol. 102, 857–872. doi: 10.1111/1365-2745.12240
Résumé publié/ Texte intégral croisé / Google Scholar
Wiley, E.O. (1978). Le concept d’espèce évolutive reconsidéré. Syst. Biol. 27, 17–26. doi: 10.2307/2412809
CrossRef Texte intégral/Google Scholar
Wilson, E.O. (2017). La recherche sur la biodiversité nécessite plus de bottes sur le terrain. NAT. Ecol. Evol. 1, 1590–1591. doi: 10.1038/s41559-017-0360- y
Résumé publié par PubMed / Texte intégral croisé / Google Scholar
Yang, Z. H., et Rannala, B. (2014). Délimitation non guidée des espèces à l’aide de données de séquences d’ADN provenant de locus multiples. Mol. Biol. Evol. 31, 3125–3135. il s’agit de la première édition de la série de jeux vidéo de l’Université de Californie à Los Angeles en 2011, et de la première édition de la série de jeux vidéo de l’Université de Californie à Los Angeles en 2011. Taxonomie intégrative ou taxonomie itérative ? Sys. Ent. 36, 209–217. doi: 10.1111/j.1365-3113.2010.00558.x
CrossRef Full Text | Google Scholar