A principios de la década de 1960, los biólogos comenzaron a investigar en diferentes especies evolucionan a nivel de secuencia (Zuckerkandl 1962, Margoliash 1963, Doolittle& Blombäck 1964). Las proteínas analizadas incluyeron hemoglobina (Zuckerkandl & Pauling 1962), citocromo C (Margoliash 1963) y fibrinopéptidos (Doolittle & Blombäck 1964). Estas primeras investigaciones llevaron a un descubrimiento notable: parecía que el número de diferencias entre secuencias de proteínas de diferentes especies era aproximadamente proporcional al tiempo transcurrido desde la divergencia de especies (Figura 1).
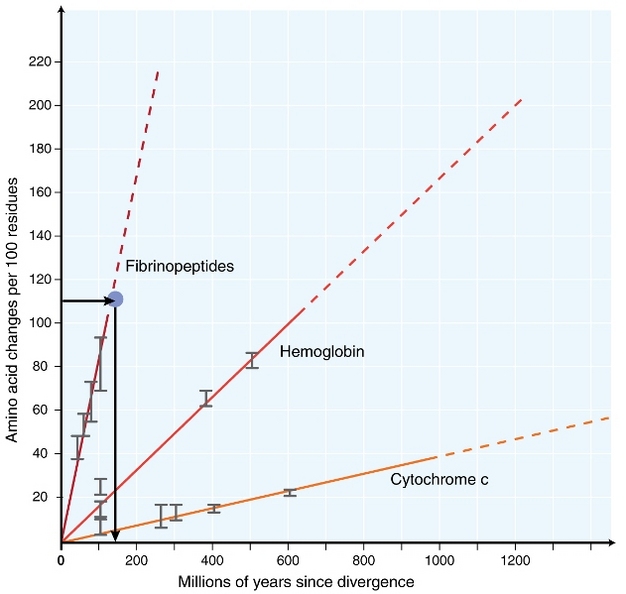
Zuckerkandl & Pauling (1965) comparó la acumulación constante de sustituciones de aminoácidos a lo largo del tiempo con las «marcas» regulares de relojes, y afirmó que «puede existir un reloj evolutivo molecular». Por lo tanto, el término’ reloj molecular ‘ fue acuñado inicialmente para describir los cambios en los aminoácidos que ocurren en proporción al tiempo desde la divergencia de las especies.
Desde su primer uso ,el término ‘reloj molecular’ se ha utilizado en muchos contextos diferentes. Hoy en día, a menudo se usa para referirse simplemente al número de cambios, o ‘sustituciones’, acumulados en la secuencia de ADN o proteínas en un linaje dado. El número de sustituciones por una unidad de tiempo definida puede describirse como la «tasa» del reloj molecular, que en este contexto es equivalente a la «tasa evolutiva». Tenga en cuenta que la connotación inicial todavía se mantiene en algunos casos. Por ejemplo, cuando la gente dice «asumimos un reloj molecular» (particularmente en análisis filogenéticos), significa que se suponía que el número de sustituciones ocurría constantemente a lo largo del tiempo.
El concepto de un reloj molecular constante tiene implicaciones extraordinarias para los biólogos evolutivos. Si realmente existiera un reloj molecular constante como se propuso inicialmente, inferir el tiempo de los eventos evolutivos se convertiría en un problema bastante sencillo (Figura 1). Sin embargo, ha quedado muy claro que las sustituciones no ocurren constantemente a lo largo del tiempo en diferentes linajes (Kumar 2005). Sin embargo, el concepto de reloj molecular ha sido extremadamente influyente en el campo de la evolución molecular. Una de las ideas más importantes inspiradas en el concepto del reloj molecular es la teoría neutra de la evolución molecular.
El Reloj molecular y la Neutralidad
Cuando surgió por primera vez la idea de un reloj molecular constante, se pensó que la fuerza evolutiva predominante subyacente a las sustituciones de aminoácidos o nucleótidos era la selección natural. Siguiendo esta línea de pensamiento, un reloj molecular constante indicaría que las sustituciones adaptativas en diferentes especies ocurren constantemente a lo largo del tiempo. Sin embargo, es difícil explicar cómo sucederían las sustituciones adaptativas de una manera similar a un reloj. Teóricamente, el destino de las mutaciones adaptativas está determinado por varios parámetros evolutivos, como la fuerza de la ventaja selectiva de esa mutación, el tamaño de la población efectiva y las tasas de mutación adaptativa (Kimura 1983). Es probable que estos parámetros difieran entre especies, e incluso dentro de una especie, dependiendo de mutaciones específicas y sus interacciones con el medio ambiente.
En cambio, Kimura (1968, 1969) propuso que la mayoría de los cambios a nivel molecular tienen pocas consecuencias funcionales, o son «neutros». Si una mutación no tiene consecuencias de aptitud, su destino en la población se determina completamente por casualidad. Esto significa que no podemos predecir si una mutación neutra específica eventualmente se fijará en la población. Sin embargo, la velocidad a la que ocurren sustituciones neutras en la población se puede predecir porque depende de un solo parámetro, a saber, la tasa de mutación (Kimura 1968).
imaginemos una población con N número de individuos haploides. Si las mutaciones neutras ocurren a una velocidad de u por individuo por generación, el número total de mutaciones en una generación será de N por u. Dado que todas estas nuevas mutaciones son neutras, su destino está completamente determinado por el azar. En otras palabras, todas las mutaciones tienen las mismas posibilidades de alcanzar la fijación (lo que conduce a una «sustitución»). La probabilidad de que cada nueva mutación neutra alcance la fijación, dado que se produjo una sustitución, es simplemente 1 / N. La tasa de sustituciones se calcula como el número de nuevas mutaciones en cada generación (Nu) multiplicado por la probabilidad de que cada nueva mutación alcance la fijación (1/N), lo que equivale a u. En otras palabras, para mutaciones neutras, la tasa de sustitución es igual a la tasa de mutación.
Por lo tanto, si la mayoría de las mutaciones son neutrales (como se propone en la teoría neutral) y si las tasas de mutación son constantes a lo largo del tiempo, las sustituciones también deben ocurrir constantemente a lo largo del tiempo. Entonces deberíamos observar tasas regulares de sustituciones similares a un reloj a nivel molecular. Kimura (1969) consideró la observación de un reloj molecular relativamente constante en secuencias de proteínas como un fuerte apoyo para la teoría neutra de la evolución molecular.
Probando Cómo funciona el Reloj Molecular Neutro
De acuerdo con la teoría neutral, la pregunta de si las tasas de sustitución son constantes en el tiempo o no es equivalente a si las tasas de mutación neutras son constantes en el tiempo. Por esta razón, muchos estudios posteriores se centraron en analizar datos de sitios neutros para determinar si las tasas de mutación neutras son constantes a lo largo del tiempo. Revisaremos brevemente cómo se implementan estos estudios, antes de discutir los debates teóricos sobre los relojes moleculares. Los análisis de relojes moleculares de proteínas también continuaron, pero los debates en torno a la variación en los relojes moleculares de proteínas son muy diferentes de los relativos a los relojes moleculares neutrales, y no se incluirán en este artículo. Los lectores interesados deben consultar Gillespie (1991), Kumar (2005), Kim y Yi (2008), y Bedford et al. (2008).
La mayoría de los análisis empíricos de relojes moleculares neutros se basan en el teorema de que las tasas de mutación neutras pueden inferirse de las tasas de sustitución neutras (Kimura 1968, 1969). En la práctica, cada estudio define un cierto tipo de sitios en el genoma como sitios neutrales, y compara las tasas de sustitución de esos sitios entre linajes.
No se puede determinar por completo qué sitios en el genoma son verdaderamente neutros, pero los científicos crearon varios proxies útiles. Antes de la era de la secuenciación del genoma, la mayoría de los datos de secuencia disponibles eran los de secuencias de ADN codificantes de proteínas. Los estudios a menudo dividen las secuencias de ADN codificantes de proteínas en dos tipos de sitios (Wu & Li 1985). El primer tipo de sitios incluye aquellos para los que cualquier cambio daría lugar a sustituciones de aminoácidos, o «sitios no sinónimos». El segundo tipo de sitios, «sitios sinónimos», incluye aquellos que codifican posiciones «degeneradas» en la tabla de codones, donde un cambio no conduce a una sustitución de aminoácidos. Por ejemplo, TCT y TCC codifican serina. Si la tercera posición de estos codones cambia, seguirán produciendo los mismos aminoácidos. Tales sustituciones serían menos visibles para la selección natural. En consecuencia, los relojes moleculares en sitios sinónimos deben estar más cerca del reloj molecular neutro que los relojes no sinónimos.
A medida que avanzaban las técnicas de secuenciación, algunos estudios utilizaron intrones como proxies para sitios neutros, ya que no se incorporan a los ARNm maduros y, por lo tanto, es más probable que sean neutros (Yi et al. 2002). Secuencias de elementos transponibles inactivos que se insertaron mucho antes de que la divergencia de especies también se empleara a menudo (a menudo se les conoce como ‘repeticiones ancestrales’, por ejemplo, Thomas et al., 2003). Finalmente, algunos estudios utilizaron secuencias de ADN no codificantes (todas las secuencias después de eliminar secuencias de ADN codificantes de proteínas) extraídas de alineaciones de genoma completo para probar relojes moleculares neutros (Elango et al. 2006, Huttley et al. 2007).
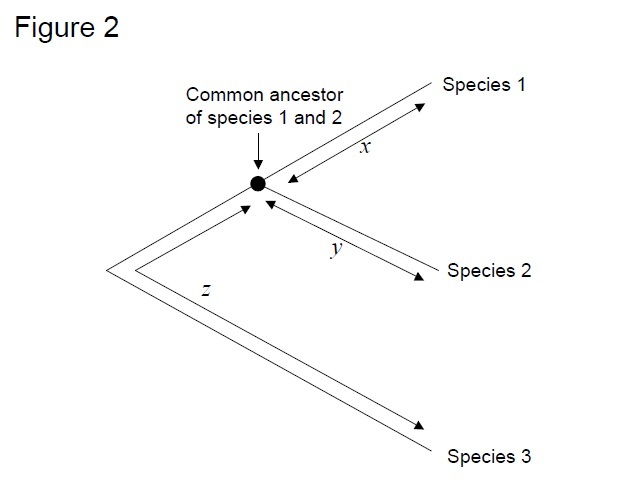
La prueba más utilizada es la llamada «prueba de tasa relativa»(Sarich & Wilson 1973). Inicialmente, las tasas de sustitución por unidad de tiempo se estimaron dividiendo el número total de diferencias (sustituciones) entre proteínas de diferentes especies por el tiempo de divergencia, estimado a partir de registros fósiles (Figura 1). Sin embargo, los registros fósiles no están disponibles para muchas comparaciones, y están asociados con grandes márgenes de error. La prueba de tasa relativa supera la necesidad de registros fósiles (Figura 2). Mientras exista una secuencia de grupos externos a los dos linajes de interés, podemos determinar si las dos ramas siguen los mismos o diferentes relojes moleculares, sin el conocimiento del tiempo absoluto de divergencia (Figura 2).
¿cuáles son los Determinantes de la Neutral Relojes Moleculares?
Casi todas las controversias en el corazón de los debates sobre los relojes moleculares neutrales se derivan de la cuestión de cuáles son las principales fuentes de mutaciones. Esta pregunta es directamente relevante para comprender los patrones de mutación, que son la fuente última del cambio evolutivo y de la enfermedad genética. Además, comprender cómo varían las tasas de mutación entre los linajes y dentro de los genomas es una cuestión fundamental en la genómica comparativa, que tiene como objetivo usar comparaciones de secuencias para identificar regiones genómicas que son funcionalmente importantes.
Entonces, ¿qué determina las tasas de mutación neutras? Uno de los contribuyentes más importantes a los relojes moleculares neutrales es la variación específica del linaje en los tiempos de generación. Desde el principio, la idea de un reloj molecular neutral constante se percibió como incompatible con los mecanismos moleculares de la mutación de la línea germinal. Durante mucho tiempo se ha considerado que la mayoría de las mutaciones surgen de errores en la replicación del ADN en las líneas germinales (Haldane 1947, Muller 1954). Dado que las mutaciones ocurren cuando el ADN de la línea germinal se replica para la próxima generación, deben acumularse en proporción al número de generaciones, en lugar de la cantidad absoluta de tiempo. Por lo tanto, si comparamos el número de sustituciones que se han acumulado en dos linajes desde su divergencia, el linaje con un tiempo de generación más largo, habiendo sufrido menos eventos de replicación de ADN, albergaría menos sustituciones en comparación con el linaje con un tiempo de generación más corto. En consecuencia, el reloj molecular debería correr más lentamente en especies con tiempos de generación más largos. Esta idea se conoce como el «efecto de tiempo de generación».
De hecho, el efecto del tiempo de generación se observó por primera vez en estudios de primates, incluso antes del debate sobre el reloj molecular. Morris Goodman, que estaba usando métodos inmunológicos para investigar la relación de especies en ese momento, observó que la velocidad a la que algunas proteínas divergen parecía disminuir en los simios, en particular en los humanos, en comparación con los monos del Viejo Mundo (Goodman 1961, 1962, 1963). Este efecto se conoce como «desaceleración de la tasa hominoide». Dado que los hominoides tienen tiempos de generación más largos que los monos del Viejo Mundo, esta observación puede explicarse por el efecto de tiempo de generación.
Wu& Li (1985) fueron los primeros en probar la hipótesis del efecto en tiempo de generación utilizando datos de secuencias de ADN. Utilizaron datos de 11 genes de primates y roedores. Dado que los primates tienen un tiempo de generación mucho más largo que los roedores, el reloj molecular debería ser más rápido en los roedores en comparación con los primates. De hecho, encontraron que para sitios sinónimos, los roedores muestran aproximadamente dos veces la tasa de evolución molecular en comparación con los primates (Wu & Li 1985). Sin embargo, en el caso de sitios no sinónimos, no se encontró tal efecto. En otras palabras, el reloj molecular neutro, pero no el reloj molecular de aminoácidos, marca más rápido en el linaje de roedores en comparación con el linaje de primates, lo que encaja bien con la idea de un efecto de tiempo de generación.
Los estudios posteriores proporcionaron más apoyo a la desaceleración de la tasa de hominoides (Li & Tanimura 1987, Bailey et al. 1991)y la diferencia de velocidad entre los linajes de roedores y primates (Gu & Li 1992, Huttley et al. 2007). Además, se observó una diferencia de velocidad en escalas filogenéticas aún más pequeñas, especialmente en primates: por ejemplo, el reloj molecular humano corre más lento que el reloj molecular del chimpancé(Elango et al. 2006); las tasas en monos del Nuevo Mundo son más rápidas que las tasas en hominoides y monos del Viejo Mundo (Steiper & Young 2006). Las diferentes tasas de relojes moleculares observadas en estos estudios son cualitativamente consistentes con el efecto de tiempo de generación.
Sin embargo, las diferencias reales entre los linajes no son cuantitativamente consistentes con la diferencia en los tiempos de generación. Por ejemplo, Kumar & Subramanian (2002) mostró que a pesar de que la diferencia en los tiempos de generación entre primates y roedores es mucho mayor que la entre humanos y monos del Viejo Mundo, las diferencias observadas en los relojes moleculares son similares en estas dos comparaciones. Vale la pena señalar que Kumar & Subramanian (2002) utilizó filtros estadísticos específicos para eliminar datos que mostraban patrones de sustitución ‘heterogéneos’, lo que podría haber causado un sesgo hacia secuencias de evolución lenta (Yi et al. 2002). Sin embargo, la diferencia en los relojes moleculares de primates y roedores parece mucho menor de lo propuesto originalmente por Wu & Li (1985). Por ejemplo, Huttley et al. (2007) analizaron las alineaciones del genoma completo de varias especies, incluida la zarigüeya, y mostraron que la diferencia de tasa entre los linajes eutherianos y el linaje de la zarigüeya (~30%) es mucho mayor que la diferencia de tasa entre los linajes humano y ratón (~14%). Estos ejemplos demuestran que el grado de diferencias en los relojes moleculares varía significativamente entre los diferentes estudios debido a las diferencias en los conjuntos de datos y los métodos estadísticos. También muestran que las diferencias de tasas entre linajes no pueden explicarse completamente por la diferencia en los tiempos de generación por sí sola. Claramente, hay otros contribuyentes a los relojes moleculares neutrales.
De hecho, los rasgos de la historia de vida distintos de los tiempos de generación parecen co-variar con los relojes moleculares. Martin & Palumbi (1993) mostró que los relojes moleculares de ADN funcionan más rápido en especies con un tamaño corporal pequeño. Esta observación llevó a la hipótesis de que las tasas metabólicas son determinantes importantes de los relojes moleculares. Una alta tasa metabólica produce un gran número de radicales mutagénicos de oxígeno, lo que aumentaría las tasas de mutación (Rand 1994). Debido a que las tasas metabólicas y el tamaño corporal generalmente co-varían con los tiempos de generación, ha sido difícil distinguir cuál de estos constituye el principal determinante de las tasas de reloj molecular. Tsantes & Steiper (2009) ha propuesto, basándose en datos de primates, que la edad en la primera reproducción, en lugar del tamaño corporal, es el principal determinante de los relojes moleculares. Dado que la edad en la primera reproducción refleja el efecto del tiempo de generación, este estudio apoya la idea de que el efecto del tiempo de generación es el principal determinante del reloj molecular. Sin embargo, este estudio todavía se basa en un número limitado de linajes (se utilizaron cuatro pares de especies). Por lo tanto, distinguir entre el tamaño corporal, el efecto de tiempo de generación y las tasas metabólicas sigue siendo un tema importante para generalizar y comprender los relojes moleculares neutros.
Además, la importancia de los factores que no co-varían con los tiempos de generación, como la metilación del ADN, se ha apreciado cada vez más. La metilación del ADN es una modificación química del ADN genómico que se encuentra en diversos taxones. En los genomas animales, la metilación del ADN ocurre casi exclusivamente en citosinas seguidas de guaninas (las llamadas «CpG»). Citosinas metiladas, a su vez, tienden a mutar rápidamente a timinas debido a la inestabilidad química (Bird 1980). De hecho, en el genoma humano, las mutaciones causadas por la metilación del ADN ocurren más de un orden de magnitud con más frecuencia que otras mutaciones (Nachman & Crowell 2000, Elango et al. 2008). Debido a que las mutaciones causadas por la metilación del ADN ocurren en gran medida independientemente de la replicación del ADN, tales mutaciones pueden seguir diferentes relojes moleculares que otras. Específicamente, en lugar de la dependencia del tiempo de generación, las mutaciones causadas por la metilación del ADN pueden seguir un reloj molecular dependiente del tiempo, que es similar a lo que propuso inicialmente Zuckerkandl & Pauling (Kim et al. 2006)!
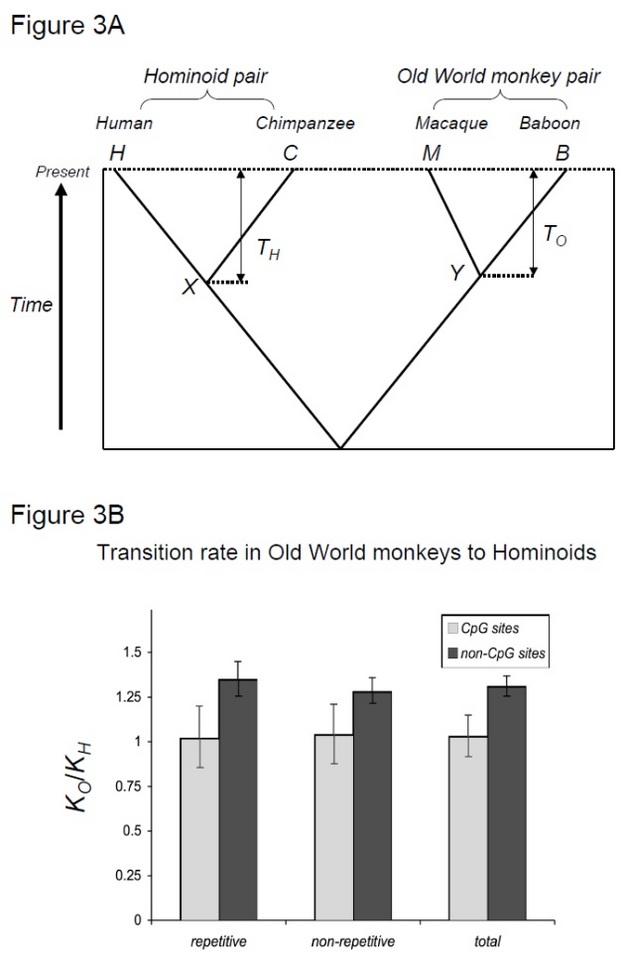
Para probar esta hipótesis, Kim et al. (2006) compararon la divergencia hombre-chimpancé con la divergencia macaco-mandril, dos parejas de especies que comparten tiempos de divergencia similares pero con tiempos de generación diferentes (Steiper et al. 2004). El par humano-chimpancé (el par hominoide) tiene tiempos de generación mucho más largos en comparación con el par macaco-babuino (el par de monos del Viejo Mundo). Este estudio mostró que para sitios no CpG, la pareja de monos del Viejo Mundo acumuló aproximadamente un 30% más de sustituciones, lo que puede explicarse por el efecto de desaceleración de la tasa de hominoides mencionado anteriormente. En contraste, los relojes moleculares en sitios de GpC mostraron números similares de sustituciones en pares de monos hominoides y del Viejo Mundo (Figura 3). Por lo tanto, los relojes moleculares dependientes del tiempo y de la generación coexisten dentro de los mismos genomas. La suposición de que un solo reloj molecular puede existir para un linaje dado ya no es válida, porque las fuerzas mutacionales predominantes varían entre las regiones genómicas.
Conclusiones
El concepto de una constante reloj molecular fue inicialmente propuesto basado en una cantidad limitada de datos de secuencias proteicas. A pesar de que estudios posteriores mostraron que tal observación no es un patrón general en aminoácidos, ha tenido una influencia significativa en el campo de la evolución molecular, en particular en el desarrollo de la teoría neutra de la evolución molecular para los datos de secuencias de ADN. Siguiendo la teoría neutra, los estudios se centraron en dilucidar los patrones de variación en las tasas de mutación neutra. Durante las últimas décadas, hemos observado que los relojes moleculares funcionan a diferentes velocidades entre linajes. Además, el grado de variación podría variar en función de los diferentes tipos de datos y de los métodos estadísticos específicos utilizados. El efecto de tiempo de generación se mantiene a un nivel cualitativo, pero es insuficiente para explicar la variación cuantitativa de las tasas de mutación neutra entre los linajes. Los rasgos de la historia de vida y las mutaciones no dependientes de la replicación, como las causadas por la metilación del ADN, también contribuyen de manera importante a los relojes moleculares genómicos. De hecho, se observan diferentes tipos de relojes moleculares incluso dentro de un genoma, porque las entradas mutacionales predominantes varían entre diferentes regiones genómicas. Por lo tanto, en lugar de asumir un solo reloj molecular neutro para cada genoma, los estudios futuros deberían apuntar a revelar la variación de los relojes moleculares neutros genómicos, para aprender sobre paisajes mutacionales genómicos. Esta información no solo es útil para comprender la materia prima que rige la evolución molecular y la enfermedad genética, sino que también constituye un componente crítico que influye en los análisis genómicos comparativos y funcionales para identificar regiones genómicas funcionales.