In den frühen 1960er Jahren begannen Biologen zu untersuchen, wie sich Proteine in verschiedenen Spezies auf Sequenzebene entwickeln (Zuckerkandl 1962, Margoliash 1963, Doolittle & Blombäck 1964). Zu den analysierten Proteinen gehörten Hämoglobin (Zuckerkandl & Pauling 1962), Cytochrom C (Margoliash 1963) und Fibrinopeptide (Doolittle & Blombäck 1964). Diese frühen Untersuchungen führten zu einer bemerkenswerten Entdeckung: Es schien, dass die Anzahl der Unterschiede zwischen Proteinsequenzen verschiedener Spezies ungefähr proportional zur Zeit seit der Divergenz der Spezies war (Abbildung 1).
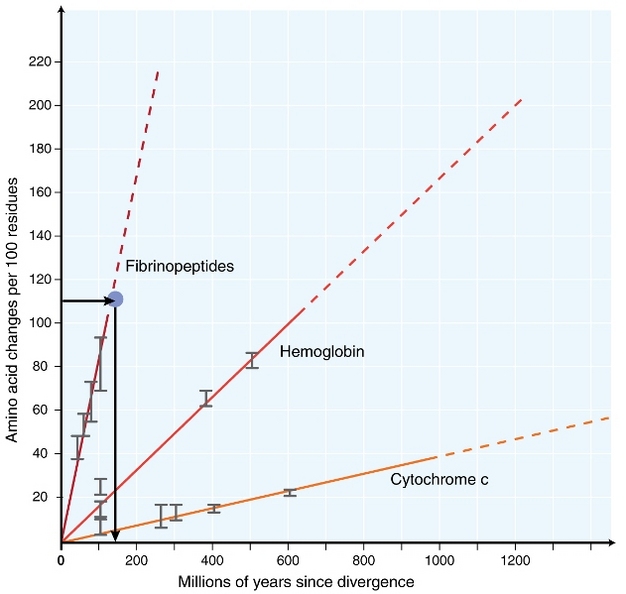
Zuckerkandl & Pauling (1965) verglich die ständige Anhäufung von Aminosäuresubstitutionen im Laufe der Zeit mit regelmäßigen „Ticks“ von Uhren und erklärte, dass „es eine molekulare evolutionäre Uhr geben könnte“. Daher wurde der Begriff ‘molekulare Uhr‘ ursprünglich geprägt, um Veränderungen in Aminosäuren zu beschreiben, die proportional zur Zeit seit der Divergenz der Spezies auftreten.
Seit seiner ersten Verwendung wurde der Begriff ‘molekulare Uhr‘ in vielen verschiedenen Kontexten verwendet. Heutzutage wird es oft verwendet, um sich einfach auf die Anzahl der Änderungen oder ‘Substitutionen‘ zu beziehen, die in der Sequenz von DNA oder Proteinen in einer bestimmten Linie angesammelt sind. Die Anzahl der Substitutionen pro definierter Zeiteinheit kann als ‘Rate‘ der molekularen Uhr beschrieben werden, was in diesem Zusammenhang der ‘evolutionären Rate‘ entspricht. Beachten Sie, dass die anfängliche Konnotation in einigen Fällen immer noch gilt. Zum Beispiel, wenn Leute sagen‘ dass wir eine molekulare Uhr angenommen haben (besonders in phylogenetischen Analysen), bedeutet das, dass die Anzahl der Substitutionen im Laufe der Zeit konstant angenommen wurde.
Das Konzept einer konstanten molekularen Uhr hat außergewöhnliche Implikationen für Evolutionsbiologen. Wenn eine konstante molekulare Uhr, wie ursprünglich vorgeschlagen, wirklich existierte, würde das Ableiten des Zeitpunkts evolutionärer Ereignisse zu einem ziemlich einfachen Problem werden (Abbildung 1). Es ist jedoch überdeutlich geworden, dass Substitutionen im Laufe der Zeit in verschiedenen Linien nicht ständig auftreten (Kumar 2005). Dennoch war das Konzept der molekularen Uhr auf dem Gebiet der molekularen Evolution äußerst einflussreich. Eine der wichtigsten Ideen, die vom Konzept der molekularen Uhr inspiriert sind, ist die neutrale Theorie der molekularen Evolution.
Die molekulare Uhr und Neutralität
Als die Idee einer konstanten molekularen Uhr zum ersten Mal auftauchte, wurde angenommen, dass die vorherrschende evolutionäre Kraft, die Aminosäuren- oder Nukleotidsubstitutionen zugrunde liegt, die natürliche Selektion ist. Nach dieser Denkweise würde eine konstante molekulare Uhr anzeigen, dass adaptive Substitutionen in verschiedenen Arten im Laufe der Zeit ständig auftreten. Es ist jedoch schwer zu erklären, wie adaptive Substitutionen in einer solchen uhrartigen Weise auftreten würden. Theoretisch werden die Schicksale adaptiver Mutationen durch mehrere evolutionäre Parameter bestimmt, wie die Stärke des selektiven Vorteils dieser Mutation, die Größe der effektiven Population und adaptive Mutationsraten (Kimura 1983). Diese Parameter unterscheiden sich wahrscheinlich zwischen den Arten und sogar innerhalb einer Art, abhängig von spezifischen Mutationen und ihren Wechselwirkungen mit der Umwelt. Stattdessen schlug Kimura (1968, 1969) vor, dass die meisten Veränderungen auf molekularer Ebene wenig funktionelle Konsequenzen haben oder ‘neutral‘ sind. Wenn eine Mutation keine gesundheitlichen Folgen hat, wird ihr Schicksal in der Population vollständig durch Zufall bestimmt. Dies bedeutet, dass wir nicht vorhersagen können, ob eine spezifische neutrale Mutation schließlich in der Population fixiert wird. Die Rate, mit der neutrale Substitutionen in der Population auftreten, kann jedoch vorhergesagt werden, da sie von einem einzigen Parameter abhängt, nämlich der Mutationsrate (Kimura 1968).
Stellen wir uns eine Population mit N Anzahl haploider Individuen vor. Wenn neutrale Mutationen mit der Rate u pro Individuum und Generation auftreten, beträgt die Gesamtzahl der Mutationen in einer Generation N-mal u. Da alle diese neuen Mutationen neutral sind, werden ihre Schicksale vollständig durch Zufall bestimmt. Mit anderen Worten, alle Mutationen haben die gleiche Chance, eine Fixierung zu erreichen (was zu einer ‘Substitution‘ führt). Die Wahrscheinlichkeit, dass jede neue neutrale Mutation die Fixierung erreicht, da eine Substitution stattgefunden hat, ist einfach 1 / N. Die Substitutionsrate wird berechnet als die Anzahl der neuen Mutationen in jeder Generation (Nu) multipliziert mit der Wahrscheinlichkeit, dass jede neue Mutation die Fixierung erreicht (1 / N), was gleich u ist. Mit anderen Worten, für neutrale Mutationen ist die Substitutionsrate gleich der Mutationsrate! Wenn also die meisten Mutationen neutral sind (wie in der neutralen Theorie vorgeschlagen) und die Mutationsraten über die Zeit konstant sind, sollten Substitutionen auch über die Zeit konstant auftreten. Wir sollten dann uhrartige regelmäßige Substitutionsraten auf molekularer Ebene beobachten. Kimura (1969) betrachtete daher die Beobachtung einer relativ konstanten molekularen Uhr in Proteinsequenzen als starke Unterstützung für die neutrale Theorie der molekularen Evolution.
Testen, wie die neutrale molekulare Uhr läuft
Nach der neutralen Theorie ist die Frage, ob Substitutionsraten über die Zeit konstant sind oder nicht, äquivalent dazu, ob neutrale Mutationsraten über die Zeit konstant sind. Aus diesem Grund konzentrierten sich viele nachfolgende Studien auf die Analyse von Daten von neutralen Stellen, um festzustellen, ob neutrale Mutationsraten tatsächlich über die Zeit konstant sind. Wir werden kurz überprüfen, wie diese Studien durchgeführt werden, bevor wir theoretische Debatten über molekulare Uhren diskutieren. Die Analysen der proteinmolekularen Uhren wurden ebenfalls fortgesetzt, aber die Debatten um die Variation der proteinmolekularen Uhren unterscheiden sich sehr von denen, die neutrale molekulare Uhren betreffen, und werden in diesem Artikel nicht enthalten sein. Interessierte Leser sollten Gillespie (1991), Kumar (2005), Kim und Yi (2008) und Bedford et al. (2008). Die meisten empirischen Analysen neutraler molekularer Uhren beruhen auf dem Theorem, dass neutrale Mutationsraten aus neutralen Substitutionsraten abgeleitet werden können (Kimura 1968, 1969). In der Praxis definiert jede Studie eine bestimmte Art von Stellen im Genom als neutrale Stellen und vergleicht die Substitutionsraten dieser Stellen zwischen den Linien.
Welche Stellen im Genom wirklich neutral sind, kann nicht vollständig bestimmt werden, aber Wissenschaftler haben mehrere nützliche Proxies entwickelt. Vor der Ära der Genomsequenzierung waren die meisten verfügbaren Sequenzdaten diejenigen aus proteinkodierenden DNA-Sequenzen. Studien unterteilen proteinkodierende DNA-Sequenzen häufig in zwei Arten von Stellen (Wu & Li 1985). Die erste Art von Sites umfasst diejenigen, für die jede Änderung zu Aminosäuresubstitutionen führen würde, oder ‘nonsynonymous Sites‘. Die zweite Art von Sites, ‘synonyme Sites‘, umfasst diejenigen, die ‘degenerierte‘ Positionen in der Codontabelle kodieren, bei denen eine Änderung nicht zu einer Aminosäuresubstitution führt. Zum Beispiel kodieren TCT und TCC beide Serin. Wenn sich die dritte Position dieser Codons ändert, produzieren sie immer noch die gleichen Aminosäuren. Solche Substitutionen wären für die natürliche Selektion weniger sichtbar. Folglich sollten molekulare Uhren an Synonymstellen näher an der neutralen molekularen Uhr liegen als nichtsynonyme Uhren.
Mit fortschreitenden Sequenzierungstechniken verwendeten einige Studien Introns als Proxies für neutrale Stellen, da sie nicht in die reifen mRNAs eingebaut sind und daher eher neutral sind (Yi et al. 2002). Häufig wurden auch Sequenzen inaktiver transponierbarer Elemente eingesetzt, die lange vor der Divergenz der Spezies eingefügt wurden (diese werden oft als ‘Ancestral Repeats‘ bezeichnet, z. B. Thomas et al., 2003). Schließlich verwendeten einige Studien nicht-kodierende DNA-Sequenzen (alle Sequenzen nach Entfernung von Protein-kodierenden DNA-Sequenzen), die aus ganzen Genom-Alignments extrahiert wurden, um neutrale molekulare Uhren zu testen (Elango et al. 2006, Huttley et al. 2007).
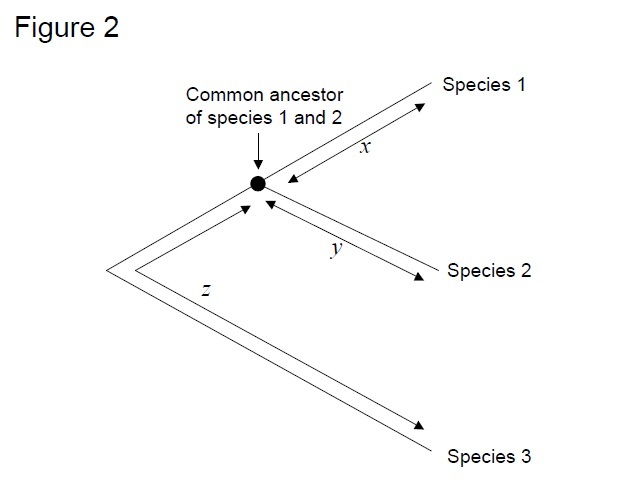
Der am häufigsten verwendete Test ist der sogenannte „relative Rate test“ (Sarich & Wilson 1973). Zunächst wurden die Substitutionsraten pro Zeiteinheit geschätzt, indem die Gesamtzahl der Unterschiede (Substitutionen) zwischen Proteinen verschiedener Spezies durch die aus fossilen Aufzeichnungen geschätzte Divergenzzeit dividiert wurde (Abbildung 1). Fossile Aufzeichnungen sind jedoch für viele Vergleiche nicht verfügbar und mit großen Fehlermargen verbunden. Die relative Rate Test überwindet die Notwendigkeit für fossile Aufzeichnungen (Abbildung 2). Solange eine Outgroup-Sequenz zu den beiden interessierenden Linien existiert, können wir bestimmen, ob die beiden Zweige der gleichen oder verschiedenen molekularen Uhren folgen, ohne die absolute Divergenzzeit zu kennen (Abbildung 2).
Was sind die Determinanten neutraler molekularer Uhren?
Fast alle Kontroversen, die im Mittelpunkt der Debatten über neutrale molekulare Uhren stehen, beruhen auf der Frage, was die Hauptursachen für Mutationen sind. Diese Frage ist direkt relevant für das Verständnis von Mutationsmustern, die die ultimative Quelle für evolutionäre Veränderungen und genetische Krankheiten sind. Darüber hinaus ist das Verständnis, wie Mutationsraten zwischen Linien und innerhalb von Genomen variieren, eine grundlegende Frage in der vergleichenden Genomik, die darauf abzielt, Sequenzvergleiche zu verwenden, um genomische Regionen zu identifizieren, die funktionell wichtig sind.
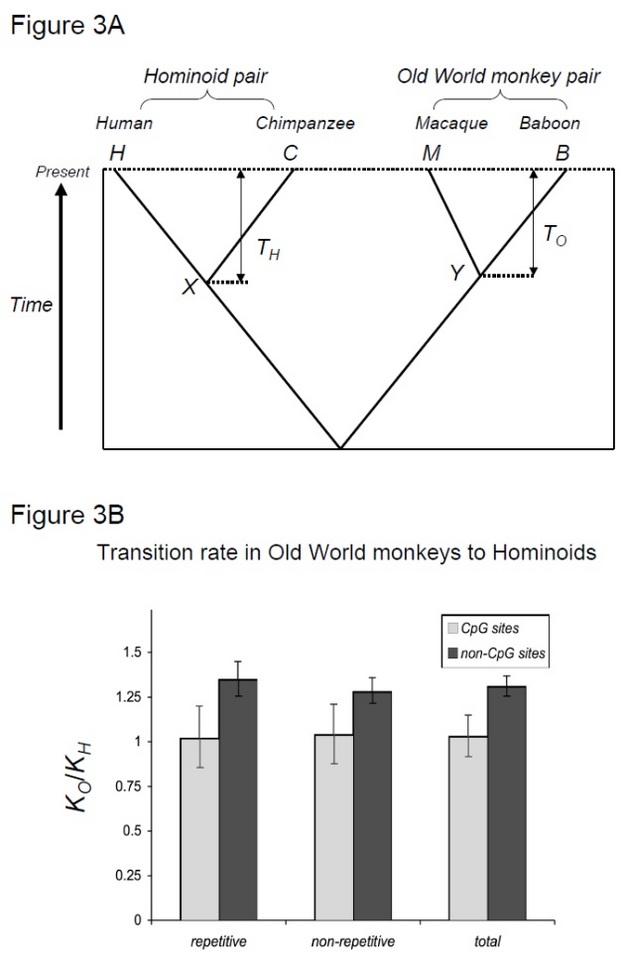
Also, was bestimmt neutrale Mutationsraten? Einer der wichtigsten Faktoren für neutrale molekulare Uhren ist die abstammungsspezifische Variation der Generationszeiten. Schon früh wurde die Idee einer konstanten neutralen molekularen Uhr als im Widerspruch zu den molekularen Mechanismen der Keimbahnmutation stehend wahrgenommen. Es wurde lange angenommen, dass die meisten Mutationen durch Fehler bei der DNA-Replikation in Keimbahnen entstehen (Haldane 1947, Müller 1954). Da Mutationen auftreten, wenn Keimbahn-DNA für die nächste Generation repliziert wird, sollten sie sich proportional zur Anzahl der Generationen und nicht zur absoluten Zeit ansammeln. Wenn wir also die Anzahl der Substitutionen vergleichen, die sich seit ihrer Divergenz in zwei Linien angesammelt haben, würde die Linie mit längerer Generationszeit, die weniger DNA-Replikationsereignisse durchlaufen hat, weniger Substitutionen aufweisen als die Linie mit der kürzeren Generationszeit. Folglich sollte die molekulare Uhr bei Arten mit längeren Generationszeiten langsamer laufen. Diese Idee wird als ‘Generation-Time-Effekt‘ bezeichnet.Tatsächlich wurde der Generationszeiteffekt erstmals in Studien an Primaten beobachtet, noch vor der Debatte über die molekulare Uhr. Morris Goodman, der zu dieser Zeit immunologische Methoden zur Untersuchung der Artverwandtschaft verwendete, beobachtete, dass die Rate, mit der einige Proteine divergieren, bei Affen, insbesondere beim Menschen, im Vergleich zu Affen der Alten Welt verringert zu sein schien (Goodman 1961, 1962, 1963). Dieser Effekt wird als ‘Hominoid Rate Slowdown‘ bezeichnet. Da Hominoide längere Generationszeiten haben als Altweltaffen, Diese Beobachtung kann durch den Generationszeiteffekt erklärt werden.
Wu & Li (1985) waren die ersten, die die Hypothese des Generationszeiteffekts anhand von DNA-Sequenzdaten testeten. Sie verwendeten Daten von 11 Genen von Primaten und Nagetieren. Da Primaten eine viel längere Generationszeit haben als Nagetiere, sollte die molekulare Uhr bei Nagetieren im Vergleich zu Primaten schneller sein. Tatsächlich fanden sie heraus, dass Nagetiere für Synonymstellen im Vergleich zu Primaten etwa die zweifache Geschwindigkeit der molekularen Evolution aufweisen (Wu & Li 1985). Für nicht-anonyme Websites wurde jedoch kein solcher Effekt gefunden. Mit anderen Worten, die neutrale molekulare Uhr, aber nicht die aminosäuremolekulare Uhr, tickt in der Nagetierlinie schneller als in der Primatenlinie, was gut zur Idee eines Generationszeiteffekts passt.
Nachfolgende Studien lieferten weitere Unterstützung für die Verlangsamung der hominoiden Rate (Li & Tanimura 1987, Bailey et al. 1991) und die Ratendifferenz zwischen den Nagetier- und Primatenlinien (Gu & Li 1992, Huttley et al. 2007). Darüber hinaus wurde ein Ratenunterschied in noch kleineren phylogenetischen Skalen beobachtet, insbesondere bei Primaten: zum Beispiel läuft die molekulare Uhr des Menschen langsamer als die molekulare Uhr des Schimpansen (Elango et al. 2006); Raten bei Neuweltaffen sind schneller als die Raten bei Hominoiden und Altweltaffen (Steiper & Young 2006). Die in diesen Studien beobachteten unterschiedlichen Raten molekularer Uhren stimmen qualitativ mit dem Generationszeiteffekt überein.
Die tatsächlichen Unterschiede zwischen den Linien stimmen jedoch quantitativ nicht mit dem Unterschied in den Generationszeiten überein. Zum Beispiel zeigte Kumar & Subramanian (2002), dass, obwohl der Unterschied in den Generationszeiten zwischen Primaten und Nagetieren viel größer ist als der zwischen Menschen und Altweltaffen, die beobachteten Unterschiede in den molekularen Uhren in diesen beiden Vergleichen ähnlich sind. Es ist erwähnenswert, dass Kumar & Subramanian (2002) spezifische statistische Filter verwendete, um Daten zu entfernen, die „heterogene“ Substitutionsmuster zeigten, die möglicherweise eine Tendenz zu sich langsam entwickelnden Sequenzen verursacht haben (Yi et al. 2002). Dennoch scheint der Unterschied in den molekularen Uhren von Primaten und Nagetieren viel geringer zu sein als ursprünglich von Wu & Li (1985) vorgeschlagen. Zum Beispiel, Huttley et al. (2007) analysierten die Ausrichtung des gesamten Genoms mehrerer Arten, einschließlich des Opossums, und zeigten, dass der Ratenunterschied zwischen eutherianischen Linien und der Opossum-Linie (~ 30%) viel größer ist als der Ratenunterschied zwischen menschlichen und Mauslinien (~ 14%). Diese Beispiele zeigen, dass der Grad der Unterschiede in den molekularen Uhren zwischen verschiedenen Studien aufgrund von Unterschieden in Datensätzen und statistischen Methoden signifikant variiert. Sie zeigen auch, dass Ratenunterschiede zwischen den Linien nicht vollständig durch den Unterschied in den Generationszeiten allein erklärt werden können. Offensichtlich gibt es andere Mitwirkende an neutralen molekularen Uhren. Tatsächlich scheinen andere Merkmale der Lebensgeschichte als die Generationszeiten mit den molekularen Uhren zu variieren. Martin & Palumbi (1993) zeigte, dass molekulare DNA-Uhren bei Spezies mit kleiner Körpergröße schneller laufen. Diese Beobachtung führte zu der Hypothese, dass Stoffwechselraten wichtige Determinanten molekularer Uhren sind. Eine hohe Stoffwechselrate erzeugt eine große Anzahl mutagener Sauerstoffradikale, was die Mutationsraten erhöhen würde (Rand 1994). Da Stoffwechselraten und Körpergröße im Allgemeinen mit den Generationszeiten variieren, Es war schwierig zu unterscheiden, welche davon die Hauptdeterminante der molekularen Uhrraten darstellt. Tsantes & Steiper (2009) haben basierend auf Daten von Primaten vorgeschlagen, dass das Alter bei der ersten Fortpflanzung und nicht die Körpergröße die Hauptdeterminante der molekularen Uhren ist. Da das Alter bei der ersten Reproduktion den Generationszeiteffekt widerspiegelt, unterstützt diese Studie die Idee, dass der Generationszeiteffekt die Hauptdeterminante der molekularen Uhr ist. Diese Studie basiert jedoch immer noch auf einer begrenzten Anzahl von Linien (vier Artenpaare wurden verwendet). Daher ist die Unterscheidung zwischen Körpergröße, Generationszeiteffekt und Stoffwechselraten nach wie vor ein wichtiges Thema bei der Verallgemeinerung und dem Verständnis neutraler molekularer Uhren.
Darüber hinaus wurde die Bedeutung von Faktoren, die nicht mit den Generationszeiten variieren, wie DNA-Methylierung, zunehmend geschätzt. DNA-Methylierung ist eine chemische Modifikation der genomischen DNA in verschiedenen Taxa gefunden. In tierischen Genomen erfolgt die DNA-Methylierung fast ausschließlich an Cytosinen, gefolgt von Guaninen (sogenannten CPG‘s). Methylierte Cytosine wiederum neigen aufgrund chemischer Instabilität dazu, schnell zu Thyminen zu mutieren (Bird 1980). Tatsächlich treten im menschlichen Genom Mutationen, die durch DNA-Methylierung verursacht werden, mehr als eine Größenordnung häufiger auf als andere Mutationen (Nachman & Crowell 2000, Elango et al. 2008). Da Mutationen, die durch DNA-Methylierung verursacht werden, weitgehend unabhängig von der DNA-Replikation auftreten, können solche Mutationen anderen molekularen Uhren folgen als andere. Insbesondere können Mutationen, die durch DNA-Methylierung verursacht werden, anstelle einer Generationszeitabhängigkeit einer zeitabhängigen molekularen Uhr folgen, die dem ähnelt, was ursprünglich von Zuckerkandl & Pauling (Kim et al. 2006)!
Um diese Hypothese zu testen, haben Kim et al. (2006) verglich die Mensch-Schimpansen-Divergenz mit der Makaken-Pavian-Divergenz, zwei Artenpaaren, die ähnliche Divergenzzeiten, aber unterschiedliche Generationszeiten aufweisen (Steiper et al. 2004). Das Mensch-Schimpansen-Paar (das Hominoid-Paar) hat viel längere Generationszeiten als das Makaken-Pavian-Paar (das Affenpaar der Alten Welt). Diese Studie zeigte, dass das Affenpaar der Alten Welt für Nicht-CpG-Standorte ungefähr 30% mehr Substitutionen akkumulierte, was durch den oben genannten Effekt der Verlangsamung der Hominoidrate erklärt werden kann. Im Gegensatz dazu zeigten molekulare Uhren an CpG-Standorten eine ähnliche Anzahl von Substitutionen in hominoiden und Altweltaffenpaaren (Abbildung 3). Somit existieren zeitabhängige und generationszeitabhängige molekulare Uhren innerhalb desselben Genoms nebeneinander. Die Annahme, dass eine einzelne molekulare Uhr für eine bestimmte Linie existieren kann, ist nicht mehr gültig, da die vorherrschenden Mutationskräfte zwischen den genomischen Regionen variieren.
Schlussfolgerungen
Das Konzept einer konstanten molekularen Uhr wurde ursprünglich auf der Grundlage einer begrenzten Menge von Proteinsequenzdaten vorgeschlagen. Obwohl nachfolgende Studien zeigten, dass eine solche Beobachtung kein allgemeines Muster in Aminosäuren ist, hatte sie einen signifikanten Einfluss auf das Gebiet der molekularen Evolution, insbesondere auf die Entwicklung der neutralen Theorie der molekularen Evolution für DNA-Sequenzdaten. Nach der neutralen Theorie konzentrierten sich die Studien auf die Aufklärung von Variationsmustern in neutralen Mutationsraten. In den letzten Jahrzehnten haben wir beobachtet, dass molekulare Uhren zwischen den Linien unterschiedlich schnell laufen. Darüber hinaus kann der Grad der Variation in Abhängigkeit von den verschiedenen Datentypen und den verwendeten spezifischen statistischen Methoden variieren. Der Generationszeiteffekt hält weiterhin auf qualitativer Ebene an, reicht jedoch nicht aus, um die quantitative Variation neutraler Mutationsraten zwischen den Linien zu erklären. Lebensgeschichtliche Merkmale und nicht replikationsabhängige Mutationen, wie sie durch DNA-Methylierung verursacht werden, sind ebenfalls wichtige Faktoren für genomische molekulare Uhren. Tatsächlich werden sogar innerhalb eines Genoms verschiedene Arten molekularer Uhren beobachtet, da die vorherrschenden Mutationseingaben zwischen verschiedenen genomischen Regionen variieren. Anstatt also eine einzelne neutrale molekulare Uhr für jedes Genom anzunehmen, sollten zukünftige Studien darauf abzielen, die Variation genomischer neutraler molekularer Uhren aufzudecken, um mehr über genomische Mutationslandschaften zu erfahren. Solche Informationen sind nicht nur nützlich für das Verständnis des Rohmaterials, das die molekulare Evolution und genetische Erkrankungen steuert, sondern stellen auch eine kritische Komponente dar, die vergleichende und funktionelle genomische Analysen zur Identifizierung funktioneller genomischer Regionen beeinflusst.