Introduction
Die internationale Naturschutzpolitik erkennt die biologische Vielfalt auf drei Ebenen an: Ökosystem, Arten und Genetik, und das Management sollte darauf abzielen, alle drei zu erhalten (Übereinkommen über die biologische Vielfalt, 2007). Dies spiegelt sich deutlich in der 2010 vereinbarten Konvention über die biologische Vielfalt Aichi Biodiversity Targets wider (https://www.cbd.int/sp/targets/), in der Ziele und Vorgaben nicht nur auf Ökosysteme und Arten, sondern auch auf die genetische Vielfalt Bezug nehmen. Die derzeitigen Ansätze zur Erhaltung der biologischen Vielfalt basieren jedoch weitgehend auf geografischen Gebieten, Ökosystemen, ökologischen Gemeinschaften und Arten, wobei der genetischen Vielfalt und dem Kontinuum zwischen Arten und Populationen weniger Aufmerksamkeit geschenkt wird. Tatsächlich konzentriert sich die Mehrheit der Naturschutzfachleute auf Arten als Metrik der biologischen Vielfalt, wobei intraspezifische Einheiten innerhalb intensiv bewirtschafteter bedrohter Arten berücksichtigt werden. In diesem Zusammenhang gibt es einen natürlichen Wunsch, einfache Kategorisierungen zu verwenden — benannte Arten und andere benannte Taxa (d. H. Unterarten) und manchmal Erhaltungseinheiten innerhalb von Arten. Insbesondere Arten stellen ein gemeinsames Maß für die biologische Vielfalt dar, das viele wissenschaftliche und Managementbemühungen untermauert (Mace, 2004; Wilson, 2017). Als grundlegende Einheit der Erhaltung, Arten werden häufig verwendet, um den Wert der biologischen Vielfalt durch Artenlisten in Schutzgebieten zu quantifizieren, Identifizierung bedrohter Arten innerhalb von Gerichtsbarkeiten, und als Grundlage für Biosicherheitsverfahren. Darüber hinaus sind Arten in der Regel die Einheiten, mit denen wir Naturschutzfragen an die Öffentlichkeit kommunizieren.
Dieser Impuls, diskrete Einheiten in der Natur einfach zu identifizieren, widerspricht mehreren, verwandten Realitäten. Erstens ist die Speziation nicht augenblicklich. Vielmehr handelt es sich typischerweise um einen langwierigen Prozess (Dynesius und Jansson, 2014; Roux et al., 2016), die über lange Zeiträume von isolierten Populationen, die möglicherweise unterschiedlichem Selektionsdruck unterliegen, zu eindeutigen Arten fortschreiten, wobei häufige Umkehrungen durch Umweltveränderungen oder einfache genetische Verschmelzung verursacht werden, wenn sich die Bereiche wieder verbinden (Abbildung 1; Seehausen et al., 2008; Rosenblum et al., 2012). Zweitens sind die meisten anerkannten Arten nicht genetisch einheitlich. Divergenz (Drift und langwierige Isolation) und Retikulation (Genfluss, Hybridisierung, Introgression und Rekombination) prägen effektiv die geografische und genomische Variation innerhalb der Arten (Cutter und Payseur, 2013; Edwards et al., 2016) und einige Arten können in lange isolierte Populationen strukturiert werden, von denen einige die Anerkennung als vollständige, aber vielleicht morphologisch kryptische Arten rechtfertigen könnten (Bickford et al., 2007; Jorger und Schrödl, 2013). Drittens, so lange anerkannt, sind benannte Arten nicht gleichwertig in Bezug darauf, wie viel evolutionäres Erbe sie repräsentieren — einfach ausgedrückt, ein Schnabeltier (als Vertreter einer alten divergierenden Linie) kann als mehr wert angesehen werden als ein kürzlich divergierendes Nagetier (Isaac et al., 2007). Viertens sind Artenbezeichnungen nicht in Stein gemeißelt, sondern Hypothesen, die getestet werden müssen, sobald neue Beweise verfügbar werden (Hey et al., 2003). Jedes dieser Probleme schafft Komplexität und Mehrdeutigkeit bei der Abgrenzung von Arten im Allgemeinen und der Zuweisung von Erhaltungsaufmerksamkeit für sie oder ihre Komponentenpopulationen. Darüber hinaus unterliegt die Abgrenzung von Arten auch unterschiedlichen Artenkonzepten und Diagnosemethoden, was bei Naturschutzbiologen große Bestürzung hervorgerufen hat (Mace, 2004; Frankham et al., 2012; Garnett und Christidis, 2017).
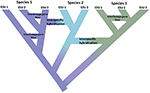
Abbildung 1. Speziation als Kontinuum, das von isolierten Populationen zu eindeutigen Arten fortschreitet, die durch die Farbverschiebung mit den Farben, die verschiedene Arten darstellen, gezeigt werden. Einige Abstammungslinien, die aufgrund der Isolation der Population entstehen, entwickeln sich zu echten Arten, wobei die internen Knoten des Baumes die Einleitung des Speziationsprozesses darstellen. Andere Abstammungslinien entwickeln sich nicht zu echten Arten, sondern divergieren im Laufe der Zeit, was darauf hinweist, dass Arten genetisch nicht einheitlich sind. Diese Linien werden als evolutionär signifikante Einheiten (ESUs) dargestellt. Der Genfluss zwischen den Linien ist für Spezies 1 und Spezies 3 angegeben. In diesem Fall kann die evolutionäre Unabhängigkeit vorübergehend sein, da zwei Linien derselben Art nicht reproduktiv isoliert sind. Während sie als unterschiedliche Linien (Arten) bleiben, kann eine interspezifische Hybridisierung mit begrenztem Genaustausch (Farbverschiebung) zwischen Arten auftreten.Die Umsetzung wirksamer Erhaltungsstrategien, die darauf abzielen, sowohl Arten als auch die genetische Vielfalt zu schützen, würde sowohl von einer konsistenteren Sichtweise darüber profitieren, wie wir Arten und andere Einheiten im gesamten Populations-Arten-Kontinuum abgrenzen und wie diese Einheiten in verschiedenen Gesetzen weltweit interpretiert werden. Neue Fähigkeiten aus der Genomik bieten sowohl Chancen als auch Herausforderungen, um diese Ziele zu erreichen. Im Folgenden betrachten wir: (i) wie Analysen auf Genomebene mit einem konservativen Ansatz zur Abgrenzung und Benennung von Arten in Einklang gebracht werden können, während gleichzeitig Hauptkomponenten der genetischen Vielfalt innerhalb von Arten identifiziert werden, (ii) wie bestehende Naturschutzgesetze auf mehreren Kontinenten, die „Arten“ definieren, um eine breite Palette von infraspezifischen Kategorien einzuschließen, angesichts der hohen Auflösung, die Genomscreens bieten, verwendet werden können, und (iii) wie die Erkenntnisse über Speziationsprozesse und Strukturierung von Variationen aus der Genomik mit der Gesetzgebung und dann mit dem Management in Einklang gebracht werden. Wir schlagen dann einige Wege vor, um Naturschutzeinheiten mit legislativen Ansätzen in Einklang zu bringen, um das gesamte Spektrum der Vielfalt von Populationen durch Arten zu schützen.
Genomik, Speziation und Taxonomie
Wie in vielen Bereichen der biologischen Wissenschaft wird die Systematik, die Praxis der Benennung von Taxa und das Verständnis ihrer Beziehungen und ihrer Entstehung durch Genomik und statistische Modelle erheblich vorangetrieben. Ob durch Ganzgenomsequenzierung (z.B. Nater et al., 2017) oder genome Sampling (SNPs, target Capture-sequencing; Jones und Good, 2016; Leache und Oaks, 2017) haben wir jetzt eine beispiellose Auflösung von Mustern der genetischen Vielfalt (Funk et al., 2012). In Verbindung mit immer leistungsfähigeren statistischen Modellen zur Ableitung von Genom- und Artendivergenzverläufen liefern diese Daten neue Einblicke in die Evolutionsprozesse, die Arten und genetische Variation erzeugen.
Die Anwendung dieser neuen Werkzeuge erzeugt zwei gegensätzliche Erkenntnisse. Einerseits zeigen viele Studien sehr unterschiedliche genetische Populationen innerhalb benannter Arten, sogenannter kryptischer Arten (Bickford et al., 2007; Jorger und Schrödl, 2013; Struck et al., 2017). Dies ist nichts Neues, da es auf einer langen Geschichte der räumlichen Populationsgenetik und molekularen Phylogeographie aufbaut, die seit drei Jahrzehnten ein produktives Unterfangen ist (Avise et al., 1987). Was die Genomik zu diesem Bestreben bringt, ist sowohl eine erhöhte Auflösung der Bevölkerungsstruktur als auch Daten, die ausreichen, um statistisch auf Bevölkerungsverläufe zu schließen und alternative Divergenzmodelle zu testen (Degnan und Rosenberg, 2009; Sukumaran und Knowles, 2017). Andererseits zeigen Analysen auf Genomebene auch, dass (wie für Pflanzen seit langem bekannt) der genetische Austausch (Introgression) zwischen Tierarten sowohl während als auch nach der Artbildung häufiger ist als bisher angenommen und sogar neue adaptive Strahlungen auslösen kann (Mallet, 2007; Rieseberg und Willis, 2007; Arnold und Kunte, 2017) Abbildung 1. Zusammengenommen unterstreichen diese Erkenntnisse weiter, dass die Speziation langwierig ist (Roux et al., 2016) und zeigen die entgegengesetzten Kräfte auf, die durch das Phylogeographie-Phylogenetik-Kontinuum wirken (Edwards et al., 2016).
Wie hängt das alles mit Taxonomie und Naturschutzzielen zusammen? Es gibt starke und berechtigte Bedenken, dass die erhöhte Auflösung, die Genomscreens bieten, zu einer grassierenden Überspaltung von Arten führen könnte (Isaac et al., 2004), die möglicherweise die Flexibilität des Managements einschränken und kleine genetisch unterschiedliche Populationen der Inzucht und dem eventuellen Aussterben aussetzen (Frankham et al., 2012). Umgekehrt haben Gippoliti et al. (2018) argumentieren, dass die taxonomische Trägheit, die dazu führte, dass viele Arten afrikanischer Huftiere zuvor nicht erkannt wurden, zu einer Depression der Auszucht nach dem Mischen und dem Versäumnis geführt hatte, geografisch eingeschränkte Taxa zu schützen. Ihre Ansicht (und siehe auch Morrison et al., 2009) ist, dass es keine Hinweise darauf gibt, dass die taxonomische Inflation negative Auswirkungen auf den Huftierschutz hat (Gippoliti et al., 2018). Ein damit verbundenes Problem ist, dass eine instabile Taxonomie sowohl die Naturschutzpraxis als auch die öffentliche Wahrnehmung verzögert (Garnett und Christidis, 2017). Diese Bedenken werden teilweise durch die lang andauernde Debatte über Artenkonzepte und Diagnosemethoden in der systematischen Biologie angetrieben, insbesondere durch die Anwendung des phylogenetischen Artenkonzepts, das besagt, dass Arten minimal diagnostizierbare Einheiten sind (Cracraft, 1983). Die Mehrheit der Taxonomen ist jedoch von Natur aus konservativ und benennt Arten nur dann, wenn Übereinstimmungen zwischen mehreren Beweislinien bestehen (Dayrat, 2005; Padial et al., 2010; Schlick-Steiner et al., 2010: Köhler et al., 2011). Wir sind der Ansicht, dass Arten robuste, sich unabhängig entwickelnde Linien darstellen sollten, die im Kontakt mit nahen Verwandten weitgehend intakt bleiben — in Übereinstimmung mit der Absicht des evolutionären Artenkonzepts (Wiley, 1978) und des generalisierten Linienartenkonzepts (de Queiroz, 1998, 2007). In der Praxis erfordert dies den direkten Nachweis einer reproduktiven Isolation oder vernünftiger Surrogate derselben, während ein begrenzter genetischer Austausch ermöglicht wird (Singhal et al., 2018).
Genomische Daten bieten mehr Möglichkeiten als zuvor, Differenzierung und Divergenz innerhalb und zwischen Arten zu identifizieren, was eine stärkere Berücksichtigung der Auswirkungen auf die Abgrenzung taxonomischer Einheiten erfordert. Wo Populationen sowohl genetisch als auch phänotypisch stark von bereits benannten Arten abweichen, gibt es starke Gründe, diese als separate Arten zu bezeichnen. Wenn Populationen phänotypisch stark divergieren und nur wenig genomisch getrennt sind, wird beurteilt, ob diese phänotypischen Unterschiede (i) vererbbar sind und (ii) wahrscheinlich eine substanzielle reproduktive Isolation verursachen. Genom-Scans können solche Situationen lösen, indem sie auf eingeschränkten Genfluss testen. Das schwierigere Problem ist, wo morphologisch ähnliche, aber genetisch divergierende Populationen nachgewiesen werden. Solche Populationen können leicht als separate Evolutionslinien diagnostiziert werden, indem statistische Abgrenzungsmethoden auf multifokale Daten angewendet werden (Carstens et al., 2013; Rannala, 2015), aber sind sie ephemere Populationen oder dauerhafte Arten (Sukumaran und Knowles, 2017)? Es ist durchaus möglich, innerhalb der Regeln der Nomenklatur Arten allein aus DNA-Sequenzen zu erkennen, was eine sequenzbasierte Diagnostik ermöglicht (Tautz et al., 2003; Koch et al., 2010). Zum Beispiel Murphy et al. (2015) begrenzen und beschreiben sechs australische Wüstenfrühlings-Amphipodenarten basierend auf DNA-Sequenzdaten, obwohl keine klaren morphologischen Unterschiede bestehen. Dies ist jedoch ein allgemeiner Bereich für Verbesserungen, in dem Systematiker den Begriff „kryptische Spezies“ konsistenter und strenger anwenden müssen (Struck et al., 2017).
In Situationen, in denen DNA-Sequenzdaten ein potenzielles Schlüsselmerkmal für die Erkennung einer Spezies darstellen, schlagen wir vor, dass es besonders wichtig ist, andere Beweislinien anzuwenden, um eine Überspaltung zu vermeiden (Oliver et al., 2015). Ein Ansatz besteht darin, auf eine erhebliche reproduktive Isolation zu testen, wenn solche Populationen natürliche Kontaktzonen bilden. In einer vergleichenden genomischen Analyse von Kontaktzonen zwischen phänotypisch nicht unterscheidbaren Abstammungspaaren von Carlia-Eidechsen korrelierten beispielsweise die Indizes der genetischen Isolation stark mit der Divergenzzeit (Singhal und Moritz, 2013; Singhal und Bi, 2017) (Abbildung 2). Die divergenteren dieser Abstammungspaare wurden nun formal als separate Arten anerkannt, während eng verwandte (aber statistisch diagnostizierbare) Abstammungslinien mit Hinweisen auf einen genomisch ausgedehnten genetischen Austausch über Kontaktzonen hinweg dies nicht waren (Singhal et al., 2018). Angesichts der starken empirischen Beziehung zwischen Divergenz und reproduktiver Isolation in diesen Taxa trennten diese Autoren zwei allopatrische und kryptische Isolate (einer eng verwandten Spezies), die eine noch höhere genomische Divergenz aufwiesen als die oben abgegrenzten. Die Ausweitung dieser reproduktiven Isolation auf andere kongenere Arten mit stark divergierenden, aber morphologisch ähnlichen phylogeographischen Linien (Potter et al., 2016, 2018; Afonso Silva et al., 2017a) legt nahe, dass noch mehr Arten beschrieben werden müssen, insbesondere auf Inseln (Abbildung 2, Carlai amax und C. rufilatus), und tatsächlich wurden einige solcher Arten nach komplementärer phänotypischer Analyse beschrieben (Afonso Silva et al., 2017b). Andere Abstammungslinien mit neueren Divergenzen, die jedoch statistisch immer noch als separate Abstammungslinien unter Verwendung eines großen Satzes von Kerngenen abgegrenzt sind, können als evolutionär signifikante Einheiten (ESUs) innerhalb von Arten dargestellt werden.
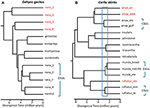
Abbildung 2. Phylogenomische Auflösung von Artengrenzen bei Eidechsen aus den australischen Monsuntropen (AMT). In jedem Fall werden Beziehungen und relative Divergenzzeiten aus 100 Exons unter Verwendung der in StarBEAST2 implementierten Multispezies-Koaleszenz geschätzt, aber die Zeitskalen zwischen den beiden Beispielen sind nicht unbedingt gleich. (A) Ergebnisse für die Nana-Gruppe der Gehyra-Geckos (Moritz et al., 2018). Die phylogenetische Vielfalt der Abstammungslinien innerhalb von G. nana umfasste, wie bereits bekannt, vier weitere bereits beschriebene und morphologisch unterschiedliche Arten. Nachfolgende Analysen von Bereichsüberschneidungen und Morphologie haben dazu geführt, dass jede der phylogenetisch am stärksten divergierenden Linien (rote Taxonmarkierungen) als separate Arten erkannt wurde (Doughty et al., 2018). Die (morphologisch untrennbaren) Linien, die in G. nana verbleiben, wie sie jetzt erkannt werden, können als ESUs betrachtet werden. (B) Laufende Auflösung der Artengrenzen in Carlia Skinks aus dem AMT. Einige Abstammungslinien wurden bereits als Arten anerkannt, basierend auf einer Kombination aus genetischer Artabgrenzung und Post-hoc-Identifizierung diagnostischer morphologischer Unterschiede (C. insularis und C. isostriacantha; Afonso Silva et al., 2017b). Für andere Komplexe (Potter et al., 2016, 2018), taxonomische Revisionen sind im Gange. Das blaue Rechteck zeigt den Bereich der Divergenzzeiten an, in denen in Kontaktzonen zwischen morphologisch kryptischen Linien verwandter Arten eine erhebliche reproduktive Isolation, die mit der Divergenz korreliert, beobachtet wurde (Singhal und Bi, 2017). Konservativ und folgend (Singhal et al., 2018) erkennen wir als Kandidatenarten drei genetische Linien mit Divergenzen, die größer als diese empirische Schwelle sind. Dies würde genetisch unterschiedliche ESUs innerhalb von Carlia amax, Carlia munda und Carlia rufilatus (blaue Klammern) hinterlassen. Die Skalenbalken stellen Schätzungen der Divergenzzeit in Millionen von Jahren dar.
Im Extremfall zeigen genomische Methoden, dass einzelne taxonomische Arten nicht nur genetisch heterogen sind, sondern im phylogenetischen Sinne auch andere benannte Arten enthalten. Eine solche „Paraphylie“ kann durch die jüngste Speziation aus einer einzigen, genetisch unterteilten Art entstehen (Patton und Smith, 1994), erzeugt aber auch die Hypothese, dass das genetisch heterogene Taxon mehrere Arten repräsentieren kann. Auch hier sind weitere Beweise erforderlich. Ein typisches Beispiel ist die phylogenomische Analyse eines Komplexes von Gehyra-Gecko-Eidechsen aus Nordaustralien, die neun Evolutionslinien abgrenzte, wobei vier bereits benannte Arten phylogenetisch zwischen ihnen eingebettet waren (Moritz et al., 2018; Abbildung 2). Nachfolgende Hinweise auf geografische Überschneidungen (unter Beibehaltung der phylogenetischen Trennung) und subtile morphologische Unterschiede unterstützten die Benennung der vier phylogenetisch divergentesten Linien (Doughty et al., 2018). Umgekehrt hatten die enger verwandten (aber statistisch trennbaren) Linien eher benachbarte als überlappende Verteilungen, waren aber morphologisch nicht diagnostizierbar und wurden dementsprechend als eine einzige Art (G. nana sensu stricto) beibehalten, wenn auch mit mehreren ESUs, bis weitere Untersuchungen anstehen (Abbildung 2). Nachfolgende Genomscans von Proben über die Grenzen dieser ESUs hinweg zeigten, dass zumindest einige einen erheblichen genetischen Austausch aufweisen, Validierung des ursprünglichen, konservativer Ansatz zur Erkennung von Arten.In anderen Fällen können pflanzengenomische Studien, selbst durch einfache Ordinierung von SNPs, starke und unerwartete Divergenzen zwischen Populationsproben aufdecken, was auf das Vorhandensein kryptischer Taxa hindeutet, die dann auf diagnostische phänotypische Merkmale analysiert werden können. Zum Beispiel zeigten SNP-basierte Beweise für genomische Divergenz zwischen Populationen und ökotypische Unterschiede in Eukalyptus Salubris zwei verschiedene molekulare Linien, die ihre Unterscheidungskraft in einem Gebiet geografischer Überlappung beibehielten, wobei die ökotypische Anpassung als wichtiger Faktor bei der Minimierung des Genflusses zwischen den Linien angesehen wurde. In: Steane et al., 2015; Abbildung 3). In einem anderen Fall, in der australischen Pelargonie, genomische Analyse, kombiniert mit morphologischen und reproduktiven Studien, hat zu signifikanten Veränderungen in der Taxonomie geführt, sowohl Spaltung als auch Klumpung von Taxa, insbesondere dort, wo zuvor verwendete morphologische Merkmale labil und nicht mit der Phylogenie übereinstimmen (Nicotra et al., 2016).
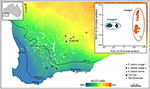
Abbildung 3. Die genomische Analyse unter Verwendung von SNP-Daten identifizierte kryptische Abstammungslinien im weit verbreiteten Eukalyptus Salubris. Die Lage der Probenpopulationen jeder Linie wird über einen Gradienten des Trockenheitsindex im Südwesten Westaustraliens angezeigt. Der PCoA, der auf binären genetischen Distanzen basiert, die von 16.122 DArTseq-Markern abgeleitet wurden, zeigt zwei verschiedene Abstammungslinien. Angepasst von Steane et al. (2015).
Die obigen Fälle veranschaulichen den progressiven Hypothesentestansatz, der in der integrativen Taxonomie verwendet wurde (Padial et al., 2010; Wiegand et al., 2011). Darüber hinaus verwenden diese Fälle eine Form der Genomreduktionssequenzierung, um qualitativ hochwertige Daten von 1000 unabhängigen Genen zu erhalten, was ein viel größeres Vertrauen in die Ergebnisse gibt, als dies bei Verwendung nur von Plastidenloci oder einigen Kerngenen der Fall ist. Ein Beispiel dafür, wie die Anwendung einer integrativen Taxonomie die Wahrnehmung von Vielfalt dramatisch verändern kann, ist eine kombinierte genetische, akustische und morphologische Untersuchung von Fröschen aus Madagaskar, die die bekannte Artenvielfalt fast verdoppelt hat (Vieites et al., 2009). Diese Autoren schlugen eine Reihe von Begriffen vor, die den Stand der taxonomischen Auflösung und Kongruenz widerspiegeln: Bestätigte Kandidatenarten für genetisch und phänotypisch divergierende, aber noch nicht benannte Taxa; Unbestätigte Kandidatenarten für genetisch divergierende Taxa, bei denen Morphologie und Akustik noch nicht untersucht worden waren; und tief konspezifische Abstammungslinien für Entitäten, von denen bekannt ist, dass sie genetisch divergent und dennoch phänotypisch kryptisch sind. Genomische Analysen müssen noch auf diese Taxa angewendet werden, würden aber sicherlich Entscheidungen über Artgenossen vs. Kandidatenarten treffen.
Genomische Ansätze werden, selbst wenn sie konservativ angewendet werden, zweifellos zu einer weiteren Spaltung morphologisch konservativer Taxa führen. Umgekehrt gibt es jedoch klare Beispiele, bei denen dieselben Ansätze überspaltete oder falsch diagnostizierte Arten aufdecken (Nicotra et al., 2016). Natürlich gibt es bereits viele molekulare Studien zu Pflanzen- und Tierartenkomplexen, die auf kleineren Datensätzen basieren (z. Mikrosatelliten, mtDNA und cpDNA), die identifiziert haben, wo Arten falsch diagnostiziert wurden, oder wo mehrere Arten taxonomisch umschrieben wurden, aber jetzt als eine einzige Art betrachtet werden. Zum Beispiel Rabosky et al. (2014) verwendeten mitochondriale und nukleare Genphylogenien, um den Scincid Ctenotus inornatus-Komplex zu überarbeiten, was zu einer Nettoreduktion der Arten und einer Neuausrichtung der Artengrenzen führte; Die zuvor angewendeten morphologischen Merkmale waren zwar für Feldbiologen geeignet, jedoch hoch labil und entsprachen den genetisch diagnostizierten Artengrenzen weitgehend nicht. In Pflanzen finden sich einige der bekanntesten Beispiele in den Orchideen, wie der europäischen Orchideengattung Ophrys und der australischen Orchideengattung Caladenia, wo molekulare Studien deutlich weniger Entitäten anzeigen, die als Arten erkannt würden, als solche, die auf morphologischen Merkmalen und pseudokopulatorischen Bestäubungssyndromen beruhen (siehe Devey et al., 2008; Swarts et al., 2014).
Daten auf Genomskala werden, wenn sie konservativ und idealerweise mit parallelen Analysen von Phänotypen interpretiert werden, weiterhin zu taxonomischen Veränderungen führen, insbesondere bei morphologisch konservativen Taxa (Fišer et al., 2018). Im Gegensatz zu Bedenken einiger (Garnett und Christidis, 2017) sind wir jedoch der Ansicht, dass dies das richtige Streben nach evolutionärer Systematik ist, auch wenn die Ergebnisse vorübergehend ungünstig sind.
Genomik und intraspezifische Erhaltungseinheiten
Obwohl weniger umstritten als die taxonomische Abgrenzung und die Debatte auf Artenebene, aber in vielerlei Hinsicht genauso komplex, war die bedeutende Diskussion über die Definition und Abgrenzung von Erhaltungseinheiten als wichtige Elemente der intraspezifischen Vielfalt, die bei Erhaltungsmaßnahmen berücksichtigt werden müssen (Ryder, 1986; Waples, 1991; Dizon et al., 1992; Moritz, 1994; Vogler und de Salle, 1994; Crandall et al., 2000; Fraser und Bernatchez, 2001; Avise, 2004; Funk et al., 2012) (Abbildung 4). Angesichts des vorsichtigen Ansatzes, Arten anhand genomischer Beweise zu erkennen, den wir oben befürworten (und siehe auch Oliver et al., 2015; Singhal et al., 2018) ist es unvermeidlich, dass einige taxonomische Arten noch lange isolierte Populationen enthalten. Konzepte wie evolutionär signifikante Einheiten (ESUs) oder bezeichnbare Einheiten (DUs) wurden vorgeschlagen, um die bestehende Taxonomie zu ergänzen (Ryder, 1986; Waples, 1991; Moritz, 1994; Mee et al., 2015). Wie bei Arten variieren Interpretationen und Kriterien, aber die gemeinsame Absicht ist es, wichtige Elemente der intraspezifischen Vielfalt für Erhaltungsmaßnahmen zu erkennen (Funk et al., 2012). Moritz (1994, 2002) befürwortete einen vereinfachten, aber praktischen molekularen Ansatz zur Identifizierung sich unabhängig entwickelnder Segmente innerhalb von Arten, der „historischen“ Achse der Vielfalt; während andere mehr Wert auf die „adaptive“ Achse legen, um funktionelle Vielfalt einzubeziehen (Ryder, 1986; Crandall et al., 2000; Fraser und Bernatchez, 2001; Waples et al., 2001; Mee et al., 2015) (Abbildung 4). Unabhängig von den relativen Vorteilen dieser Alternativen ist es klar, dass die technischen und analytischen Werkzeuge der Populationsgenomik nun beide Komponenten der intraspezifischen Diversität robust identifizieren können (Funk et al., 2012; Hoban et al., 2013; Whitlock, 2014; Yang und Rannala, 2014).
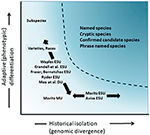
Abbildung 4. Trennung der genetischen Vielfalt in zwei Komponenten: adaptive Variation, die durch natürliche Selektion entsteht und durch Analyse von Phänotypen und historische Isolation oder neutrale Divergenz durch genomische Divergenz bewertet wird. Der dunkler schattierte Bereich über der Kurve zeigt Bedingungen an, unter denen Populationen nach den meisten Konzepten wahrscheinlich als separate Arten betrachtet werden. Verschiedene Konzepte von Erhaltungseinheiten und infraspezifische taxonomische Kategorien werden in Bezug auf die beiden Achsen der genetischen Vielfalt gezeigt (nach Moritz, 2002).
Dies führt zu der Frage: Warum nicht einfach ESUs als Unterart benennen? Unterarten wurden traditionell sowohl von Botanikern als auch von Zoologen als phänotypisch unterschiedliche, allopatrische Populationen definiert und angesehen, die sich an geografischen Grenzen ineinander übergehen können (Mayr, 1942; Grant, 1981). Als solche spiegeln sie möglicherweise adaptive Divergenz wider, ohne notwendigerweise historisch isoliert worden zu sein. Unterartbezeichnungen sind in Pflanzen weit verbreitet, wobei geografische und ökologische Unterschiede die primären Mittel sind, mit denen sie unterschieden werden (Hamilton und Reichard, 1992). Soweit Unterarten lokale Anpassung manifestieren, mit oder ohne laufenden Genfluss, Wir schlagen vor, dass phänotypisch definierte Unterarten weiterhin in der Erhaltung anerkannt werden sollten, und nicht mit ESUs verschmolzen, zumindest nicht ESUs wie definiert, um historisch isolierte Populationen darzustellen.In der gleichen Weise, wie die Populationsgenomik eine weitaus größere Präzision bei der Identifizierung von ESUs bietet, indem sowohl neutrale als auch adaptive Loci verwendet werden, bietet sie auch ein Mittel zur Verwendung einer großen Anzahl neutraler Loci, um Managementeinheiten (MUs) leichter als demografisch unabhängige Einheiten abzugrenzen, die durch einen eingeschränkten Genfluss gekennzeichnet sind (Funk et al., 2012). Während wir uns hier hauptsächlich auf ESU und analoge Einheiten wie DUs konzentrieren, die als die wichtigsten intraspezifischen Einheiten für die Erhaltung anerkannt werden können und in einigen Gerichtsbarkeiten zum Schutz zugelassen wurden, wurden auch MUs identifiziert, die demografisch unabhängige Gruppen von Populationen darstellen (siehe Palsbøll et al., 2007), die verwaltet werden können, um die größere ESU beizubehalten (Moritz, 1994; Funk et al., 2012). Managementeinheiten können für die Überwachung und das Management von Populationen von Arten wichtig sein und wurden auch als „Bestände“ bezeichnet, was insbesondere für das Fischereimanagement relevant ist (Avise, 2004; Laikre et al., 2005). Die Definition von MUs durch Moritz (1994) als „Populationen mit signifikanter Divergenz der Allelfrequenzen an nuklearen oder mitochondrialen Loci, unabhängig von der phylogenetischen Unterscheidungskraft der Allele“ wird allgemein akzeptiert, obwohl Palsbøll et al. (2007), demografische Konnektivität ist der Schlüsselfaktor, nicht das Niveau des historischen Genflusses.
Die Abgrenzung von Erhaltungseinheiten als wichtige Elemente intraspezifischer Diversität hat auch erhebliche Auswirkungen auf die genetische Rettung kleiner Inzuchtpopulationen durch Wiederherstellung des Genflusses oder durch Augmentation, und es wird immer deutlicher, dass die genetische Rettung breiter betrachtet werden muss, wenn ein verstärktes Aussterben der Population abgewendet werden soll (Love Stowell et al., 2017; Ralls et al., 2018). Moritz (1999) schlug vor, dass das Mischen von MUs, aber nicht von ESUs als geeignete Strategie für die genetische Rettung angesehen werden könnte. Wie von Frankham et al. (2012) Die Vermischung taxonomisch anerkannter Arten würde von Managern im Allgemeinen nicht als akzeptabel angesehen, In diesem Fall könnte eine taxonomische Überspaltung die Möglichkeiten zur Rettung kleiner Inzuchtpopulationen einschränken. Wir schlagen vor, dass ein strengerer Ansatz zur Abgrenzung intraspezifischer Einheiten wie MUs dieses Problem lindern könnte, Es ist auch klar, dass die genomische Schätzung der Migrationsraten und der adaptiven Divergenz das Potenzial hat, viel robustere Leitlinien nicht nur für die Definition von Artengrenzen und intraspezifischen Variationen zu liefern, aber bei der Gestaltung genetischer Rettungsstrategien.
Phylogenomik und Konservierung
Genomik in Kombination mit phylogenetischen Methoden kann Beziehungen und evolutionäre Entfernungen zwischen Taxa sowie die Abgrenzung von Arten leicht auflösen. Dies wird am besten mit Artenbaummethoden anstelle der Verkettung erreicht, da erstere die Heterogenität des Genbaums erklären und eine Überinflation der Spitzenlängen aufgrund von Ahnenpolymorphismus vermeiden (Edwards et al., 2016; Ogilvie et al., 2016). Während eine große Anzahl von Loci oft nicht wesentlich ist, verbessert die Einbeziehung von mehr Loci oft die Auflösung sowohl von Beziehungen als auch von Verzweigungslängen (z. B. Blom et al., 2016). Die Bedeutung für die Erhaltung ist zweifach. Erstens ermöglichen phylogenetische Bewertungen die Berücksichtigung der Unterscheidungskraft von Arten oder ESUs, nicht nur, ob sie beschrieben werden. Das EDGE-Programm (Isaac et al., 2007) befürwortet die Gewichtung von Arten für den Naturschutz auf der Grundlage der einzigartigen Evolutionsgeschichte, die sie repräsentieren, zusätzlich zu den üblichen IUCN-Kriterien. Zweitens können die Astlängen selbst Biodiversitätswerte darstellen, unabhängig von der Taxonomie (Faith, 1992), die sich bei der Priorisierung von Naturschutzgebieten als wertvoll erwiesen hat (Laity et al., 2015; Rosauer et al., 2016, 2018). In diesem Sinne, Die Annahme phylogenetischer Metriken unter Verwendung von Maßnahmen wie phylogenetische Vielfalt und phylogenetischer Endemismus ermöglicht es, Bereiche mit hoher evolutionärer Vielfalt hervorzuheben, auf die der Schutz abzielen kann, und in jedem Planungsprozess für einen verbesserten Erhalt der biologischen Vielfalt hohe Priorität erhalten. Dieser Ansatz kann den Schutz der genetischen Vielfalt und evolutionärer Prozesse für verschiedene Organismengruppen auf verschiedenen geografischen Skalen verbessern (Laity et al., 2015) Entscheidungen über Bereiche zu priorisieren robuster zu den Launen, wie genomische Beweise auf die Anerkennung von Arten oder intra-spezifischen Einheiten übersetzt werden.
The Real World—Legislative Definitions
Die politischen und rechtlichen Rahmenbedingungen für den Erhalt der biologischen Vielfalt reichen von internationalen Konventionen und Strategien über nationale Strategien und Gesetze bis hin zu staatlichen oder regionalen Gesetzen und Strategien innerhalb von Ländern. Zu den wichtigsten internationalen Instrumenten zur Erhaltung der biologischen Vielfalt gehören das Übereinkommen über die biologische Vielfalt, das durch das Übereinkommen der Vertragsparteien (COP) umgesetzt wird, das Welterbekonvention, die Globale Biodiversitätsstrategie, der Montreal-Prozess für die Erhaltung der Wälder, das Ramsar-Übereinkommen über Feuchtgebiete, das Übereinkommen über wandernde Arten, das Übereinkommen über den internationalen Handel mit gefährdeten Arten (CITES), die Rote Liste der Internationalen Union für die Erhaltung der Natur (IUCN) über bedrohte Arten und das Rahmenübereinkommen der Vereinten Nationen über Klimaänderungen. Alle diese Chartas verkörpern das Prinzip der Erhaltung von Arten und genetischer Vielfalt als grundlegendes Ziel der Erhaltung der biologischen Vielfalt, wobei Arten die Einheit sind, die allgemein für den Schutz definiert ist. Die Definitionen von Arten in Gesetzen und internationalen Richtlinien sind jedoch äußerst unterschiedlich. Sie beinhalten die taxonomische Erkennung von Arten und intraspezifischen Taxa, können aber auch genetische / evolutionäre Konzepte enthalten, die häufig unter die rechtliche Definition einer „Art“ fallen (Tabelle 1).

Tabelle 1. Weltgesetzgebung und Richtlinien, die „Arten“ für die Auflistung und den Schutz definieren.
Angesichts des breiten Spektrums an infra-spezifischen Ebenen, die geschützt werden können (Tabelle 1), sollten die Gesetzgebung und die damit verbundenen Listing-Prozesse der Länder die Erhaltung der genetischen Vielfalt erleichtern. Zum Beispiel kann die Definition einer „Art“, wenn man die Gesetzgebung auf der ganzen Welt betrachtet (Tabelle 1), Folgendes umfassen: arten, Unterarten, Sorten und geografisch und / oder genetisch unterschiedliche Populationen, während es nach südafrikanischer Gesetzgebung auch Sorten, geografische Rassen, Stämme und Hybriden umfassen kann. Daher umfasst eine „Art“ nach den meisten Gesetzen weltweit Einheiten, die verschiedene infraspezifische Ebenen widerspiegeln, die alle Teil einer breiteren Artenverteilung sind und in unterschiedlichem Maße das Populations-Arten-Kontinuum widerspiegeln. Dies entspricht der unvermeidlichen Schwierigkeit, mit der Taxonomen, Evolutionsbiologen und Naturschutzgenetiker konfrontiert sind, um einen konsistenteren und einheitlicheren Weg zu finden, Einheiten innerhalb dieses Kontinuums zu spezifizieren.Einige argumentieren, dass Bedenken hinsichtlich der politischen Implikationen von Arten- und infraspezifischer Unsicherheit fehl am Platz sind (Hey et al., 2003), aber wir sind immer noch der Ansicht, dass die Gesetzgebung zwar einen Mechanismus zum Schutz der genetischen Vielfalt auf verschiedenen Ebenen bieten kann, die Biologen jedoch mehr Klarheit und Konsistenz darüber schaffen müssen, wie diese Ebenen von Arten bis zu einzelnen Populationen abgegrenzt werden sollten. Gegenwärtig stehen Manager und Gesetzgeber vor einer verwirrenden Reihe von Artenkonzepten und infraspezifischen Einheiten, die von Wissenschaftlern bereitgestellt werden, wenn sie Einheiten über das Populations-Arten-Kontinuum hinweg abgrenzen. Zum Beispiel betrachten alle Jurisdiktionen Arten, die von Taxonomen anerkannt werden, als Schlüsseleinheiten für die Erhaltung (Tabelle 1), aber es gibt immer noch eine breite Debatte unter Taxonomen über Artenkonzepte, und wie Arten in der Praxis diagnostiziert werden, variiert innerhalb und zwischen Gruppen. Dies hat kürzlich zu einer Forderung nach einem stärker regulierten Ansatz für die Taxonomie geführt (Garnett und Christidis, 2017), auf die andere gekontert haben, dass die Regulierung der Taxonomie nicht die Antwort ist (Thomson et al., 2018). Obwohl wir einigen der von Thomson et al. (2018) wir schlagen vor, dass es ziemlich naiv ist vorzuschlagen, dass die Gesetzgebung selbst geändert werden muss, um explizit auf das spezifische Taxonkonzept zu verweisen, das durch einen Namen impliziert wird. Diese und frühere Debatten unterstreichen die Notwendigkeit, dass Evolutionsbiologen und Taxonomen die Führung übernehmen, um Klarheit (Transparenz) darüber zu gewährleisten, wie sie genomische und andere Beweise in die Abgrenzung von Arten oder intraspezifischen Einheiten übersetzen und wie sich diese Taxa auf die einschlägigen Rechtsvorschriften beziehen Erhaltung und Schutz.
Übersetzung in das Management
Wie oben diskutiert, erkennen alle Gesetze und Richtlinien die Artenebene als grundlegende Einheit der Erhaltung an, sehen aber auch die Anerkennung verschiedener infraspezifischer Einheiten vor. Viele haben auch eine implizite Anforderung an die formale Benennung und taxonomische Beschreibung von Arten, wenn sie für die Priorisierung der Erhaltung und den formellen Schutz in Betracht gezogen werden. Beispielsweise wird nach den Richtlinien der IUCN zur Roten Liste davon abgeraten, unbenannte Arten aufzulisten, und es besteht die Anforderung, dass die neue Artbeschreibung innerhalb von vier Jahren nach Aufnahme der Art in die Rote Liste der IUCN veröffentlicht oder entfernt wird. Dies unterstreicht die Bedeutung der taxonomischen Benennung und Beschreibung von Entitäten sowohl auf arten- als auch auf infraspezifischer Ebene, die begrenzten taxonomischen Informationen für viele Pflanzen- und Tiergruppen (Mace, 2004; Oliver und Lee, 2010; Wilson, 2017) und die Notwendigkeit, die taxonomischen Anstrengungen zu beschleunigen, insbesondere in biodiversitätsreichen Gebieten (Riedel et al., 2013; Wege et al., 2015; Wilson, 2017).Taxonomisch benannte Arten und intraspezifische taxonomische Kategorien wie Unterarten und Varietäten bilden im Allgemeinen die Grundeinheiten für die formale Auflistung, die Priorisierung der Erhaltung und den Schutz, in einigen Gerichtsbarkeiten werden jedoch andere Kategorien intraspezifischer Erhaltungseinheiten formell anerkannt. Diese werden im Allgemeinen anhand kombinierter genomischer und phänotypischer Daten identifiziert. Zum Beispiel erkennt der USA Endangered Species Act verschiedene Populationssegmente (DPSs) an, die weitgehend auf ESUs basieren (Waples, 1991), während der Canadian Species at Risk Act DUs anerkennt. Eine operative Interpretation von „unterschiedlichen Populationen“ im Rahmen des kanadischen Species at Risk Act, DUs zielt darauf ab, diskrete Populationssätze zu identifizieren, die wichtige Bestandteile des evolutionären Erbes von Arten darstellen und die wahrscheinlich nicht durch natürliche Dispersion ersetzt werden (Mee et al., 2015).
Sowohl DUs als auch DPSs überschneiden sich konzeptionell mit ESUs (Waples et al., 2013) und wurden am häufigsten bei nordamerikanischen Fischen angewendet, einschließlich Nordwest-USA-Salmoniden (Waples, 1995) und kanadischen Coregoniden (Mee et al., 2015) Interessanterweise gelten DPSs in den USA nur für Wirbeltiere, während DUs in Kanada auf jeden Organismus angewendet werden kann, der unter seinem Gesetz aufgeführt werden kann (siehe Waples et al., 2013), wobei DUs derzeit für Arthropoden, Weichtiere und Pflanzen aufgeführt ist. In Australien, wie Kanada, können verschiedene Populationen für jeden Organismus aufgeführt werden, der unter dem Environment Protection and Biodiversity Conservation Act 1999 (EPBC Act) anerkannt ist, obwohl sie keine speziell benannte Kategorie sind, wie DUSs und DPSs. Bisher wurden sie jedoch nur für Wirbeltiere erkannt. In den USA sind 127 DPSS für Wirbeltiere zur Erhaltung aufgeführt (https://ecos.fws.gov/ecp/); In Kanada wurden 70 DUs für alle Organismen aufgeführt (http://www.registrelep-sararegistry.gc.ca/species/schedules_e.cfm?id=1); und in Australien wurden Populationen von 7 Wirbeltierarten, die Säugetiere, Fische und Vögel abdecken, gemäß dem EPBC-Gesetz als spezifisch eingestuft Erhaltungszustand (http://www.environment.gov.au/cgi-bin/sprat/public/publicthreatenedlist.pl). Während Bewertungen dieser Populationen in den USA und Kanada im Allgemeinen genetische Informationen verwenden (Waples et al., 2013) basieren sie in Australien normalerweise auf geografischer und jurisdiktionaler Identifizierung, obwohl sie in einigen dieser Fälle durch die Analyse der genetischen Differenzierung für Säugetiere unterstützt wurden (Firestone et al., 1999; Armstrong, 2006) und Fisch (Colgan und Paxton, 1997; Ahonen et al., 2009).Die Auflistung von infraspezifischen Schutzeinheiten für Pflanzen und Wirbellose in Kanada (DUs), jedoch nicht in den USA oder Australien, hebt ein weiteres Problem hervor, das nicht speziell mit der Abgrenzung von Schutzeinheiten zusammenhängt, sondern im weiteren Sinne mit erheblichen Inkonsistenzen bei der Priorisierung der Erhaltung der biologischen Vielfalt. ikonische Wirbeltiere wie Vögel, Säugetiere und Fische stehen in vielen Ländern im Mittelpunkt, andere Organismen jedoch nicht. Diese bevorzugte Auflistung von Populationen bestimmter Organismen spiegelt eine Reihe von Fragen wider, die weniger mit der Wissenschaft der Abgrenzung von Erhaltungseinheiten als vielmehr mit der Auflistung von Populationen zu tun haben, die auf dem öffentlichen Bewusstsein, der politischen Zweckmäßigkeit und der charismatischen Natur des Organismus beruhen aufgeführt werden (siehe Waples et al., 2013).Während in Australien einige Populationen bekannter Säugetiere, Vögel und Fische unter dem EPBC Act geschützt sind, gibt es jetzt eine bedeutende Menge an Informationen, die auf molekularen Studien an Reptilien, Wirbellosen und Pflanzen basieren, bei denen Populationen als ESUs oder MUS erkannt werden können, und viele von ihnen sind aufgrund ihres engen geografischen Verbreitungsgebiets, ihrer geringen Populationsgröße, ihrer Rückgangsrate und ihrer Anfälligkeit für bedrohliche Prozesse von Bedeutung für den Naturschutz. Markante Beispiele für stark strukturierte Populationen und stark divergierende Abstammungslinien innerhalb von Arten, die leicht den Anforderungen einer ESU entsprechen würden, finden sich beispielsweise in einer Reihe von Wirbeltieren mit geringer Verbreitung aus den Tropen (Abbildung 2) und anderswo. In ähnlicher Weise unterstützen bei australischen Wirbellosen wie Amphipoden und Tausendfüßlern signifikante genetische Strukturen und phylogeografische Muster die Abgrenzung diskreter Populationscluster als ESUs, die geografisch unterschiedlich sind, mit engen geografischen Bereichen, die auf bestimmte Lebensräume beschränkt sind und nur von wenigen Populationen vertreten werden (Finston et al., 2007; Murphy et al., 2013; Nistelberger et al., 2014a). Oft sind diese Kurzstreckenendemiten Taxa von Naturschutzbedenken (Harvey et al., 2011) und sind co-lokalisiert in Endemismus-Hotspots (z.B. Oliver et al., 2017), die selbst hohe Prioritäten für die Multi-Taxon-Erhaltung darstellen (Rosauer et al., 2018), wie die Kimberley-Region und die Stirling Range im Südwesten Westaustraliens.In australischen Pflanzen gibt es auch eine signifikante Anzahl von populationsgenetischen und phylogeographischen Studien, die verschiedene Populationsgruppen innerhalb einer Reihe von Arten aus verschiedenen Gattungen identifizieren, die disjunkte und historisch isolierte Populationssysteme, geologische und edaphische Komplexitäten widerspiegeln und kontrastierende Lebensräume in Bezug auf Vegetation und Klima besetzen (siehe Broadhurst et al., 2017). Genetisch unterschiedliche Populationen zeigen sich besonders bei Arten aus Südwestaustralien (Coates, 2000; Byrne et al., 2014) mit einer Reihe von Studien, die eine signifikante genetische Struktur und stark divergierende Linien in einer Reihe von Pflanzengattungen und -familien hervorheben, darunter: Akazie (Mimosaceae; Coates, 1988), Atriplex (Amaranthaceae; Shepherd et al., 2015), Banksia (Proteaceae; Coates et al., 2015), Lambertia (Proteaceae; Byrne et al., 2002), Hakea (Proteaceae; Sampson et al., 2014), Eremophila (Scrophulariaceae; Llorens et al., 2015), Pultanaea (Fabaceae; Millar und Byrne, 2013) und Calothamnus (Myrtaceae; Nistelberger et al., 2014b). In vielen Fällen werden diese Populationen von den staatlichen Regierungsbehörden in der Naturschutzplanung anerkannt und in Umweltverträglichkeitsprüfungen berücksichtigt (siehe Shepherd et al., 2015), obwohl sie nicht gesetzlich zum Schutz aufgeführt sind. Wir erkennen jedoch an, dass die formale Anerkennung dieser Einheiten ihre Erhaltung langfristig verbessern würde.
Diese australischen Beispiele unterstreichen die zunehmende Notwendigkeit, diese Schicht genetischer Vielfalt unterhalb der Artenebene zu erkennen. Die Herausforderung besteht darin, sicherzustellen, dass stark strukturierte Populationen und stark divergierende Abstammungslinien, die für den Naturschutz von Belang sind, angesichts konkurrierender Anforderungen an die Aufmerksamkeit für den Naturschutz geschützt werden. Abgesehen von ikonischen Arten ist diese Herausforderung mit Ausnahme Kanadas auch weltweit offensichtlich. Das Versäumnis, klar abgegrenzte Naturschutzeinheiten zu schützen, wird wahrscheinlich das Risiko eines kryptischen Aussterbens und des Verlusts einer signifikanten genetischen Vielfalt erhöhen, was in vielen Fällen Abstammungslinien und historisch isolierte Populationen betreffen kann, die seit Millionen von Jahren in Arten bestehen (Moritz, 2002).
Schlussfolgerungen und Empfehlungen
Die Auflistung von Arten und verschiedenen infraspezifischen Kategorien im Rahmen der Gesetzgebung zu bedrohten Arten oder im Rahmen internationaler Organisationen wie der IUCN zeigt ein Engagement für den Schutz der genetischen Vielfalt im gesamten Artenpopulationskontinuum. Die derzeitigen Ansätze zur Erhaltung der biologischen Vielfalt konzentrieren sich jedoch weitgehend auf das Artenende dieses Kontinuums, wobei der genetischen Vielfalt und der infraspezifischen Variation weniger Aufmerksamkeit geschenkt wird. Dieser eingeschränktere Fokus auf die genetische Variation innerhalb der Arten legt nahe, dass Naturschutzmanager zwar zunehmend den Wert der expliziten Berücksichtigung genetischer Informationen bei Managementmaßnahmen und Priorisierungen erkennen, ihnen jedoch häufig umsetzbare Informationen von Biodiversitätswissenschaftlern fehlen (Waples et al., 2008; Laikre et al., 2010; Cook und Sgrò, 2017; Taylor et al., 2017). Diese Kluft zwischen Wissenschaft und Naturschutzpraxis behindert einen effektiveren globalen Ansatz zur Erhaltung und zum Management der genetischen Vielfalt.
Wir schlagen vor, dass der Mangel an Klarheit von Biodiversitätswissenschaftlern über Naturschutzeinheiten weitgehend auf Inkonsistenzen auf zwei Ebenen beruht. Die erste betrifft die Konzepte mehrerer Arten und die verschiedenen diagnostischen Methoden zur Abgrenzung von Arten, die zu Inkonsistenzen zwischen taxonomischen Gruppen führen. Die zweite bezieht sich auf einen Mangel an Konsistenz und Übereinstimmung von Taxonomen, Evolutionsbiologen und Naturschutzbiologen darüber, wie infraspezifische Taxa und andere Entitäten wie ESUs, DUs und MUs zu behandeln sind.Während wir der Ansicht sind, dass die Gesetzgebung in Bezug auf die Definition von Taxa (oder „Arten“) oft bereits ausreichend flexibel ist, argumentieren wir, dass es notwendig ist, das Kontinuum der Populationsarten systematisch anzugehen und Einheiten für die Erhaltung klarer zu definieren, wobei Konzepte wie ESU und deren Beziehung zu relevanten Rechtsvorschriften stärker anerkannt werden. Wir betonen, dass diese Frage angesichts der Fähigkeit der Genomik, Differenzierung auf immer feineren Skalen zu erkennen, und ihrer zunehmenden Verwendung zur Abgrenzung von Arten und infraspezifischen Einheiten zweifellos kritischer werden wird.
Aus der vorangegangenen Analyse bieten wir folgende Empfehlungen an:
(i) Genomische Daten sollten bei der Abgrenzung von Arten konservativ interpretiert werden, wobei ein integrativer taxonomischer Ansatz verwendet wird, der typischerweise Beweise enthält, die über die genetischen Daten hinausgehen.
(ii) Wenn Genetiker mehrere Abstammungslinien oder unterschiedliche Populationen innerhalb einer Art identifizieren und diese keine Erhöhung auf Artenebene rechtfertigen und nicht taxonomisch anerkannt sind, dass es stärkere Impulse und einen einheitlicheren Ansatz zur Identifizierung eindeutig begründeter intraspezifischer Einheiten für die praktische Bewertung des Erhaltungszustands oder von Folgenabschätzungen gibt und dass diese ausdrücklich als Einheiten für die Erhaltung im Rahmen der für diese Gerichtsbarkeit relevanten Rechtsvorschriften anerkannt werden.
(iii)Die IUCN sollte über ihre Fachgruppe für Erhaltungsgenetik und ihre regionalen Untergruppen erwägen, Richtlinien für die Anerkennung und Kennzeichnung intraspezifischer Einheiten zu entwickeln, die den besten wissenschaftlichen Praktiken entsprechen.
(iv) Die differenziertere Sichtweise, die sich aus der evolutionären Genomik von Arten und dem Kontinuum von Populationen zu Arten ergibt, sollte von Wissenschaftlern den Naturschutzmanagern und politischen Entscheidungsträgern wirksamer vermittelt werden.
Im weiteren Sinne wiederholen wir Empfehlungen, die kürzlich in einer Reihe von Reviews (Frankham, 2010; Santamaría und Méndez, 2012; Hoban et al., 2013; Moritz und Potter, 2013; Laien et al., 2015; Pierson et al., 2016; Cook und Sgrò, 2017; Taylor et al., 2017), die die Notwendigkeit für Wissenschaftler hervorheben, die Kommunikation zu verbessern und das Bewusstsein mit politischen Entscheidungsträgern, Regierungsbehörden, Nichtregierungsorganisationen und Praktikern zu schärfen, dass Ergebnisse aus molekularer Systematik, molekularer Taxonomie, Phylogeographie und populationsgenetischer Forschung kritische Informationen über die Erhaltung der genetischen Vielfalt und das Management evolutionärer Prozesse liefern können.
Autorenbeiträge
DC, MB und CM haben alle zur Vorbereitung und Überarbeitung des Manuskripts beigetragen.
Erklärung zum Interessenkonflikt
Die Autoren erklären, dass die Forschung in Abwesenheit von kommerziellen oder finanziellen Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Danksagung
Dieses Projekt wurde vom Australian Research Council (ARC) Grant No. LP120200063), die Australian National University und das Western Australian Department of Biodiversity, Conservation und Attraktionen.
Afonso Silva, A. C., Bragg, J. G., Potter, S., Fernandes, C., Coelho, M. M. und Moritz, C. (2017a). Tropenspezialist versus Klimageneralist: Diversifizierung und demografische Geschichte von Schwesterarten von Carlia Skinks aus Nordwestaustralien. Mol. Ecol. 26:4045–4058. doi: 10.1111/mec.14185
CrossRef Volltext / Google Scholar
Afonso Silva, A. C., Santos, N., Ogilvie, H. A. und Moritz, C. (2017b). Validierung und Beschreibung von zwei neuen North-Western Australian Rainbow Skinks mit Multispezies Koaleszenzmethoden und Morphologie. PeerJ 5:e3724. doi: 10.7717/peerj.3724
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Ahonen, H., Harcourt, RG und Stow, AJ (2009). Nukleare und mitochondriale DNA zeigt die Isolierung gefährdeter Populationen von Ammenhaien (Carcharias taurus). Mol. Ecol. 18, 4409–4421. Ursprungsbezeichnung: 10.1111/j.1365-294X.2009.04377.x
PubMed Zusammenfassung | CrossRef Volltext / Google Scholar
Armstrong, KN (2006). Phylogeographische Struktur in Rhinonicteris aurantia (Chiroptera: Hipposideridae): Implikationen für die Erhaltung. Acta Chiropterologica 8, 63-81. doi: 10.3161/1733-5329(2006)82.0.CO
CrossRef Volltext | Google Scholar
Arnold, M. L., und Kunte, K. (2017). Adaptiver genetischer Austausch: eine verschlungene Geschichte der Beimischung und evolutionären Innovation. In: Trends Ecol. In: Evol. 32, 601–611. doi: 10.1016/j.Baum.2017.05.007
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Avise, J. C. (2004). Molekulare Marker, Naturgeschichte und Evolution. 2. Aufl. Sunderland, MA; Oxford: Oxford University Press.
Google Scholar
Avise, J. C., Arnold, J., Ball, R. M., Bermingham, E., Lamb, T., Neigel, J. E., et al. (1987). Intraspezifische Phylogeographie: die mitochondriale DNA-Brücke zwischen Populationsgenetik und Systematik. Ann. Rev. Ecol. Sys. 18, 489–522. doi: 10.1146/annurev.es.18.110187.002421
CrossRef Volltext | Google Scholar
Bickford, D., Lohman, D. J., Sodhi, N. S., Ng, P. K., Meier, R., Winker, K., et al. (2007). Kryptische Arten als Fenster zur Vielfalt und zum Schutz. In: Trends Ecol. In: Evol. 22, 148–155. doi: 10.1016/j.Baum.2006.11.004
PubMed Abstract / CrossRef Full Text / Google Scholar
Blom, M. P. K., Bragg, J. G., Potter, S. und Moritz, C. (2016). Berücksichtigung der Unsicherheit bei der Schätzung von Genbäumen: Zusammenfassung – koaleszierende Artenbauminferenz in einer herausfordernden Studie australischer Eidechsen. Syst. Biol. 66, 352–366. doi: 10.1093/sysbio/syw089
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Broadhurst, L., Breed, M., Lowe, A., Bragg, J., Catullo, R., Coates, DJ, et al. (2017). Genetische Vielfalt und Struktur der australischen Flora. Taucher. Distrib. 23, 41–52. zertifikate: 10.1111/ddi.12505
CrossRef Volltext / Google Scholar
Byrne, M., Coates, D. J., Forest, F., Hopper, S. D., Krauss, SL, Sniderman, JMK, et al. (2014). „Eine vielfältige Flora – Arten und genetische Beziehungen“, in Plant Life on the Sandplains in Southwest Australia, einem globalen Biodiversitäts-Hotspot, Herausgeber H. Lambers (Crawley, WA: University of Western Australia Publishing), 81-99.
PubMed Abstract
Byrne, M., Macdonald, B. und Coates, DJ (2002). Phylogeographische Muster und hohe cpDNA-Diversität innerhalb der Acacia acuminata (Leguminosae:Mimosoideae). komplex in Westaustralien. In: J. Evol. Biol. 15, 576–587. doi: 10.1046/j.1420-9101.2002.00429.x
CrossRef Full Text / Google Scholar
Carstens, B. C., Pelletier, T. A., Reid, N. M. und Satler, J. D. (2013). Wie man an der Artenabgrenzung scheitert. Mol. Ecol. 22, 4369–4383. doi: 10.1111/mec.12413
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Coates, D. J. (1988). Genetische Vielfalt und Populationsgenstruktur im seltenen Chittering Grass Wattle, Acacia Anomala Court. Aust. In: J. Bot. 36:273–286. doi: 10.1071/BT9880273
CrossRef Volltext / Google Scholar
Coates, D. J. (2000). Definition von Erhaltungseinheiten in einer reichen und fragmentierten Flora: Auswirkungen auf das Management genetischer Ressourcen und evolutionärer Prozesse in Pflanzen im Südwesten Australiens. Aust. In: J. Bot. 48, 329–339. doi: 10.1071/BT99018
CrossRef Volltext / Google Scholar
Coates, DJ, McArthur, SL und Byrne, M. (2015). Signifikanter Verlust der genetischen Vielfalt nach dem Aussterben der pathogengesteuerten Population bei der seltenen endemischen Banksia brownii (Proteaceae). Biol. Conserv. 192, 353–360. doi: 10.1016/j.biocon.2015.10.013
CrossRef Volltext / Google Scholar
Colgan, D. J. und Paxton, J. R. (1997). Biochemische Genetik und Erkennung eines westlichen Bestands des gemeinen Gemfisches, Rexea solandri (Scombroidea: Gempylidae)., in Australien. Mar. Freshw. 48, 103-118. doi: 10.1071/MF96048
CrossRef Volltext / Google Scholar
Übereinkommen über die biologische Vielfalt (2007). Online verfügbar unter: https://www.cbd.int/doc/meetings/cop-bureau/cop-bur-2007/cop-bur-2007-10-14-en.pdf
Cook, C. N. und Sgrò, C. M. (2017). Wissenschaft und Politik aufeinander abstimmen, um evolutionär aufgeklärten Naturschutz zu erreichen. Conserv. Biol. 31, 501–512. Ursprungsbezeichnung: 10.1111/cobi.12863
PubMed Abstract / CrossRef Full Text / Google Scholar
Cook, LG, Edwards, RD, Crisp, MD und Hardy, NB (2010). Muss Morphologie immer für neue Artenbeschreibungen erforderlich sein? Invertiert. Sys. 24, 322–332. doi: 10.1071/IS10011
CrossRef Volltext / Google Scholar
Cracraft, J. (1983). Artenkonzepte und Speziationsanalyse. Curr. Ornithologie. 1, 159–187. doi: 10.1007/978-1-4615-6781-3_6
CrossRef Volltext | Google Scholar
Crandall, K. A., Bininda-Emonds, O. R., Mace, G. M. und Wayne, R. K. (2000). Evolutionäre Prozesse in der Naturschutzbiologie. In: Trends Ecol. In: Evol. 15, 290–295. doi: 10.1016/S0169-5347(00)01876-0
PubMed Zusammenfassung | CrossRef Volltext | Google Scholar
Cutter, A. D. und Payseur, B. A. (2013). Genomische Signaturen der Selektion an verknüpften Stellen: Vereinheitlichung der Unterschiede zwischen den Arten. Nat. In: Rev. Genet. 14, 262–274. doi: 10.1038/nrg3425
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Dayrat, B. (2005). Auf dem Weg zur integrativen Taxonomie. Biol. J. Linn. Soc. 85, 407–415. doi: 10.1111/j.1095-8312.2005.00503.x5
CrossRef Volltext / Google Scholar
de Queiroz, K. (1998). „Das allgemeine Linienkonzept von Arten, Artenkriterien und der Prozess der Artbildung“, in Formen: Arten und Artbildung, D. J. Howard,R. S. H. Berlocher (New York, NY: Oxford University Press), 57-75.
Google Scholar
de Queiroz, K. (2007). Artenbegriffe und Artenabgrenzung. Syst. Biol. 56, 879–886. doi: 10.1080/10635150701701083
PubMed Zusammenfassung | CrossRef Volltext / Google Scholar
Degnan, J. H. und Rosenberg, N. A. (2009). Genbaum-Diskordanz, phylogenetische Inferenz und die Multispezies-Koaleszenz. In: Trends Ecol. In: Evol. 24, 332–340. doi: 10.1016/j.Baum.2009.01.009
PubMed Abstract / CrossRef Full Text / Google Scholar
Devey, D. S., Bateman, R. M., Fay, M. F. und Hawkins, J. A. (2008). Freunde oder Verwandte? Phylogenetik und Artenabgrenzung in der umstrittenen europäischen Orchideengattung Ophrys. Ann. Bot. 101, 385–402. doi: 10.1093/aob/mcm299
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Dizon, A. E., Lockyer, C, Perrin, W. F., Demaster, D. P. und Sisson, J. (1992). Das Bestandskonzept überdenken – ein phylogeographischer Ansatz. Conserv. Biol. 6, 24–36. doi: 10.1046/j.1523-1739.1992.610024.x
CrossRef Volltext / Google Scholar
Doughty, P., Bourke, G., Tedeschi, L., Pratt, R., Oliver, P., Palmer, R., et al. (2018). Artenabgrenzung in der Gehyra nana (Squamata: Gekkonidae). komplex: Kryptische und divergente morphologische Evolution in den australischen Monsun-Tropen mit der Beschreibung von vier neuen Arten. Zootaxa, 4403:201-244. doi: 10.11646/zootaxa.4403.2.1
PubMed Abstract / CrossRef Full Text / Google Scholar
Dynesius, M. und Jansson, R. (2014). Persistenz von Abstammungslinien innerhalb von Arten: eine vernachlässigte Kontrolle der Speziationsraten. Evolution 68, 923-934. doi: 10.1111/evo.12316
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Edwards, S. V., Potter, S., Schmitt, CJ, Bragg, JG und Moritz, C. (2016). Retikulation, Divergenz und das Phylogeographie–Phylogenetik-Kontinuumc. Prok. Natl. Acad. Sci. USA 113, 8025-8032. doi: 10.1073/pnas.1601066113
CrossRef Volltext / Google Scholar
Glaube, D. P. (1992). Erhaltungsbewertung und phylogenetische Vielfalt. Biol. Nachteil. 61, 1–10. doi: 10.1016/0006-3207(92)91201-3
CrossRef Full Text | Google Scholar
Finston, T. L., Johnson, M. S., Humphreys, W. F., Eberhard, S. M. und Halse, S. A. (2007). Die kryptische Speziation in zwei weit verbreiteten unterirdischen Amphipodengattungen spiegelt historische Entwässerungsmuster in einer alten Landschaft wider. Mol. Ecol. 16, 355–365. Ursprungsbezeichnung: 10.1111/j.1365-294X.2006.03123.x
PubMed Abstract / CrossRef Full Text / Google Scholar
Firestone, KB, Elphinstone, MS, Sherwin, WB und Houlden, BA (1999). Phylogeographische Populationsstruktur von Tiger quolls Dasyurus maculatus (Dasyuridae: Marsupalia), einem vom Aussterben bedrohten fleischfressenden Beuteltier. Mol. Ecol. 8, 1613–1625. Ursprungsbezeichnung: 10.1046/j.1365-294x.1999.00745.x
CrossRef Full Text / Google Scholar
Fišer, C., Robinson, C. T. und Malard, F. (2018). Kryptische Arten als Fenster in den Paradigmenwechsel des Artenkonzepts. Mol. Ecol. 27, 613–635. doi: 10.1111/mec.14486
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Frankham, R. (2010). Herausforderungen und Chancen genetischer Ansätze für den biologischen Naturschutz. Biol. Conserv. 143,1919–1927. doi: 10.1016/j.biocon.2010.05.011
CrossRef Volltext / Google Scholar
Frankham, R., Ballou, J. D., Dudash, M. R., Eldridge, MD, Fenster, CB, Lacy, RC, et al. (2012). Implikationen verschiedener Artenkonzepte für den Erhalt der Biodiversität. Biol. Conserv. 153, 25–31. doi: 10.1016/j.biocon.2012.04.034
CrossRef Volltext / Google Scholar
Fraser, D. J. und Bernatchez, L. (2001). Adaptive evolutionäre Erhaltung: Auf dem Weg zu einem einheitlichen Konzept zur Definition von Erhaltungseinheiten. Mol. Ecol. 10, 2741–2752. Ursprungsbezeichnung: 10.1046/j.1365-294X.2001.t01-1-01411.x
PubMed Zusammenfassung | CrossRef Volltext / Google Scholar
Funk, W. C., Mckay, J. K., Hohenlohe, P. A., und Allendorf, F. W. (2012). Nutzung der Genomik zur Abgrenzung von Erhaltungseinheiten. In: Trends Ecol. In: Evol. 27, 489–496. doi: 10.1016/j.Baum.2012.05.012
PubMed Abstract / CrossRef Full Text / Google Scholar
Garnett, S. T. und Christidis, L. (2017). Taxonomie Anarchie behindert die Erhaltung. Natur 546, 25-27. doi: 10.1038/546025a
PubMed Abstract / CrossRef Full Text / Google Scholar
Gippoliti, S., Cotterill, F. P. D., Zinner, D. und Groves, C. P. (2018). Auswirkungen der taxonomischen Trägheit auf die Erhaltung der afrikanischen Huftiervielfalt: Überblick. Biol. Offenbarung 93, 115-130. Ursprungsbezeichnung: 10.1111/brv.12335
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Grant, V. (1981). Pflanzenspeziation. New York, NY: Columbia University Press.
PubMed Abstract / Google Scholar
Hamilton, CW und Reichard, SH (1992). Aktuelle Praxis bei der Verwendung von Unterarten, Sorten und Formen bei der Klassifizierung von Wildpflanzen. Taxon 41, 485-498. doi: 10.2307/1222819
CrossRef Volltext / Google Scholar
Harvey, M. S., Rix, M. G., Framenau, V. W., Hamilton, Z. R., Schmidt, M., Schmidt, R., et al. (2011). Schutz der Unschuldigen: Die Untersuchung endemischer Taxa mit kurzer Reichweite verbessert die Ergebnisse des Naturschutzes. Invertiert. Syst. 25, 1–10. doi: 10.1071/IS11011
CrossRef Volltext / Google Scholar
Hey, J., Waples, RS, Arnold, ML, Butlin, RK und Harrison, RG (2003). Artenunsicherheit in Biologie und Naturschutz verstehen und konfrontieren. In: Trends Ecol. In: Evol. 18, 597–603. doi: 10.1016/j.Baum.2003.08.014
CrossRef Volltext / Google Scholar
Hoban, S. M., Hauffe, H. C., Perez-Espona, S., Arntzen, J. W., Bertorelle, G., Bryja, J., et al. (2013). Die genetische Vielfalt in den Vordergrund der Naturschutzpolitik und des Naturschutzmanagements rücken. Conserv. Genet. Resour. 5, 593–598. ust-IDNR.: 10.1007/s12686-013-9859- y
CrossRef Volltext / Google Scholar
Isaac, NJ, Mallet, J. und Mace, GM (2004). Taxonomische Inflation: ihr Einfluss auf Makroökologie und Naturschutz. In: Trends Ecol. In: Evol. 19, 464–469. doi: 10.1016/j.Baum.2004.06.004
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Isaac, N. J., Turvey, S. T., Collen, B., Waterman, C., und Baillie, J. E. (2007). Säugetiere am RANDE: Erhaltungsprioritäten basierend auf Bedrohung und Phylogenie. Plus EINS 2: e296. doi: 10.1371/Zeitschrift.pone.0000296
PubMed Abstract / CrossRef Full Text / Google Scholar
Jones, M. R. und Good, J. M. (2016). Gezielte Erfassung in der evolutionären und ökologischen Genomik. Mol. Ecol. 25, 185–202. doi: 10.1111/mec.13304
PubMed Abstract / CrossRef Full Text / Google Scholar
Jorger, K. M. und Schrödl, M. (2013). Wie beschreibt man eine kryptische Spezies? Praktische Herausforderungen der molekularen Taxonomie. Front. Zool. 10:59. doi: 10.1186/1742-9994-10-59
PubMed Zusammenfassung | CrossRef Volltext / Google Scholar
Laikre, L., Allendorf, F., Aroner, L. C., Baker, S., Gregovich, D. P., Hansen, M. M., et al. (2010). Vernachlässigung der genetischen Vielfalt bei der Umsetzung des Übereinkommens über die biologische Vielfalt. Conserv. Biol. 24, 86–88. doi: 10.1111/j.1523-1739.2009.01425.x
PubMed Abstract / CrossRef Full Text / Google Scholar
Laikre, L., Palm, S. und Ryman, N. (2005). Genetische Populationsstruktur von Fischen: Implikationen für das Küstenzonenmanagement. Ambio 34,111-119. doi: 10.1579/0044-7447-34.2.111
PubMed Zusammenfassung | CrossRef Volltext / Google Scholar
Laity, T., Laffan, S. W., González-Orozco, CE, Glaube, D. P., Rosauer, D. F., Byrne, M., et al. (2015). Phylodiversität zur Information der Naturschutzpolitik: Ein australisches Beispiel. Sci. Gesamt Environ. 534, 131–143. doi: 10.1016/j.scitotenv.2015.04.113
PubMed Abstract / CrossRef Full Text / Google Scholar
Leache, A. D. und Oaks, J. R. (2017). Der Nutzen des Einzelnukleotidpolymorphismus (SNP). daten in der Phylogenetik. Ann. Rev. Ecol. Syst. 48, 69–84. doi: 10.1146/annurev-ecolsys-110316-022645
CrossRef Full Text | Google Scholar
Llorens, T. M., Macdonald, B., McArthur, S., Coates, D. J., and Byrne, M. (2015). Disjunct, highly divergent genetic lineages within two rare Eremophila (Scrophulariaceae: Myoporeae). species in a biodiversity hotspot: implications for taxonomy and conservation. Biol. J. Linn. Soc. 177, 96–111. doi: 10.1111/boj.12228
CrossRef Full Text | Google Scholar
Love Stowell, S. M., Pinzone, C. A., and Martin, A. P. (2017). Überwindung von Hindernissen für aktive Interventionen für die genetische Vielfalt. Biodiversität. Conserv. 26, 1753–1765. ust-IDNR.: 10.1007/s10531-017-1330- z
CrossRef Volltext / Google Scholar
Mace, G. M. (2004). Die Rolle der Taxonomie im Artenschutz. Philos. Transeuropäischen. R. Soc. In: Biol. Sci. 359, 711–719. Ursprungsbezeichnung: 10.1098/rstb.2003.1454
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Mallet, J. (2007). Hybride Speziation. Natur 446, 279-283. doi: 10.1038/nature05706
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Mayr, E. (1942). Systematik und Ursprung der Arten. New York, NY: Columbia University Press.
Google Scholar
Mee, J. A., Bernatchez, L., Reist, J. D., Rogers, S. M. und Taylor, E. B. (2015). Identifizierung benennbarer Einheiten für die Priorisierung der intraspezifischen Erhaltung: Ein hierarchischer Ansatz für den Lake whitefish species complex (Coregonus spp.). In: Evol. Appl. 8, 423–441. Ursprungsbezeichnung: 10.1111/eva.12247
PubMed Abstract | CrossRef Full Text | Google Scholar
Millar, M. A., and Byrne, M. (2013). Cryptic divergent lineages of Pultenaea pauciflora M.B. Scott (Fabaceae: Mirbelieae) exhibit different evolutionary history. Biol. J. Linn. Soc. 108, 871–881. doi: 10.1111/bij.12002
CrossRef Full Text | Google Scholar
Moritz, C. (1994). Defining evolutionarily significant units for conservation. Trends Ecol. Evol. 9, 373–375. doi: 10.1016/0169-5347(94)90057-4
CrossRef Full Text | PubMed Abstract
Moritz, C. (1999). Erhaltungseinheiten und Translokationen: Strategien zur Erhaltung evolutionärer Prozesse. Hereditas 130, 217-228. doi: 10.1111/j.1601-5223.1999.00217.x
CrossRef Volltext / Google Scholar
Moritz, C. (2002). Strategien zum Schutz der biologischen Vielfalt und der Evolutionsprozesse, die sie erhalten. Syst. Biol. 51, 238–254. doi: 10.1080/10635150252899752
PubMed Zusammenfassung | CrossRef Volltext / Google Scholar
Moritz, C. und Potter, S. (2013). Die Bedeutung einer evolutionären Perspektive in der naturschutzpolitischen Planung. Mol. Ecol. 22, 5969–5971. doi: 10.1111/mec.12565
PubMed Abstract | CrossRef Full Text / Google Scholar
Moritz, C., Pratt, R. C., Bank, S., Bourke, G., Bragg, J. G., Doughty, P., et al. (2018). Kryptische Abstammungsvielfalt, Körpergrößenabweichung und Sympatrie in einem Artenkomplex australischer Eidechsen (Gehyra). Evolution 27, 54-66. doi: 10.1111/evo.13380
CrossRef Volltext / Google Scholar
Morrison, W. R., Lohr, J. L., Duchen, P., Wilches, R., Trujillo, D., Mair, M., et al. (2009). Die Auswirkungen taxonomischer Veränderungen auf den Naturschutz: Tötet es, kann es retten oder ist es einfach irrelevant? Biol. Nachteil. 142, 3201–3206. doi: 10.1016/j.biocon.2009.07.019
CrossRef Volltext / Google Scholar
Murphy, N. P., Adams, M., Guzik, M. T. und Austin, A. D. (2013). Außergewöhnlicher Mikroendemismus in australischen Wüstenfrühlings-Amphipoden. Mol. Phylogenet. In: Evol. 66, 645–653. doi: 10.1016/j.ympev.2012.10.013
PubMed Abstract / CrossRef Full Text / Google Scholar
Murphy, N. P., King, R. A. und Delean, S. (2015). Arten, ESUs oder Populationen? Abgrenzung und Beschreibung der morphologisch kryptischen Vielfalt in australischen Wüstenfrühlings-Amphipoden. Invertiert. Syst. 29, 457–467. doi: 10.1071/IS14036
CrossRef Volltext / Google Scholar
Nater, A., Mattle-Greminger, M. P., Nurcahyo, A., Nowak, M. G., de Manual, M., Desai, T., et al. (2017). Morphometrische, Verhaltens- und genomische Beweise für eine neue Orang-Utan-Art. Curr. In: Cell Biol. 27, 3487–3498. Ursprungsbezeichnung: 10.1016/j.cub.2017.09.047
CrossRef Volltext / Google Scholar
Nicotra, A. B., Chong, C., Bragg, J. G., Ong, C. R., Aitken, N. C., Chuah, A., et al. (2016). Population und phylogenomische Zersetzung durch Genotypisierung durch Sequenzierung in australischer Pelargonie. Mol. Ecol. 25, 2000–2014. doi: 10.1111/mec.13584
PubMed Abstract / CrossRef Full Text / Google Scholar
Nistelberger, H., Byrne, M., Coates, D. und Roberts, JD (2014a). Eine starke phylogeographische Struktur in einem Tausendfüßler weist auf eine pleistozäne Vikarianz zwischen Populationen auf gebänderten Eisenformationen im halbtrockenen Australien hin. Plus EINS 9: e0093038. doi: 10.1371/Zeitschrift.pone.0093038
PubMed Abstract / CrossRef Full Text / Google Scholar
Nistelberger, H., Gibson, N., Macdonald, B., Tapper, S.-L. und Byrne, M. (2014b). Phylogeographische Nachweise für zwei mesische Refugien in einem Biodiversitäts-Hotspot. Vererbung 113, 454-463. doi: 10,1038/hdy.2014.46
PubMed Abstract / CrossRef Full Text / Google Scholar
Ogilvie, H. A., Heled, J., Xie, D. und Drummond, A. J. (2016). Rechenleistung und statistische Genauigkeit von Messungen und Vergleichen mit anderen Methoden. Syst. Biol. 65, 381–396. doi: 10.1093/sysbio/syv118
PubMed Abstract / CrossRef Full Text / Google Scholar
Oliver, P. M., Keogh, J. S. und Moritx, C. (2015). Neue Ansätze zur Katalogisierung und zum Verständnis der evolutionären Vielfalt: eine Perspektive aus der australischen Herpetologie. Aust. J. Zool. 62, 417–430. doi: 10.1071/ZO14091
CrossRef Volltext / Google Scholar
Oliver, P. M., Laver, R. J., Demello Martins, F., Pratt, R. C., Hunjan, S. und Moritz, C. (2017). Ein neuartiger Hotspot des Wirbeltierendemismus und ein evolutionäres Refugium im tropischen Australien. Taucher. Distrib. 23, 53–66. zertifikate: 10.1111/ddi.12506
CrossRef Volltext / Google Scholar
Oliver, P. M. und Lee, M. S. Y. (2010). Die botanischen und zoologischen Codes behindern die Biodiversitätsforschung, indem sie die Veröffentlichung unbenannter neuer Arten verhindern. Taxon 59, 1201-1205. doi: 10.2307/20773990
CrossRef Volltext / Google Scholar
Padial, J., Miralles, A., De la Riva, I. und Vences, M. (2010). Die integrative Zukunft der Taxonomie. Front. Zool. 7:16. doi: 10.1186/1742-9994-7-16
PubMed Abstract | CrossRef Full Text / Google Scholar
Palsbøll, PJ, Bérubé, M. und Allendorf, FW (2007). Identifizierung von Managementeinheiten anhand populationsgenetischer Daten. In: Trends Ecol. In: Evol. 22, 11–16. doi: 10.1016/j.Baum.2006.09.003
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Patton, JL und Smith, MF (1994). Paraphylie, Polyphylie und die Art der Artengrenzen in Pocket Gophers (Gattung Thomomys). Syst. Biol. 43, 11–26. doi: 10.1093/sysbio/43.1.11
CrossRef Volltext / Google Scholar
Pierson, J. C., Coates, D. J., Oostermeijer, J. G. B., Beissinger, S. R., Bragg, J. G., Sunnucks, P., et al. (2016). Genetische Faktoren in Wiederauffüllungsplänen für bedrohte Arten auf drei Kontinenten. Front. Ecol. ENVIRON. 14, 433–440. doi: 10.1002/Gebühr.1323
CrossRef Volltext / Google Scholar
Potter, S., Bragg, J. G., Peter, B. M., Bi, K. und Moritz, C. (2016). Phylogenomik an den Spitzen: Abstammungslinien und ihre demografische Geschichte in einer tropischen Eidechse ableiten, Carlia amax. Mol. Ecol. 25, 1367–1380. doi: 10.1111/mec.13546
PubMed Abstract / CrossRef Full Text / Google Scholar
Potter, S., Xue, A. T., Bragg, J. G., Rosauer, D. F., Roycroft, E. J. und Moritz, C. (2018). Pleistozäne Klimaveränderungen treiben die Diversifizierung in einer tropischen Savanne voran. Mol. Ecol. 27, 520–532. doi: 10.1111/mec.14441/voll
PubMed Abstract | CrossRef Volltext / Google Scholar
Rabosky, D. L., Hutchinson, M. N., Donnellan, S. C., Talaba, A. L. und Lovette, I. J. (2014). Phylogenetische Demontage von Artengrenzen in einer weit verbreiteten Gruppe australischer Skinke (Scincidae: Ctenotus). Mol. Phylogenet. In: Evol. 77, 71–82. doi: 10.1016/j.ympev.2014.03.026
PubMed Zusammenfassung | CrossRef Volltext / Google Scholar
Ralls, K., Ballou, J. D., Dudash, M. R., Eldridge, M. D. B., Fenster, C. B., Lacy, R. C., et al. (2018). Fordern Sie einen Paradigmenwechsel im genetischen Management fragmentierter Populationen. Conserv. Lett. 11, 1–6. Ursprungsbezeichnung: 10.1111/conl.124126
CrossRef Volltext / Google Scholar
Rannala, B. (2015). Die Kunst und Wissenschaft der Artenabgrenzung. Curr. Zool. 61, 846–853. doi: 10.1093/czoolo/61.5.846
CrossRef Volltext / Google Scholar
Riedel, A., Sagata, K., Suhardjono, Y. R., Tänzler, R. und Balke, M. (2013). Integrative Taxonomie auf der Überholspur – für mehr Nachhaltigkeit in der Biodiversitätsforschung. Front. Zool. 10:15. doi: 10.1186/ 1742-9994-10-15
PubMed Zusammenfassung | CrossRef Volltext / Google Scholar
Rieseberg, LH, und Willis, JH (2007). Pflanzenspeziation. Wissenschaft 317, 910-914. doi: 10.1126/Wissenschaft.1137729
PubMed Abstract | CrossRef Full Text / Google Scholar
Rosauer, D. F., Blom, M. P. K., Bourke, G., Catalano, S., Donnellan, S., Mulder, E., et al. (2016). Phylogeographie, Hotspots und Naturschutzprioritäten: ein Beispiel vom oberen Ende Australiens. Biol. Nachteil. 204, 83–93. doi: 10.1016/j.biocon.2016.002
CrossRef Volltext / Google Scholar
Rosauer, D. F., Byrne, M., Blom, M. P. K., Coates, D. J., Donnellan, S., Doughty, P., et al. (2018). Reale Naturschutzplanung für die evolutionäre Vielfalt in der Kimberley, Australien, umgeht unsichere Taxonomie Conserv. Lett. 11:e12438. Ursprungsbezeichnung: 10.1111/conl.12438
CrossRef Volltext / Google Scholar
Rosenblum, E. B., Sarver, B. A. J., Brown, J. W., Des Roches, S., Hardwick, K. M., Hether, T. D., et al. (2012). Goldlöckchen trifft Santa Rosalia: Ein ephemeres Speziationsmodell erklärt Diversifikationsmuster über Zeitskalen hinweg. In: Evol. Biol. 39, 255–261. doi: 10.1007/s11692-012-9171- x
PubMed Abstract / CrossRef Full Text / Google Scholar
Roux, C., Fraïsse, C., Romiguier, J., Anciaux, Y., Galtier, N. und Bierne, N. (2016). Licht auf die Grauzone der Speziation entlang eines Kontinuums genomischer Divergenz werfen. PLoS Biol. 14:e2000234. doi: 10.1371/Zeitschrift.pbio.2000234
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Ryder, O. A. (1986). Artenschutz und Systematik – das Dilemma der Unterarten. In: Trends Ecol. In: Evol. 1, 9–10. doi: 10.1016/0169-5347(86)90059-5
CrossRef Volltext | Google Scholar
Sampson, J. F., Byrne, M., Yates, C. J., Gibson, N., Thavornkanlapachai, R., Stankowski, S., et al. (2014). Die zeitgenössische pollenvermittelte Geneinwanderung spiegelt die historische Isolation eines seltenen Strauchs in einer fragmentierten Landschaft wider. Vererbung 112, 172-181. doi: 10,1038/hdy.2013.89
PubMed Abstract / CrossRef Full Text / Google Scholar
Santamaría, L. und Méndez, P. F. (2012). Evolution in der Biodiversitätspolitik: aktuelle Lücken und zukünftige Bedürfnisse. In: Evol. Appl. 5, 202–218. doi: 10.1111/j.1752-4571.2011.00229.x
PubMed Abstract | CrossRef Full Text / Google Scholar
Schlick-Steiner, B. C., Steiner, F. M., Seifert, B., Stauffer, C., Christian, E. und Crozier, R. H. (2010). Integrative Taxonomie: ein Multisource-Ansatz zur Erforschung der Biodiversität. Jahr. In: Rev. Entomol. 55, 421–438. doi: 10.1146/annurev-ento-112408-085432
PubMed Abstract | CrossRef Full Text | Google Scholar
Seehausen, O., Takimoto, G., Roy, D. und Jokela, J. (2008). Speziationsumkehr und Biodiversitätsdynamik mit Hybridisierung in sich verändernden Umgebungen. In: Mol Ecol. 17, 30–44. Ursprungsbezeichnung: 10.1111/j.1365-294X.2007.03529.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Schäfer, K. A., Thiele, K. R., Sampson, J., Coates, D. J., und Byrne, M. (2015). Eine seltene, neue Art von Atriplex (Chenopodiaceae) mit zwei genetisch unterschiedlichen, aber morphologisch kryptischen Populationen im trockenen Westaustralien: Implikationen für Taxonomie und Naturschutz. Aust. Syst. Bot. 28, 234–245. doi: 10.1071/SB15029
CrossRef Volltext / Google Scholar
Singhal, S. und Bi, K. (2017). Geschichte räumt Durcheinander auf: Der Einfluss der Zeit auf die Divergenz und Introgression in einer tropischen Nahtzone. Evolution 71, 1888-1899. doi: 10.1111/evo.13278
PubMed Abstract / CrossRef Full Text / Google Scholar
Singhal, S., Hoskin, C., Couper, P., Potter, S. und Moritz, C. (2018). Rahmen für die Lösung kryptischer Arten: eine Fallstudie von den Eidechsen der australischen feuchten Tropen. Syst. Biol. . doi: 10.1093/sysbio/syy
PubMed Abstract | CrossRef Full Text / Google Scholar
Singhal, S. und Moritz, C. (2013). Reproduktive Isolation zwischen phylogeographischen Linien skaliert mit Divergenz. Prok. Roy. Soc B 280:20132246. Ursprungsbezeichnung: 10.1098/rspb.2013.2246026
PubMed Zusammenfassung | CrossRef Volltext / Google Scholar
Steane, D. A., Potts, B. M., McLean, E., Collins, L., Prober, S. M., Stock, W. D., et al. (2015). Genomweite Scans zeigen kryptische Populationsstruktur in einem trocken angepassten Eukalyptus. Baum Genet. Genome 11:33. doi: 10.1007/s11295-015-0864- z
CrossRef Volltext / Google Scholar
Struck, T. H., Feder, J. L., Bendiksby, M., Birkeland, S., Cerca, J., Gusarov, V. I., et al. (2017). Evolutionäre Prozesse finden, die in kryptischen Arten verborgen sind. In: Trends Ecol. In: Evol. 33, 153–163. doi: 10.1016/j.Baum.2017.11.007
PubMed Abstract / CrossRef Full Text / Google Scholar
Sukumaran, J. und Knowles, L. L. (2017). Die Multispezies-Koaleszenz begrenzt die Struktur nicht Arten. Prok. Nat. Aca. Sci. USA 114, 1607-1612. doi: 10.1073/pnas.1607921114
PubMed Abstract / CrossRef Full Text / Google Scholar
Swarts, ND, Clements, MA, Bower, CC und Miller, JT (2014). Definition von Erhaltungseinheiten in einem Komplex morphologisch ähnlicher, sexuell täuschender, stark gefährdeter Orchideen. Biol. Nachteil. 174, 55–64. doi: 10.1016/j.biocon.2014.03.017
CrossRef Full Text / Google Scholar
Tautz, D., Arctander, P., Minelli, A., Thomas, R. H. und Vogler, A. P. (2003). Ein Plädoyer für DNA-Taxonomie. In: Trends Ecol. In: Evol. 18, 70–74. doi: 10.1016/S0169-5347(02)00041-1
CrossRef Volltext | Google Scholar
Taylor, HR, Dussex, N. und van Heezik, Y. (2017). Überbrückung der Lücke in der Naturschutzgenetik durch Identifizierung von Umsetzungsbarrieren für Naturschutzpraktiker. Glob. Ecol. Conserv. 10, 231–242. doi: 10.1016/j.gecco.2017.04.001
CrossRef Volltext / Google Scholar
Thomson, S. A., Pyle, R. L., Ahyong, S. T., Alonso-Zarazaga, M., Ammirati, J., Araya, J. F., et al. (2018). Eine auf Wissenschaft basierende Taxonomie ist für den globalen Naturschutz notwendig. PLoS Biol. 16:e2005075. doi: 10.1371/Zeitschrift.pbio.2005075
PubMed Abstract / CrossRef Full Text / Google Scholar
Vieites, D. R., Wollenberg, K. C., Andreonec, F., Köhler, J., Glaw, F., und Vences, M. (2009). Enorme Unterschätzung der Biodiversität Madagaskars, belegt durch ein integratives Amphibieninventar. Prok. Natl. Acad. Sci. USA 106, 8267-8272. doi: 10.1073/pnas.0810821106
CrossRef Volltext
Vogler, A. P. und de Salle, R. (1994). Diagnose von Einheiten des Naturschutzmanagements. Nachteil. Biol. 8, 354–363. doi: 10.1046/j.1523-1739.1994.08020354.x
CrossRef Volltext / Google Scholar
Waples, R. S. (1991). Pazifischer Lachs, Oncorhynchus spp., und die Definition von „Arten“ unter dem Gesetz über gefährdete Arten. Mar. Fisch. Offenbarung 53, 11-22.
Google Scholar
Waples, R. S. (1995). „Evolutionär bedeutende Einheiten und die Erhaltung der biologischen Vielfalt unter dem Endangered Species Act“, in Evolution und das aquatische Ökosystem: Definition einzigartiger Einheiten im Bevölkerungsschutz, eds JL Nielsen (Bethesda, MD: American Fisheries Society), 8-27.
Google Scholar
Waples, R. S., Gustafson, R. G., Weitkamp, L. A., Myers, J. M., Jjonson, O. W., Busby, P. J., et al. (2001). Charakterisierung der Vielfalt von Lachs aus dem pazifischen Nordwesten. J. Fisch Biol. 59, 1–41. doi: 10.1111/j.1095-8649.2001.tb01376.x
CrossRef Full Text / Google Scholar
Waples, R. S., Nammack, M., Cochrane, J. F. und Hutchings, J. A. (2013). Eine Geschichte aus zwei Akten: Praktiken zur Auflistung gefährdeter Arten in Kanada und den Vereinigten Staaten. BioScience 63, 723-734. doi: 10.1093/Biowissenschaften/63.9.723
CrossRef Volltext / Google Scholar
Waples, R. S., Punt, A. E. und Cope, J. M. (2008). Integration genetischer Daten in die Bewirtschaftung der Meeresressourcen: Wie können wir das besser machen? Fisch 9, 423-449. Ursprungsbezeichnung: 10.1111/j.1467-2979.2008.00303.x
CrossRef Full Text / Google Scholar
Wege, J. A., Thiele, K. R., Schäfer, K. A., Metzger, R., Macfarlane, T. D., und Coates, D. J. (2015). Strategische Taxonomie in einer biodiversen Landschaft: ein neuartiger Ansatz zur Maximierung der Erhaltungsergebnisse für seltene und wenig bekannte Flora. Biodiversität. Conserv. 24, 17–32. ust-IDNR.: 10.1007/s10531-014-0785-4
CrossRef Volltext / Google Scholar
Whitlock, R. (2014). Beziehungen zwischen adaptiver und neutraler genetischer Vielfalt und ökologischer Struktur und Funktionsweise: eine Metaanalyse. In: J. Ecol. 102, 857–872. doi: 10.1111/1365-2745.12240
PubMed Zusammenfassung | CrossRef Volltext | Google Scholar
Wiley, E. O. (1978). Das evolutionäre Artenkonzept überdacht. Syst. Biol. 27, 17–26. doi: 10.2307/2412809
CrossRef Volltext / Google Scholar
Wilson, E. O. (2017). Biodiversitätsforschung erfordert mehr Stiefel vor Ort. Nat. Ecol. In: Evol. 1, 1590–1591. ust-IDNR.: 10.1038/s41559-017-0360- y
PubMed Abstract / CrossRef Full Text / Google Scholar
Yang, Z. H. und Rannala, B. (2014). Ungeführte Artenabgrenzung unter Verwendung von DNA-Sequenzdaten aus mehreren Loci. Mol. Biol. In: Evol. 31, 3125–3135. doi: 10.1093/molbev/msu279
PubMed Zusammenfassung | CrossRef Volltext / Google Scholar
Yeates, D. K., Seago, A., Nelson, L., Cameron, S. L., Joseph, L. und Trueman, W. H. (2011). Integrative Taxonomie oder iterative Taxonomie? Sys. Ent. 36, 209–217. doi: 10.1111/j.1365-3113.2010.00558.x
CrossRef Full Text | Google Scholar