- Introducción
- La genómica, la especiación y la Taxonomía
- Genómica y Unidades de Conservación Intraespecíficas
- Filogenómica y Conservación
- Las Definiciones legislativas del Mundo real
- Traducción a Manejo
- Conclusiones y Recomendaciones
- Las contribuciones de los autores
- Declaración de Conflicto de Intereses
- Agradecimientos
Introducción
La política internacional de conservación reconoce la biodiversidad en tres niveles, ecosistema, especie y genética, y que la gestión debe apuntar a retener los tres (Convenio sobre la Diversidad Biológica, 2007). Esto se refleja claramente en el Convenio sobre la Diversidad Biológica, las Metas de Aichi para la Diversidad Biológica, acordadas en 2010 (https://www.cbd.int/sp/targets/), donde hay referencias específicas en los objetivos y metas, no solo a los ecosistemas y las especies, sino también a la diversidad genética. Sin embargo, los enfoques actuales para la conservación de la biodiversidad se basan en gran medida en las áreas geográficas, los ecosistemas, las comunidades ecológicas y las especies, con menos atención a la diversidad genética y al continuo de especies y poblaciones. De hecho, la mayoría de los profesionales de la conservación se centran en las especies como métrica de la biodiversidad, con cierta consideración de las unidades intraespecíficas dentro de las especies amenazadas de manejo intensivo. En este contexto, existe un deseo natural de emplear categorizaciones simples: especies con nombre, y otros taxones con nombre (es decir, subespecies), y a veces unidades de conservación dentro de las especies. Las especies en particular proporcionan una medida común de biodiversidad que sustenta gran parte del esfuerzo científico y de gestión (Mace, 2004; Wilson, 2017). Como unidad fundamental de conservación, las especies se utilizan a menudo para cuantificar el valor de la biodiversidad a través de listas de especies dentro de áreas protegidas, la identificación de especies amenazadas dentro de jurisdicciones y como base para procedimientos de bioseguridad. Además, las especies son típicamente las entidades con las que comunicamos los problemas de conservación al público.
Este impulso de simplemente identificar unidades discretas en la naturaleza va en contra de varias realidades relacionadas. Primero, la especiación no es instantánea. Más bien es típicamente un proceso prolongado (Dynesius y Jansson, 2014; Roux et al., 2016), progresando de poblaciones aisladas, tal vez sujetas a diferentes presiones de selección, a especies no ambiguas en escalas de tiempo largas, con frecuentes reversiones causadas por cambios ambientales o simples fusiones genéticas a medida que los rangos se reconectan (Figura 1; Seehausen et al., 2008; Rosenblum et al., 2012). En segundo lugar, la mayoría de las especies reconocidas no son genéticamente uniformes. La divergencia (deriva y aislamiento prolongado) y la reticulación (flujo genético, hibridación, introgresión y recombinación) dan forma efectiva a la variación geográfica y genómica dentro de las especies (Cutter y Payseur, 2013; Edwards et al., 2016) y algunas especies pueden estructurarse en poblaciones aisladas largas, algunas de las cuales podrían justificar el reconocimiento como especies completas pero quizás morfológicamente crípticas (Bickford et al., 2007; Jorger y Schrödl, 2013). En tercer lugar, como las especies con nombre reconocidas desde hace mucho tiempo no son equivalentes en términos de cuánto legado evolutivo representan, en pocas palabras, un ornitorrinco (como representante de un linaje antiguo divergente) puede considerarse que vale más que un roedor recientemente divergido (Isaac et al., 2007). En cuarto lugar, las designaciones de especies no están hechas de piedra, sino que son hipótesis que se deben probar a medida que se dispone de nueva evidencia (Hey et al., 2003). Cada una de estas cuestiones crea complejidad y ambigüedad en la delimitación de las especies en general y en la asignación de atención a la conservación a ellas o a sus poblaciones componentes. Además, la delimitación de especies también está sujeta a diferentes conceptos de especies y formas de diagnosticarlas, lo que ha causado gran consternación entre los biólogos conservacionistas (Mace, 2004; Frankham et al., 2012; Garnett y Christidis, 2017).
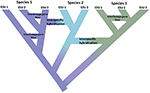
la Figura 1. La especiación como un continuo que progresa de poblaciones aisladas a especies no ambiguas, como se muestra en el cambio de color con los colores que representan diferentes especies. Algunos linajes que surgen debido al aislamiento de la población se convierten en verdaderas especies con los nodos internos del árbol que representan el inicio del proceso de especiación. Otros linajes no se convierten en especies verdaderas, pero divergen con el tiempo, lo que indica que las especies no son genéticamente uniformes. Estos linajes se muestran como Unidades Evolutivamente Significativas (UES). El flujo génico entre linajes está indicado para las Especies 1 y 3. En este caso, la independencia evolutiva puede ser transitoria porque dos linajes pertenecientes a la misma especie no están aislados reproductivamente. Si bien permanecen como linajes distintos (especies), la hibridación interespecífica puede ocurrir con un intercambio genético limitado (cambio de color) entre especies.
La implementación de estrategias de conservación efectivas que tengan como objetivo proteger tanto las especies como la diversidad genética se beneficiaría de una visión más coherente de cómo delimitamos las especies y otras unidades en el continuo población-especie, y de cómo se interpretan estas unidades en diferentes legislaciones a nivel mundial. Las nuevas capacidades de la genómica brindan oportunidades y desafíos para lograr estos objetivos. En lo siguiente consideramos: (i) cómo se pueden conciliar los análisis a escala genómica con un enfoque conservador para la delimitación y el nombramiento de especies, al tiempo que se identifican los principales componentes de la diversidad genética dentro de las especies, (ii) cómo se puede utilizar la legislación de conservación existente en varios continentes, que define «especies» para incluir una amplia gama de categorías infraespecíficas, dada la alta resolución que ofrecen las pantallas genómicas, y (iii) cómo los conocimientos sobre los procesos de especiación y la estructuración de la variación de la genómica se alinean con la legislación y luego con la gestión. A continuación, sugerimos algunas formas de alinear las unidades de conservación con los enfoques legislativos para proteger el espectro completo de la diversidad, desde las poblaciones hasta las especies.
La genómica, la especiación y la Taxonomía
Al igual que muchos campos de la ciencia biológica, la sistemática, la práctica de nombrar taxones y comprender sus relaciones y cómo se forman, está siendo significativamente avanzada por la genómica y los modelos estadísticos. Ya sea a través de la secuenciación del genoma completo (por ejemplo, Nater et al., 2017) o muestreo del genoma (SNPs, secuenciación de captura de objetivos; Jones y Good, 2016; Leache y Robles, 2017), ahora tenemos una resolución sin precedentes de los patrones de diversidad genética (Funk et al., 2012). Junto con modelos estadísticos cada vez más potentes para inferir historias de divergencia genómica y de especies, estos datos están proporcionando nuevos conocimientos sobre los procesos evolutivos que generan variaciones genéticas y de especies.
La aplicación de estas nuevas herramientas genera dos perspectivas opuestas. Por un lado, muchos estudios están revelando poblaciones genéticas altamente divergentes dentro de las especies nombradas, las llamadas especies crípticas (Bickford et al., 2007; Jorger y Schrödl, 2013; Struck et al., 2017). Esto no es nada nuevo, ya que se basa en una larga historia de genética de poblaciones espaciales y filogeografía molecular que ha sido un esfuerzo productivo durante tres décadas (Avise et al., 1987). Lo que la genómica aporta a este esfuerzo es una mayor resolución de la estructura de la población y datos que son suficientes para inferir estadísticamente las historias de la población y probar modelos alternativos de divergencia (Degnan y Rosenberg, 2009; Sukumaran y Knowles, 2017). Por otro lado, los análisis a escala genómica también están revelando que (tal como se ha reconocido durante mucho tiempo para las plantas), el intercambio genético (introgresión) entre especies animales es más común de lo que se pensaba, tanto durante como después de la especiación, e incluso puede impulsar nuevas radiaciones adaptativas (Mallet, 2007; Rieseberg y Willis, 2007; Arnold y Kunte, 2017) Figura 1. En conjunto, estas ideas enfatizan aún más que la especiación es prolongada (Roux et al., 2016) y revelan las fuerzas opuestas que operan a través del continuo filogeografía-filogenética (Edwards et al., 2016).
¿Cómo se conecta todo esto con los objetivos de taxonomía y conservación? Existe una preocupación fuerte y justificable de que el aumento de la resolución proporcionado por las pantallas genómicas podría conducir a una división excesiva desenfrenada de las especies (Isaac et al., 2004), restringiendo potencialmente la flexibilidad de manejo y relegando a pequeñas poblaciones genéticamente divergentes a la endogamia y la eventual extinción (Frankham et al., 2012). Por el contrario, Gippoliti et al. (2018) argumentan que la inercia taxonómica, que resultó en el fracaso previo para reconocer muchas especies de ungulados africanos, había resultado en la depresión de la exogamia después de la mezcla y la falta de protección de taxones geográficamente restringidos. Su punto de vista (y ver también Morrison et al., 2009) es que no hay evidencia de que la inflación taxonómica esté teniendo efectos negativos en la conservación de los ungulados (Gippoliti et al., 2018). Una preocupación relacionada es que la taxonomía inestable retrasa tanto la práctica de la conservación como las percepciones públicas (Garnett y Christidis, 2017). Estas preocupaciones están en parte impulsadas por el largo debate en torno a los conceptos de especie y los métodos de diagnóstico en biología sistemática, en particular la aplicación del Concepto de Especie Filogenética que sostiene que las especies son unidades mínimamente diagnosticables (Cracraft, 1983). Sin embargo, la mayoría de los taxónomos son inherentemente conservadores, solo nombran especies cuando hay concordancia entre múltiples líneas de evidencia (Dayrat, 2005; Padial et al., 2010; Schlick-Steiner et al., 2010: Yeates et al., 2011). Nuestra opinión es que las especies deben representar linajes robustos en evolución independiente que permanecen en gran medida intactos cuando están en contacto con parientes cercanos, de acuerdo con la intención del Concepto de Especie Evolutiva (Wiley, 1978) y el Concepto de Especie de Linaje Generalizado (de Queiroz, 1998, 2007). En la práctica, esto requiere evidencia directa de aislamiento reproductivo o sustitutos razonables del mismo, al tiempo que permite un intercambio genético limitado(Singhal et al., 2018).
Los datos genómicos proporcionan mayor poder que antes para identificar la diferenciación y la divergencia dentro de las especies y entre ellas, lo que requiere una mayor consideración de cómo esto afecta la delineación de las unidades taxonómicas. Cuando las poblaciones son muy divergentes genética y fenotípicamente en relación con las especies ya nombradas, hay un fuerte argumento para nombrarlas como especies separadas. Si las poblaciones son fenotípicamente fuertemente divergentes con poca separación genómica, entonces hay un juicio sobre si esas diferencias fenotípicas son (i) hereditarias y (ii) probables de causar aislamiento reproductivo sustantivo. Las exploraciones genómicas pueden resolver tales situaciones mediante pruebas de flujo génico restringido. El problema más difícil es dónde se detectan poblaciones morfológicamente similares, pero genéticamente divergentes. Estas poblaciones pueden diagnosticarse fácilmente como linajes evolutivos separados mediante la aplicación de métodos de delimitación estadística a datos multilocus (Carstens et al., 2013; Rannala, 2015), pero ¿son poblaciones efímeras o especies duraderas (Sukumaran y Knowles, 2017)? Ciertamente es posible, dentro de las reglas de nomenclatura, reconocer especies a partir de secuencias de ADN por sí solas, proporcionando diagnósticos basados en secuencias (Tautz et al., 2003; Cook et al., 2010). Por ejemplo, Murphy et al. (2015) delimitan y describen seis especies de anfípodos de primavera del desierto australiano en base a datos de secuencias de ADN, a pesar de que no hay diferencias morfológicas claras. Sin embargo, este es un área general de mejora donde los sistemáticos necesitan aplicar el término «especies crípticas» con mayor consistencia y rigor (Struck et al., 2017).
En situaciones en las que los datos de la secuencia de ADN proporcionan una característica definitoria clave potencial para el reconocimiento de una especie, sugerimos que es especialmente importante aplicar otras líneas de evidencia para evitar el sobreespliegue (Oliver et al., 2015). Un enfoque consiste en probar el aislamiento reproductivo sustancial donde dichas poblaciones forman zonas de contacto natural. Por ejemplo, en un análisis genómico comparativo de zonas de contacto entre pares de linajes de lagartos Carlia que son fenotípicamente indistinguibles, los índices de aislamiento genético se correlacionaron fuertemente con el tiempo de divergencia (Singhal y Moritz, 2013; Singhal y Bi, 2017) (Figura 2). Los pares de linajes más divergentes han sido reconocidos formalmente como especies separadas, mientras que los linajes más estrechamente relacionados (aunque estadísticamente diagnosticables) con evidencia de intercambio genético extenso genómicamente a través de zonas de contacto no lo fueron (Singhal et al., 2018). Dada la fuerte relación empírica entre la divergencia y el aislamiento reproductivo en estos taxones, estos autores separaron aún más dos aislados alopátricos y crípticos (de una especie estrechamente relacionada) que tenían una divergencia genómica aún mayor que los delimitados anteriormente. Extendiendo esta métrica informada de aislamiento reproductivo a otras especies congéneres con linajes filogeográficos profundamente divergentes pero morfológicamente similares (Potter et al., 2016, 2018; Afonso Silva et al., 2017a) sugiere que aún quedan más especies por describir, especialmente en las islas (Figura 2, Carlai amax y C. rufilatus), y de hecho, algunas de estas especies han sido descritas siguiendo un análisis fenotípico complementario(Afonso Silva et al., 2017b). Otros linajes con divergencias más recientes, pero aún delimitados estadísticamente como linajes separados utilizando un gran conjunto de genes nucleares, pueden representarse como Unidades Evolutivamente Significativas (UES) dentro de las especies.
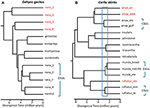
la Figura 2. Phylogenomic resolution of species boundaries in lizards from the Australian Monsoonal Tropics (AMT). En cada caso, las relaciones y los tiempos de divergencia relativos se estiman a partir de 100 exones utilizando la coalescente multiespecie implementada en StarBEAST2, pero las escalas de tiempo entre los dos ejemplos no son necesariamente las mismas. A) Resultados del grupo nana de Gehyra geckos (Moritz et al., 2018). La diversidad filogenética de linajes dentro de G. nana, como se reconoció anteriormente, incluyó otras cuatro especies ya descritas y morfológicamente distintivas. Los análisis posteriores de superposiciones y morfología del área de distribución han llevado al reconocimiento de cada uno de los linajes más divergentes filogenéticamente (etiquetas de taxones rojos) como especies separadas (Doughty et al., 2018). Los linajes (morfológicamente inseparables) que permanecen en G. nana, tal como se reconocen ahora, pueden considerarse como ESUs. B) Resolución en curso de los límites de las especies en los zancudos de Carlia del TMA. Algunos linajes ya han sido reconocidos como especies en base a una combinación de delimitación de especies genéticas e identificación post hoc de diferencias morfológicas diagnósticas (C. insularis y C. isostriacantha; Afonso Silva et al., 2017b). Para otros complejos (Potter et al., 2016, 2018), las revisiones taxonómicas están en curso. El rectángulo azul indica el rango de tiempos de divergencia donde se ha observado un aislamiento reproductivo sustancial, correlacionado con la divergencia, en zonas de contacto entre linajes morfológicamente crípticos de especies relacionadas (Singhal y Bi, 2017). Conservadoramente, y siguiendo (Singhal et al., 2018), reconocemos como especie candidata tres linajes genéticos con divergencias superiores a este umbral empírico. Esto dejaría ESUs genéticamente distintos dentro de cada Carlia amax, Carlia munda y Carlia rufilatus (corchetes azules). Las barras de escala representan estimaciones del tiempo de divergencia en millones de años.
En casos extremos, los métodos genómicos revelan que una sola especie taxonómica no solo es genéticamente heterogénea, sino que, en un sentido filogenético, también contiene otras especies nombradas. Tal «parafilia» puede surgir a través de especiaciones recientes de una sola especie, genéticamente subdividida (Patton y Smith, 1994), pero también genera la hipótesis de que el taxón genéticamente heterogéneo puede representar múltiples especies. Una vez más, se necesitan más pruebas. Como ejemplo, el análisis filogenómico de un complejo de lagartos Gehyra del norte de Australia delimitó nueve linajes evolutivos, con cuatro especies ya nombradas incrustadas filogenéticamente entre ellos (Moritz et al., 2018; Figura 2). La evidencia posterior de superposición geográfica (conservando la separación filogenética) y sutiles diferencias morfológicas respaldaron el nombramiento de los cuatro linajes más divergentes filogenéticamente (Doughty et al., 2018). Por el contrario, los linajes más estrechamente relacionados (aunque estadísticamente separables) tenían distribuciones adyacentes en lugar de superpuestas, pero no eran morfológicamente diagnosticables y, en consecuencia, se conservaron como una sola especie (G. nana sensu stricto), aunque con múltiples UDE, a la espera de una investigación adicional (Figura 2). Las exploraciones genómicas posteriores de muestras a través de los límites de estas UDE revelaron que al menos algunas tienen un intercambio genético sustancial, validando el enfoque conservador original adoptado para reconocer las especies.
En otros casos, los estudios genómicos de plantas, incluso a través de una simple ordenación de SNP, pueden revelar divergencias marcadas e imprevistas entre las muestras de población, lo que sugiere la presencia de taxones crípticos que luego se pueden analizar para determinar los caracteres fenotípicos diagnósticos. Por ejemplo, la evidencia basada en SNP para la divergencia genómica entre poblaciones y las diferencias ecotípicas en Eucalyptus salubris reveló dos linajes moleculares distintos que mantuvieron su carácter distintivo en un área de superposición geográfica, con la adaptación ecotípica considerada un factor importante para minimizar el flujo génico entre los linajes. (Steane et al., 2015; Figura 3). En otro caso, en el Pelargonio australiano, el análisis genómico, combinado con estudios morfológicos y reproductivos, ha llevado a cambios significativos en la taxonomía, tanto en la división como en el agrupamiento de taxones, especialmente cuando se encuentra que los rasgos morfológicos utilizados anteriormente son lábiles y discordantes con la filogenia (Nicotra et al., 2016).
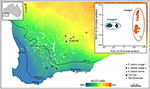
la Figura 3. El análisis genómico utilizando datos de SNP identificó linajes crípticos en el Eucalyptus salubris. La ubicación de las poblaciones de muestras de cada linaje se muestra a través de un gradiente de índice de aridez en el suroeste de Australia Occidental. El PCoA basado en distancias genéticas binarias derivadas de 16.122 marcadores de DArTseq, muestra dos linajes distintos. Adaptado de Steane et al. (2015).
Los casos anteriores ilustran el enfoque de prueba de hipótesis progresiva que se ha utilizado en la taxonomía integradora(Padial et al., 2010; Yeates et al., 2011). Además, estos casos utilizan alguna forma de secuenciación de reducción del genoma para obtener datos de alta calidad de 1000 genes independientes, lo que da mucha más confianza en los resultados que en el caso de usar solo loci de plástico o algunos genes nucleares. Un ejemplo de cómo la aplicación de la taxonomía integrativa puede alterar drásticamente las percepciones de la diversidad proviene de un estudio genético, acústico y morfológico combinado de ranas de Madagascar que casi duplicó la diversidad de especies conocida(Vieites et al., 2009). Estos autores propusieron una serie de términos que reflejan el estado de resolución y congruencia taxonómica: Especies Candidatas confirmadas, para taxones genéticamente y fenotípicamente divergentes, pero aún no nombrados; Especies Candidatas no confirmadas para taxones genéticamente divergentes donde la morfología y la acústica aún no se habían examinado; y Linajes profundamente Conespecíficos para entidades conocidas por ser genéticamente divergentes pero fenotípicamente crípticas. Los análisis genómicos aún no se han aplicado a estos taxones, pero seguramente informarían las decisiones sobre linajes conespecíficos vs.especies candidatas.
Los enfoques genómicos, incluso cuando se aplican de forma conservadora, sin duda conducirán a una mayor división de taxones morfológicamente conservadores. Pero, a la inversa, hay ejemplos claros en los que los mismos enfoques revelan especies sobre-divididas o diagnosticadas incorrectamente (Nicotra et al., 2016). Por supuesto, ya hay muchos estudios moleculares en complejos de especies de plantas y animales basados en conjuntos de datos a menor escala (p. ej., microsatélites, ADNmt y ADNcp) que han identificado dónde se han diagnosticado erróneamente especies, o dónde se han circunscrito taxonómicamente varias especies, pero ahora se consideran una sola especie. Por ejemplo, Rabosky et al. (2014) utilizaron filogenias de genes mitocondriales y nucleares para revisar el complejo scincid Ctenotus inornatus, lo que resultó en una reducción neta de especies y un realineamiento de los límites de las especies; los rasgos morfológicos aplicados anteriormente, aunque convenientes para los biólogos de campo, eran altamente lábiles y en gran medida no coincidían con los límites de las especies según se diagnosticaron genéticamente. En las plantas, algunos de los ejemplos más conocidos se encuentran en las orquídeas, como el género de orquídeas europeo Ophrys y el género de orquídeas australianas Caladenia, donde los estudios moleculares indican un número significativamente menor de entidades que serían reconocidas como especies, que las basadas en rasgos morfológicos y síndromes de polinización pseudocopulatoria (ver Devey et al., 2008; Swarts et al., 2014).
Los datos a escala genómica, cuando se interpretan de forma conservadora e ideal con análisis paralelos de fenotipos, continuarán conduciendo a cambios taxonómicos, especialmente en taxones morfológicamente conservadores (Fišer et al., 2018). Pero, en contraste con las preocupaciones expresadas por algunos (Garnett y Christidis, 2017), consideramos que esta es la búsqueda adecuada de la sistemática evolutiva, incluso si los resultados son temporalmente inconvenientes.
Genómica y Unidades de Conservación Intraespecíficas
Aunque menos polémicas que la delimitación taxonómica y el debate a nivel de especie, pero de muchas maneras igual de complejas, ha sido la discusión significativa sobre la definición y delimitación de las unidades de conservación como elementos importantes de la diversidad intraespecífica que necesitan consideración en las acciones de conservación (Ryder, 1986; Waples, 1991; Dizon et al., 1992; Moritz, 1994; Vogler y de Salle, 1994; Crandall et al., 2000; Fraser y Bernatchez, 2001; Avise, 2004; Funk et al., 2012) (gráfico 4). Dado el enfoque cauteloso para reconocer especies a partir de la evidencia genómica que defendemos anteriormente (y ver también Oliver et al., 2015; Singhal et al., 2018), es inevitable que algunas especies taxonómicas aún contengan poblaciones aisladas largas dentro de ellas. Se han propuesto conceptos como Unidades Evolutivamente Significativas (UES) o Unidades Designables (UDS) para complementar la taxonomía existente (Ryder, 1986; Waples, 1991; Moritz, 1994; Mee et al., 2015). Al igual que con las especies, las interpretaciones y los criterios varían, pero la intención común es reconocer los elementos principales de la diversidad intraespecífica para las acciones de conservación (Funk et al., 2012). Moritz (1994, 2002) abogó por un enfoque molecular simplista pero práctico para identificar segmentos en evolución independiente dentro de las especies, el eje «histórico» de la diversidad; mientras que otros pusieron más énfasis en el eje «adaptativo» para incluir la diversidad funcional (Ryder, 1986; Crandall et al., 2000; Fraser y Bernatchez, 2001; Waples et al., 2001; Mee et al., 2015) (Figura 4). Cualesquiera que sean los méritos relativos de estas alternativas, está claro que las herramientas técnicas y analíticas de la genómica de poblaciones ahora pueden identificar con solidez ambos componentes de la diversidad intraespecífica (Funk et al., 2012; Hoban et al., 2013; Whitlock, 2014; Yang y Rannala, 2014).
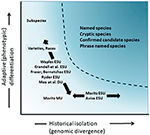
la Figura 4. Separación de la diversidad genética en dos componentes: variación adaptativa que surge a través de la selección natural, y ensayada a través del análisis de fenotipos y aislamiento histórico o divergencia neutra evaluada a través de divergencia genómica. El área sombreada más oscura por encima de la curva indica condiciones en las que es probable que las poblaciones se consideren como especies separadas en la mayoría de los conceptos. Se muestran varios conceptos de unidades de conservación y categorías taxonómicas infraespecíficas en relación con los dos ejes de la diversidad genética (después de Moritz, 2002).
Esto lleva a la pregunta: ¿por qué no nombrar a los ESUs como subespecies? Las subespecies han sido tradicionalmente definidas y vistas por botánicos y zoólogos como conjuntos fenotípicamente distintos, alopátricos de poblaciones que pueden cruzarse entre sí en límites geográficos (Mayr, 1942; Grant, 1981). Como tales, potencialmente reflejan la divergencia adaptativa sin necesariamente haber estado históricamente aislados. Las designaciones de subespecies son ampliamente utilizadas en las plantas, siendo las diferencias geográficas y ecológicas el principal medio por el que se distinguen (Hamilton y Reichard, 1992). En la medida en que las subespecies manifiesten adaptación local, con o sin flujo génico continuo, sugerimos que las subespecies fenotípicamente definidas continúen siendo reconocidas en conservación, y no mezcladas con EEU, al menos no como se define para representar poblaciones históricamente aisladas.
De la misma manera que la genómica de la población proporciona una precisión mucho mayor en la identificación de las UDE al utilizar loci neutros y adaptativos, también proporciona un medio para utilizar un gran número de loci neutros para delimitar más fácilmente las Unidades de Manejo (MUs) como unidades demográficamente independientes caracterizadas por un flujo génico restringido (Funk et al., 2012). Si bien la mayor parte de nuestro enfoque aquí se centra en unidades de ESU y unidades análogas, como DHe, que pueden ser reconocidas como las principales unidades intra-específicas para la conservación y han sido aceptadas para protección en algunas jurisdicciones, también se han identificado unidades MU que representan conjuntos de poblaciones demográficamente independientes (ver Palsbøll et al., 2007) que pueden ser manejados para retener la ESU más grande (Moritz, 1994; Funk et al., 2012). Las unidades de gestión pueden ser importantes para el monitoreo y la gestión de las poblaciones de especies y también se han denominado «poblaciones», particularmente relevantes para la gestión de la pesca (Avise, 2004; Laikre et al., 2005). La definición de MUs por Moritz (1994) como «poblaciones con divergencia significativa de frecuencias alélicas en loci nucleares o mitocondriales, independientemente de la distinción filogenética de los alelos» es generalmente aceptada, aunque, como señaló Palsbøll et al. (2007), la conectividad demográfica es el factor clave, no el nivel de flujo genético histórico.
Delimitar las unidades de conservación como elementos importantes de la diversidad intraespecífica también tiene implicaciones significativas para el rescate genético de pequeñas poblaciones endogámicas a través de la restauración del flujo genético o por aumento, y cada vez es más claro que el rescate genético debe considerarse de manera más amplia si se quiere evitar una mayor extinción de la población (Love Stowell et al., 2017; Ralls et al., 2018). Moritz (1999) propuso que mezclar MUs pero no ESUS podría considerarse una estrategia apropiada para el rescate genético. Como enfatizaron Frankham et al. (2012) la mezcla de especies taxonómicamente reconocidas generalmente no sería considerada aceptable por los administradores, en cuyo caso el sobreexplotamiento taxonómico podría restringir las oportunidades de rescatar pequeñas poblaciones endogámicas. Sugerimos que, si bien un enfoque más riguroso para delimitar unidades intraespecíficas, como las MUs, podría ayudar a aliviar este problema, también está claro que la estimación genómica de las tasas de migración y de divergencia adaptativa tiene el potencial de proporcionar una orientación mucho más sólida no solo para definir los límites de las especies y la variación intraespecífica, sino también para diseñar estrategias de rescate genético.
Filogenómica y Conservación
La genómica combinada con métodos filogenéticos puede resolver fácilmente las relaciones y la distancia evolutiva entre taxones, así como delimitar especies. Esto se logra mejor utilizando métodos de árboles de especies, en lugar de la concatenación, ya que los primeros explican la heterogeneidad de los árboles genéticos y evitan el inflado excesivo de las longitudes de las puntas debido al polimorfismo ancestral (Edwards et al., 2016; Ogilvie et al., 2016). Si bien los números masivos de loci a menudo no son esenciales, incluir más loci a menudo mejora la resolución de las relaciones y las longitudes de rama (por ejemplo, Blom et al., 2016). La importancia para la conservación es doble. En primer lugar, las evaluaciones filogenéticas permiten considerar el carácter distintivo de las especies o UDE, no solo si se describen. Por lo tanto, el programa EDGE (Isaac et al., 2007) aboga por la ponderación de las especies para la conservación basada en la cantidad de historia evolutiva única que representan, además de los criterios habituales de la UICN. En segundo lugar, las longitudes de rama en sí mismas pueden representar valores de biodiversidad, independientemente de la taxonomía (Faith, 1992), lo que ha demostrado su valor al priorizar áreas para la conservación (Laicado et al., 2015; Rosauer et al., 2016, 2018). En este sentido, la adopción de métricas filogenéticas utilizando medidas como la diversidad filogenética y el endemismo filogenético permiten resaltar áreas de alta diversidad evolutiva que pueden ser objeto de protección y recibir alta prioridad en cualquier proceso de planificación para mejorar la conservación de la biodiversidad. Este enfoque puede mejorar la protección de la diversidad genética y los procesos evolutivos para diversos grupos de organismos en una amplia gama de escalas geográficas (Laicado et al., 2015) tomar decisiones sobre áreas a priorizar más sólidas a los caprichos de cómo la evidencia genómica se traduce en el reconocimiento de especies o unidades intra específicas.
Las Definiciones legislativas del Mundo real
Los marcos normativos y legislativos para la conservación de la biodiversidad abarcan desde convenciones y estrategias internacionales, pasando por estrategias y legislación nacionales, y legislación y estrategias estatales o regionales dentro de los países. Los principales instrumentos internacionales sobre conservación biológica son el Convenio sobre la Diversidad Biológica, aplicado por conducto de la Convención de las Partes (COP), la Convención sobre el Patrimonio Mundial, la Estrategia Mundial de Biodiversidad, el Proceso de Montreal para la Conservación de los Bosques, la Convención de Ramsar sobre los Humedales, la Convención sobre las Especies Migratorias, la Convención sobre el Comercio Internacional de Especies Amenazadas de Fauna y Flora Silvestres (CITES), la lista roja de especies amenazadas de la Unión Internacional para la Conservación de la Naturaleza (UICN) y la convención Marco de las Naciones Unidas sobre el cambio climático. Todas estas cartas encarnan el principio de la conservación de las especies y la diversidad genética como objetivo básico de la conservación de la biodiversidad, siendo las especies la unidad ampliamente definida para la protección. Sin embargo, las definiciones de especies en la legislación y las directivas internacionales son extremadamente variables. Incorporan el reconocimiento taxonómico de especies y taxones intraespecíficos, pero también pueden incorporar conceptos genéticos/evolutivos que a menudo están cubiertos por la definición legal de «especie» (Tabla 1).

Cuadro 1. Legislación y directivas mundiales que definen «especies» para su inclusión en la lista y protección.
Dada la amplia gama de niveles infraespecíficos que pueden protegerse (cuadro 1), la legislación y los procesos de inclusión de nombres en las listas correspondientes de los países deberían facilitar la conservación de la diversidad genética. Por ejemplo, la definición de «especie», cuando se considera la legislación en todo el mundo (Tabla 1), puede incluir: especies, subespecies, variedades y poblaciones geográficas y/o genéticamente distintas, mientras que bajo la legislación sudafricana también puede incluir cultivares, razas geográficas, cepas e híbridos. Por lo tanto, en la mayoría de las legislaciones a nivel mundial, una «especie» incluye entidades que reflejan varios niveles infraespecíficos que forman parte de una distribución de especies más amplia y reflejan en diversos grados el continuo población-especie. Esto es paralelo a la inevitable dificultad que enfrentan los taxónomos, biólogos evolutivos y genetistas de conservación para encontrar una forma más consistente y uniforme de especificar unidades dentro de este continuo.
Algunos argumentan que las preocupaciones con respecto a las implicaciones políticas de las especies y la incertidumbre infraespecífica están fuera de lugar (Hey et al., 2003), pero seguimos opinando que, si bien la legislación puede proporcionar un mecanismo para proteger la diversidad genética en una variedad de niveles, corresponde a los biólogos proporcionar más claridad y coherencia sobre cómo deben delimitarse esos niveles desde las especies hasta las poblaciones individuales. En la actualidad, los administradores y legisladores se quedan con una desconcertante variedad de conceptos de especies y unidades infraespecíficas proporcionadas por los científicos al delimitar unidades a través del continuo población-especie. Por ejemplo, todas las jurisdicciones consideran que las especies reconocidas por los taxónomos son unidades clave para la conservación (Tabla 1), sin embargo, todavía existe un amplio debate entre los taxónomos en torno a los conceptos de especies, y la forma en que se diagnostican las especies en la práctica varía dentro de los grupos y entre ellos. Esto ha llevado a un llamamiento reciente para un enfoque más regulado de la taxonomía (Garnett y Christidis, 2017), a lo que otros han respondido que la regulación de la taxonomía no es la respuesta (Thomson et al., 2018). Aunque estamos de acuerdo con algunos de los puntos hechos por Thomson et al. (2018) sugerimos que es bastante ingenuo proponer que la propia legislación necesita cambiar para hacer referencia explícita al concepto de taxón específico que implica un nombre. Estos y otros debates anteriores destacan la necesidad de que los biólogos y taxónomos evolutivos tomen la iniciativa para garantizar la claridad (transparencia) en la forma en que traducen la evidencia genómica y de otro tipo a la delimitación de especies o unidades intraespecíficas, y cómo estos taxones se relacionan con la legislación pertinente para la conservación y protección.
Traducción a Manejo
Como se mencionó anteriormente, todas las leyes y directivas reconocen el nivel de especie como la unidad fundamental de conservación, pero también prevén el reconocimiento de varias unidades infraespecíficas. Muchos también tienen un requisito implícito para el nombramiento formal y la descripción taxonómica de las especies cuando se consideran para la priorización de la conservación y la protección formal. Por ejemplo, en virtud de la Lista Roja de la UICN Directrices listado de especies no identificadas se desanime, y hay un requisito de que la nueva descripción de la especie será publicado dentro de los cuatro años de las especies incluidas en la Lista Roja de la UICN o sería eliminado. Esto destaca la importancia de nombrar y describir taxonómicamente las entidades, tanto a nivel de especie como infraespecífico, la información taxonómica limitada para muchos grupos de plantas y animales (Mace, 2004; Oliver y Lee, 2010; Wilson, 2017), y la necesidad de acelerar el esfuerzo taxonómico, particularmente en áreas ricas en biodiversidad (Riedel et al., 2013; Wege et al., 2015; Wilson, 2017).
Las especies taxonómicamente nombradas y las categorías taxonómicas intraespecíficas, como subespecies y variedades, generalmente proporcionan las unidades básicas para la inclusión formal en la lista, la priorización de la conservación y la protección, sin embargo, en algunas jurisdicciones se reconocen formalmente otras categorías de unidades de conservación intraespecíficas. Estos se identifican generalmente utilizando datos genómicos y fenotípicos combinados. Por ejemplo, la Ley de Especies en Peligro de los Estados Unidos reconoce Segmentos de Población Distintos (DPSs) que se basan ampliamente en UDE (Waples, 1991), mientras que la Ley de Especies en Riesgo del Canadá reconoce los DHE. Una interpretación operativa de» poblaciones distintas » en virtud de la Ley de Especies en Riesgo de Canadá, DHe tiene como objetivo identificar conjuntos discretos de poblaciones que representan componentes importantes del legado evolutivo de las especies y que es poco probable que se reemplacen a través de la dispersión natural (Mee et al., 2015).
Tanto DHe como DPSs se superponen conceptualmente con UDE (Waples et al., 2013) y se han aplicado más ampliamente a los peces de América del Norte, incluidos los salmónidos del noroeste de los Estados Unidos (Waples, 1995) y los coregónidos canadienses (Mee et al., 2015) Curiosamente, los DPSS en los EE.UU. solo se aplican a los vertebrados, mientras que en Canadá los DHe se pueden aplicar a cualquier organismo que pueda enumerarse en su Ley (ver Waples et al., 2013), con DHe actualmente en la lista de artrópodos, moluscos y plantas. En Australia, al igual que en Canadá, se pueden incluir poblaciones distintas para cualquier organismo reconocido en virtud de la Ley de Protección del Medio Ambiente y Conservación de la Biodiversidad de 1999 (Ley EPBC), aunque no son una categoría específicamente nombrada, como DUSs y DPSs. Sin embargo, hasta la fecha solo se han reconocido para vertebrados. En los EE.UU. hay 127 DPSs de vertebrados enumerados para su conservación (https://ecos.fws.gov/ecp/); en Canadá se han incluido 70 DHe que abarcan todos los organismos (http://www.registrelep-sararegistry.gc.ca/species/schedules_e.cfm?id=1); y en Australia, se ha reconocido que las poblaciones de 7 especies de vertebrados que abarcan mamíferos, peces y aves tienen un estado de conservación específico en virtud de la Ley EPBC (http://www.environment.gov.au/cgi-bin/sprat/public/publicthreatenedlist.pl). Mientras que las evaluaciones de estas poblaciones en Estados Unidos y Canadá generalmente usan información genética(Waples et al., 2013), en Australia suelen basarse en la identificación geográfica y jurisdiccional, aunque en algunos de estos casos han sido respaldados por el análisis de diferenciación genética para mamíferos (Firestone et al., 1999; Armstrong, 2006) y fish (Colgan y Paxton, 1997; Ahonen et al., 2009).
La lista de unidades de conservación infraespecíficas para plantas e invertebrados en Canadá (DHe), pero no en los Estados Unidos o Australia, pone de relieve otra cuestión no relacionada específicamente con la delimitación de las unidades de conservación, sino más ampliamente con inconsistencias significativas en la priorización de la conservación de la biodiversidad; vertebrados icónicos como aves, mamíferos y peces son un foco en muchas jurisdicciones, mientras que otros organismos no lo son. Esta lista preferencial de poblaciones de ciertos organismos refleja una serie de cuestiones que tienen menos que ver con la ciencia de delimitar unidades de conservación y más que ver con la lista de poblaciones basada en la conciencia pública, la conveniencia política y la naturaleza carismática del organismo que se va a incluir (ver Waples et al., 2013).
Mientras que en Australia algunas poblaciones de mamíferos, aves y peces bien conocidos están protegidas bajo la Ley EPBC, ahora hay un cuerpo significativo de información basada en estudios moleculares en reptiles, invertebrados y plantas donde las poblaciones pueden ser reconocidas como ESUs o MUs, y muchas de estas son de importancia para la conservación debido a su estrecho rango geográfico, pequeño tamaño de población, tasa de declive y susceptibilidad a procesos amenazantes. Por ejemplo, se pueden encontrar ejemplos llamativos de poblaciones altamente estructuradas y linajes profundamente divergentes dentro de las especies que se ajustarían fácilmente a los requisitos de una UDE en una variedad de vertebrados de baja dispersión de los trópicos (Figura 2) y en otros lugares. De manera similar, en invertebrados australianos, como anfípodos y milpiés, la estructura genética significativa y los patrones filogeográficos apoyan la delimitación de grupos de población discretos como UDE que son geográficamente dispares, con rangos geográficos estrechos, restringidos a hábitats específicos y representados por solo unas pocas poblaciones (Finston et al., 2007; Murphy et al., 2013; Nistelberger et al., 2014a). A menudo, estos endémicos de corto alcance son taxones de interés para la conservación (Harvey et al., 2011) y se ubican en puntos calientes de endemismo (por ejemplo, Oliver et al., 2017), que son en sí mismas altas prioridades para la atención de conservación de múltiples taxones (Rosauer et al., 2018), como la Región de Kimberley y la Cordillera Stirling en el suroeste de Australia Occidental.
En las plantas australianas también hay un número significativo de estudios genéticos y filogeográficos de población que identifican grupos de población distintos dentro de un rango de especies de diferentes géneros que reflejan sistemas de población disjuntos e históricamente aislados, complejidades geológicas y edáficas, y ocupan hábitats contrastantes en términos de vegetación y clima (ver Broadhurst et al., 2017). Las poblaciones genéticamente distintas son particularmente evidentes en especies del suroeste de Australia occidental (Coates, 2000; Byrne et al., 2014) con una serie de estudios que destacan la estructura genética significativa y los linajes altamente divergentes en una variedad de géneros y familias de plantas que incluyen: Acacia (Mimosaceae; Coates, 1988), Atriplex (Amaranthaceae; Shepherd et al., 2015), Banksia (Proteaceae; Coates et al., 2015), Lambertia (Proteaceae; Byrne et al., 2002), Hakea (Proteaceae; Sampson et al., 2014), Eremophila (Scrophulariaceae; Llorens et al., 2015), Pultanaea (Fabaceae; Millar y Byrne, 2013), y Calothamnus (Myrtaceae; Nistelberger et al., 2014b). En muchos casos, estas poblaciones son reconocidas en la planificación de la conservación por las agencias gubernamentales estatales y se les da consideración en las evaluaciones de impacto ambiental (ver Shepherd et al., 2015) a pesar de que no están incluidos en la lista de protección de la legislación. Sin embargo, reconocemos que el reconocimiento formal de estas unidades mejoraría su conservación a largo plazo.
Estos ejemplos australianos destacan la creciente necesidad de reconocer esta capa de diversidad genética por debajo del nivel de especie. El desafío es asegurar que las poblaciones altamente estructuradas y los linajes profundamente divergentes que son motivo de preocupación para la conservación estén protegidos frente a las demandas competitivas de atención a la conservación. Más allá de las especies icónicas, este desafío también es evidente a nivel mundial, con la excepción de Canadá. La falta de protección de unidades de conservación claramente delimitadas probablemente aumentará el riesgo de extinción críptica y pérdida de diversidad genética significativa, que en muchos casos puede involucrar linajes y poblaciones históricamente aisladas que han persistido dentro de las especies durante millones de años (Moritz, 2002).
Conclusiones y Recomendaciones
La inclusión de especies y diversas categorías infraespecíficas en la legislación sobre especies amenazadas o en organizaciones internacionales como la UICN, indica un compromiso de proteger la diversidad genética en todo el espectro de poblaciones de especies. Sin embargo, los enfoques actuales para la conservación de la biodiversidad se centran en gran medida en el extremo de las especies de este continuo, con menos atención a la diversidad genética y la variación infraespecífica. Este enfoque más limitado en la variación genética a nivel intra-especie sugiere que, si bien los administradores de conservación reconocen cada vez más el valor de considerar explícitamente la información genética en las acciones de manejo y la priorización, a menudo carecen de información procesable de los científicos de la biodiversidad (Waples et al., 2008; Laikre et al., 2010; Cook y Sgrò, 2017; Taylor et al., 2017). Esta brecha entre la ciencia y la práctica de la conservación impide un enfoque global más eficaz para conservar y gestionar la diversidad genética.
Sugerimos que la falta de claridad de los científicos de biodiversidad sobre las unidades de conservación se basa en gran medida en inconsistencias en dos niveles. El primero se refiere a los conceptos de múltiples especies y los diversos métodos de diagnóstico utilizados para delimitar especies que crean inconsistencia entre los grupos taxonómicos. La segunda se refiere a la falta de coherencia y acuerdo por parte de taxónomos, biólogos evolutivos y biólogos conservacionistas sobre cómo tratar taxones infraespecíficos y otras entidades como ESU, DHe y MUs.
Si bien consideramos que la legislación a menudo ya es suficientemente flexible en términos de cómo se definen los taxones (o «especies»), argumentamos que existe la necesidad de abordar sistemáticamente el continuo de especies de población y definir más claramente las unidades para la conservación con un mayor reconocimiento de conceptos como las UDE y cómo se relacionan con la legislación pertinente. Enfatizamos que este tema sin duda se volverá más crítico dado el poder de la genómica para detectar la diferenciación a escalas progresivamente más finas y su creciente uso para delimitar especies y entidades infraespecíficas.
Del análisis anterior, ofrecemos las siguientes recomendaciones:
(i) Los datos genómicos deben interpretarse de manera conservadora al delimitar especies, utilizando un enfoque taxonómico integrador que típicamente incluye evidencia más allá de los datos genéticos solos.
(ii) Cuando los genetistas identifiquen múltiples linajes o poblaciones distintas dentro de una especie, y no justifiquen su elevación al nivel de especie y no estén reconocidos taxonómicamente, que haya un mayor impulso y un enfoque más uniforme para identificar unidades intraespecíficas claramente justificadas para la evaluación práctica del estado de conservación o las evaluaciones de impacto, y que estas se reconozcan explícitamente como unidades para la conservación en el contexto de la legislación pertinente a esa jurisdicción.
(iii) La UICN, a través de su Grupo de Especialistas en Genética de la Conservación y sus subgrupos regionales, considerará la posibilidad de elaborar directrices para reconocer y etiquetar unidades intraespecíficas que se ajusten a las mejores prácticas científicas.
(iv) La visión más matizada que surge de la genómica evolutiva de las especies y el continuo de las poblaciones a las especies debe ser comunicada de manera más efectiva por los científicos a los administradores de conservación y los responsables políticos.
En términos más generales, nos hacemos eco de las recomendaciones hechas recientemente en varias revisiones (Frankham, 2010; Santamaría y Méndez, 2012; Hoban et al., 2013; Moritz y Potter, 2013; Laicos et al., 2015; Pierson et al., 2016; Cook y Sgrò, 2017; Taylor et al., 2017) que destacan la necesidad de que los científicos mejoren la comunicación y creen conciencia con los responsables de la formulación de políticas, las agencias gubernamentales, las organizaciones no gubernamentales y los profesionales que los resultados de la sistemática molecular, la taxonomía molecular, la filogeografía y la investigación genética de poblaciones pueden proporcionar información crítica sobre la conservación de la diversidad genética y la gestión de los procesos evolutivos.
Las contribuciones de los autores
DC, MB y CM contribuyeron a la preparación y revisión del manuscrito.
Declaración de Conflicto de Intereses
Los autores declaran que la investigación se realizó en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un posible conflicto de intereses.
Agradecimientos
Este proyecto fue apoyado por el Australian Research Council (ARC Linkage Grant No. LP120200063), la Universidad Nacional de Australia y el Departamento de Biodiversidad, Conservación y Atracciones de Australia Occidental.
Afonso Silva, A.C., Bragg, J. G., Potter, S., Fernandes, C., Coelho, M. M., and Moritz, C. (2017a). Tropical specialist versus climate generalist: diversification and demographic history of sister species of Carlia skinks from northwestern Australia. Mol. Ecol. 26:4045–4058. doi: 10.1111 / mec.14185
Texto completo de CrossRef | Google Scholar
Afonso Silva, A.C., Santos, N., Ogilvie, H. A., and Moritz, C. (2017b). Validación y descripción de dos nuevas norte de Australia occidental arco iris eslizones con multiespecíficas coalescentes métodos y morfología. PeerJ 5: e3724. doi: 10.7717 / peerj.3724
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Ahonen, H., Harcourt, R. G., and Stow, A. J. (2009). El ADN nuclear y mitocondrial revela el aislamiento de poblaciones de tiburones nodriza grises en peligro (Carcharias taurus). Mol. Ecol. 18, 4409–4421. doi: 10.1111 / j. 1365-294X. 2009. 04377.x
Resumen de PubMed | Texto completo de CrossRef/Google Scholar
Armstrong, K. N. (2006). Phylogeographic structure in Rhinonicteris aurantia (Chiroptera: Hipposideridae): implications for conservation. Acta Chiropterologica 8, 63-81. doi: 10.3161/1733-5329(2006)82.0.CO
CrossRef Texto Completo | Google Scholar
Arnold, M. L. y Kunte, K. (2017). Adaptive genetic exchange: a tangled history of mixture and evolutionary innovation (en inglés). Tendencias Ecol. Evol. 32, 601–611. doi: 10.1016 / j.tree.2017.05.007
Resumen de PubMed / Texto completo Cruzado / Google Scholar
Avise, J.C. (2004). Marcadores Moleculares, Historia Natural y Evolución. 2nd Edn. Sunderland, MA; Oxford: Oxford University Press.
Google Scholar
Avise, J.C., Arnold, J., Ball, R. M., Bermingham, E., Lamb, T., Neigel, J. E., et al. (1987). Filogeografía intraespecífica: the mitochondrial DNA bridge between population genetics and systematics (en inglés). Ana. Apo. Ecol. Sys. 18, 489–522. doi: 10.1146/annurev.es.18.110187.002421
CrossRef Texto Completo | Google Scholar
Bickford, D., Lohman, D. J., Sodhi, N. S, Ng, P. K., Meier, R., Winker, K., et al. (2007). Especies crípticas como ventana a la diversidad y la conservación. Tendencias Ecol. Evol. 22, 148–155. doi: 10.1016 / j.tree.2006.11.004
Resumen de PubMed / Texto completo Cruzado / Google Scholar
Blom, M. P. K., Bragg, J. G., Potter, S., and Moritz, C. (2016). Accounting for uncertainty in gene tree estimation: summary-coalescent species tree inference in a challenging radiation of Australian lizards. Syst. Biol. 66, 352–366. doi: 10.1093 / sysbio / syw089
Resumen de PubMed / Texto completo de CrossRef/Google Scholar
Broadhurst, L., Breed, M., Lowe, A., Bragg, J., Catullo, R., Coates, D. J., et al. (2017). Diversidad genética y estructura de la flora australiana. Buzos. Distrib. 23, 41–52. doi: 10.1111 / ddi.12505
Texto completo de CrossRef / Google Scholar
Byrne, M., Coates, D. J., Forest, F., Hopper, S. D., Krauss, S.L., Sniderman, J. M. K., et al. (2014). «A diverse flora-species and genetic relationships,» in Plant Life on the Sandplains in Southwest Australia, a Global Biodiversity Hotspot, editors H. Lambers (Crawley, WA: University of Western Australia Publishing), 81-99.
PubMed Abstract
Byrne, M. Macdonald, B., y Coates, D. J. (2002). Phylogeographic patterns and high cpDNA diversity within the Acacia acuminata (Leguminosae:Mimosoideae). complejo en Australia Occidental. J. Evol. Biol. 15, 576–587. doi: 10.1046 / j. 1420-9101. 2002. 00429.x
CrossRef Full Text / Google Scholar
Carstens, B. C., Pelletier, T. A., Reid, N. M., and Satler, J. D. (2013). Cómo fallar en la delimitación de especies. Mol. Ecol. 22, 4369–4383. doi: 10.1111 / mec.12413
Resumen de PubMed / Texto completo Cruzado / Google Scholar
Coates, D. J. (1988). Diversidad genética y estructura genética de la población en la rara Acacia anomala Court. Aust. J. Bot. 36:273–286. doi: 10.1071/BT9880273
CrossRef Texto Completo | Google Scholar
Coates, D. J. (2000). Defining conservation units in a rich and fragmented flora: implications for management of genetic resources and evolutionary processes in south-west Australia plants. Aust. J. Bot. 48, 329–339. doi: 10.1071 / BT99018
Texto completo cruzado / Google Scholar
Coates, D. J., McArthur, S.L., and Byrne, M. (2015). Pérdida significativa de diversidad genética después de la extinción de la población impulsada por patógenos en la rara Banksia brownii endémica (Proteaceae). Biol. Conserv. 192, 353–360. doi: 10.1016 / j. biocon.2015.10.013
CrossRef Full Text / Google Scholar
Colgan, D. J., and Paxton, J. R. (1997). Biochemical genetics and recognition of a western stock of the common gemfish, Rexea solandri (Scombroidea: Gempylidae)., en Australia. Estropear. Freshw. Res. 48, 103 a 118. doi: 10.1071 / MF96048
CrossRef Full Text / Google Scholar
Convenio sobre la Diversidad Biológica (2007). Disponible en línea en: https://www.cbd.int/doc/meetings/cop-bureau/cop-bur-2007/cop-bur-2007-10-14-en.pdf
Cook, C. N., y Sgrò, C. M. (2017). Alinear la ciencia y la política para lograr una conservación evolutivamente ilustrada. Conserv. Biol. 31, 501–512. doi: 10.1111 / cobi.12863
Resumen de PubMed | Texto completo cruzado/Google Scholar
Cook, L. G., Edwards, R. D., Crisp, M. D., and Hardy, N. B. (2010). ¿Necesita que siempre se requiera morfología para las nuevas descripciones de especies? Invertir. Sys. 24, 322–332. doi: 10.1071/IS10011
CrossRef Texto Completo | Google Scholar
Cracraft, J. (1983). Conceptos de especies y análisis de especiación. Curr. Ornitol. 1, 159–187. doi: 10.1007/978-1-4615-6781-3_6
CrossRef Full Text / Google Scholar
Crandall, K. A., Bininda-Emonds, O. R., Mace, G. M., and Wayne, R. K. (2000). Considering evolutionary processes in conservation biology (en inglés). Tendencias Ecol. Evol. 15, 290–295. doi: 10.1016 / S0169-5347 (00)01876-0
Resumen de PubMed | Texto completo cruzado | Google Scholar
Cutter, A. D., and Payseur, B. A. (2013). Genomic signatures of selection at linked sites: unifying the disparity among species (en inglés). NAT. Reverendo Genet. 14, 262–274. doi: 10.1038/nrg3425
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Dayrat, B. (2005). Towards integrative taxonomy (en inglés). Biol. J. Linn. Soc. 85, 407–415. doi: 10.1111 / j. 1095-8312. 2005. 00503.x5
CrossRef Full Text/Google Scholar
de Queiroz, K. (1998). «The general lineage concept of species, species criteria, and the process of speciation,» in Forms: Species and Speciation, D. J. Howard,r S. H. Berlocher (New York, NY: Oxford University Press), 57-75.
Google Scholar
de Queiroz, K. (2007). Conceptos de especies y delimitación de especies. Syst. Biol. 56, 879–886. doi: 10.1080/10635150701701083
Resumen de PubMed | Texto completo cruzado/Google Scholar
Degnan, J. H., and Rosenberg, N. A. (2009). Discordancia de árboles genéticos, inferencia filogenética y coalescencia de especies múltiples. Tendencias Ecol. Evol. 24, 332–340. doi: 10.1016 / j.tree.2009.01.009
Resumen de PubMed | Texto completo cruzado/Google Scholar
Devey, D. S., Bateman, R. M., Fay, M. F., and Hawkins, J. A. (2008). Amigos o familiares? Phylogenetics and species delimitation in the controversial European orchid genus Ophrys. Ana. Robot. 101, 385–402. doi: 10.1093/aob / mcm299
Resumen PubMed / Texto completo cruzado / Google Scholar
Dizon, A. E., Lockyer, C, Perrin, W. F., Demaster, D. P., and Sisson, J. (1992). Rethinking the stock concept – a phylogeographic approach (en inglés). Conserv. Biol. 6, 24–36. doi: 10.1046 / j. 1523-1739. 1992. 610024.x
CrossRef Texto Completo | Google Scholar
Doughty, P., Bourke, G., Tedeschi, L., Pratt, R., Oliver, P., Palmer, R., et al. (2018). Species delimitation in the Gehyra nana (Squamata: Gekkonidae). complex: cryptic and divergent morphological evolution in the Australian Monsoonal Tropics, with the description of four new species. Zootaxa, 4403: 201-244. doi: 10.11646 / zootaxa.4403.2.1
Resumen de PubMed | Texto completo cruzado/Google Scholar
Dynesius, M., and Jansson, R. (2014). Persistence of Within-species linages: a neglected control of speciation rates. Evolución 68, 923-934. doi: 10.1111 / evo.12316
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Edwards, S. V., Potter, S., Schmitt, C. J., Bragg, J. G., and Moritz, C. (2016). Reticulation, divergence, and the phylogeography–phylogenetics continuumc. Proc. Natl. Acad. Sci. U. S. A. 113, 8025-8032. doi: 10.1073 / pnas.1601066113
Texto completo de CrossRef | Google Scholar
Faith, D. P. (1992). Evaluación de la conservación y diversidad filogenética. Biol. Desventaja. 61, 1–10. doi: 10.1016/0006-3207(92)91201-3
CrossRef Full Text | Google Scholar
Finston, T. L., Johnson, M. S., Humphreys, W. F., Eberhard, S. M., and Halse, S.A. (2007). La especiación críptica en dos géneros de anfípodos subterráneos muy extendidos refleja patrones históricos de drenaje en un paisaje antiguo. Mol. Ecol. 16, 355–365. doi: 10.1111 / j. 1365-294X. 2006. 03123.x
Resumen de PubMed | Texto completo cruzado/Google Scholar
Firestone, K. B., Elphinstone, M. S., Sherwin, W. B., y Houlden, B. A. (1999). Phylogeographical population structure of tiger quolls Dasyurus maculatus (Dasyuridae: Marsupalia), an endangerous carnivorous marsupial. Mol. Ecol. 8, 1613–1625. doi: 10.1046 / j. 1365-294x. 1999. 00745.x
CrossRef Texto Completo | Google Scholar
Fišer, C., Robinson, C. T., y Malard, F. (2018). Especies crípticas como una ventana al cambio de paradigma del concepto de especie. Mol. Ecol. 27, 613–635. doi: 10.1111 / mec.14486
Resumen de PubMed | Texto completo de CrossRef/Google Scholar
Frankham, R. (2010). Desafíos y oportunidades de los enfoques genéticos para la conservación biológica. Biol. Conserv. 143,1919–1927. doi: 10.1016 / j. biocon.2010.05.011
Texto completo de CrossRef | Google Scholar
Frankham, R., Ballou, J. D., Dudash, M. R., Eldridge, M. D., Fenster, C. B., Lacy, R. C., et al. (2012). Implicaciones de diferentes conceptos de especies para la conservación de la biodiversidad. Biol. Conserv. 153, 25–31. doi: 10.1016 / j. biocon.2012.04.034
CrossRef Full Text | Google Scholar
Fraser, D. J., and Bernatchez, L. (2001). Conservación evolutiva adaptativa: hacia un concepto unificado para definir unidades de conservación. Mol. Ecol. 10, 2741–2752. doi: 10.1046 / j. 1365-294X. 2001.t01-1-01411.x
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Funk, W. C., Mckay, J. K., Hohenlohe, P. A., and Allendorf, F. W. (2012). Aprovechamiento de la genómica para delinear unidades de conservación. Tendencias Ecol. Evol. 27, 489–496. doi: 10.1016 / j.tree.2012.05.012
Resumen de PubMed / Texto completo Cruzado / Google Scholar
Garnett, S. T., and Christidis, L. (2017). La anarquía taxonómica dificulta la conservación. Nature 546, 25-27. doi: 10.1038 / 546025a
Resumen de PubMed / Texto completo cruzado / Google Scholar
Gippoliti, S., Cotterill, F. P. D., Zinner, D. y Groves, C. P. (2018). Impactos de la inercia taxonómica para la conservación de la diversidad de ungulados africanos: descripción. Biol. Apo. 93, 115-130. doi: 10.1111 / brv.12335
Resumen de PubMed | Texto completo Cruzado/Google Scholar
Grant, V. (1981). Especiación de Plantas. New York, NY: Columbia University Press.
Resumen de PubMed/Google Scholar
Hamilton, C. W., y Reichard, S. H. (1992). Práctica actual en el uso de subespecies, variedades y formas en la clasificación de plantas silvestres. Taxon 41, 485-498. doi: 10.2307/1222819
CrossRef Texto Completo | Google Scholar
Harvey, M. S., Rix, M. G., Framenau, V. W., Hamilton, Z. R., Johnson, M. S., Teale, R. J., et al. (2011). Protegiendo a los inocentes: el estudio de taxones endémicos de corto alcance mejora los resultados de conservación. Invertir. Syst. 25, 1–10. doi: 10.1071 / IS11011
Texto completo cruzado / Google Scholar
Hey, J., Waples, R. S., Arnold, M. L., Butlin, R. K., and Harrison, R. G. (2003). Understanding and confronting species uncertainty in biology and conservation. Tendencias Ecol. Evol. 18, 597–603. doi: 10.1016 / j.tree.2003.08.014
Texto completo de CrossRef | Google Scholar
Hoban, S. M., Hauffe, H. C., Perez-Espona, S., Arntzen, J. W., Bertorelle, G., Bryja, J., et al. (2013). Llevar la diversidad genética a la vanguardia de la política de conservación y gestión. Conserv. Genet. Recurso. 5, 593–598. doi: 10.1007 / s12686-013-9859-y
CrossRef Full Text | Google Scholar
Isaac, N. J., Mallet, J., and Mace, G. M. (2004). Taxonomic inflation: its influence on macroecology and conservation (en inglés). Tendencias Ecol. Evol. 19, 464–469. doi: 10.1016 / j.tree.2004.06.004
Resumen de PubMed / Texto completo Cruzado / Google Scholar
Isaac, N. J., Turvey, S. T., Collen, B., Waterman, C., and Baillie, J. E. (2007). Mammals on the EDGE: conservation priorities based on threat and phylogeny (en inglés). PLoS ONE 2: e296. doi: 10.1371 / journal.ponga.0000296
Resumen de PubMed / Texto completo Cruzado / Google Scholar
Jones, M. R., and Good, J. M. (2016). Captura dirigida en genómica evolutiva y ecológica. Mol. Ecol. 25, 185–202. doi: 10.1111 / mec.13304
Resumen de PubMed | Texto completo cruzado/Google Scholar
Jorger, K. M., and Schrödl, M. (2013). ¿Cómo describir una especie críptica? Problemas prácticos de taxonomía molecular. Delantero. Zool. 10:59. doi: 10.1186/1742-9994-10-59
Resumen de PubMed / Texto completo de CrossRef/Google Scholar
Laikre, L., Allendorf, F., Aroner, L. C., Baker, S., Gregovich, D. P., Hansen, M. M., et al. (2010). Descuido de la diversidad genética en la aplicación del convenio sobre la diversidad biológica. Conserv. Biol. 24, 86–88. doi: 10.1111 / j. 1523-1739. 2009. 01425.x
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Laikre, L., Palm, S., and Ryman, N. (2005). Genetic population structure of fishes: implications for coastal zone management. Ambio 34,111-119. doi: 10.1579/0044-7447-34.2.111
Resumen de PubMed / Texto completo Cruzado/Google Scholar
Laicos, T., Laffan, S. W., González-Orozco, C. E., Faith, D. P., Rosauer, D. F., Byrne, M., et al. (2015). Phylodiversity to inform conservation policy: An Australian example (en inglés). Sci. Total Environ. 534, 131–143. doi: 10.1016 / j. scitotenv.2015.04.113
Resumen de PubMed / Texto completo Cruzado / Google Scholar
Leache, A. D., and Oaks, J. R. (2017). La utilidad del polimorfismo de nucleótido único (SNP). data in phylogenetics (en inglés). Ana. Apo. Ecol. Syst. 48, 69–84. doi: 10.1146/annurev-ecolsys-110316-022645
CrossRef Full Text | Google Scholar
Llorens, T. M., Macdonald, B., McArthur, S., Coates, D. J., and Byrne, M. (2015). Disjunct, highly divergent genetic lineages within two rare Eremophila (Scrophulariaceae: Myoporeae). species in a biodiversity hotspot: implications for taxonomy and conservation. Biol. J. Linn. Soc. 177, 96–111. doi: 10.1111/boj.12228
CrossRef Full Text | Google Scholar
Love Stowell, S. M., Pinzone, C. A., and Martin, A. P. (2017). Superar las barreras a las intervenciones activas para la diversidad genética. Biodivers. Conserv. 26, 1753–1765. doi: 10.1007 / s10531-017-1330-z
CrossRef Full Text/Google Scholar
Mace, G. M. (2004). The role of taxonomy in species conservation. Philos. Trans. R. Soc. B: Biol. Sci. 359, 711–719. doi: 10.1098 / rstb.2003.1454
Resumen de PubMed | Texto completo Cruzado/Google Scholar
Mallet, J. (2007). Especiación híbrida. Nature 446, 279-283. doi: 10.1038/nature05706
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Mayr, E. (1942). Systematics and the Origin of Species (en inglés). New York, NY: Columbia University Press.
Google Scholar
Mee, J. A., Bernatchez, L., Reist, J. D., Rogers, S. M., and Taylor, E. B. (2015). Identificación de unidades designables para priorización de conservación intraespecífica: un enfoque jerárquico aplicado al complejo de especies de peces blancos de lago (Coregonus spp.). Evol. Appl. 8, 423–441. doi: 10.1111 / eva.12247
PubMed Abstract | CrossRef Full Text | Google Scholar
Millar, M. A., and Byrne, M. (2013). Cryptic divergent lineages of Pultenaea pauciflora M.B. Scott (Fabaceae: Mirbelieae) exhibit different evolutionary history. Biol. J. Linn. Soc. 108, 871–881. doi: 10.1111/bij.12002
CrossRef Full Text | Google Scholar
Moritz, C. (1994). Defining evolutionarily significant units for conservation. Trends Ecol. Evol. 9, 373–375. doi: 10.1016/0169-5347(94)90057-4
CrossRef Full Text | PubMed Abstract
Moritz, C. (1999). Unidades de conservación y translocaciones: estrategias para la conservación de los procesos evolutivos. Hereditas 130, 217-228. doi: 10.1111 / j. 1601-5223. 1999. 00217.x
CrossRef Full Text/Google Scholar
Moritz, C. (2002). Estrategias para proteger la diversidad biológica y los procesos evolutivos que la sustentan. Syst. Biol. 51, 238–254. doi: 10.1080 | 10635150252899752
Resumen de PubMed/Texto completo cruzado/Google Scholar
Moritz, C., and Potter, S. (2013). La importancia de una perspectiva evolutiva en la planificación de políticas de conservación. Mol. Ecol. 22, 5969–5971. doi: 10.1111 / mec.12565
Resumen de PubMed / Texto completo Cruzado / Google Scholar
Moritz, C., Pratt, R. C., Bank, S., Bourke, G., Bragg, J. G., Doughty, P., et al. (2018). Diversidad de linaje críptico, divergencia de tamaño corporal y simpatría en un complejo de especies de lagartos australianos (Gehyra). Evolución 27, 54-66. doi: 10.1111 / evo.13380
CrossRef Texto Completo | Google Scholar
Morrison, W. R., Lohr, J. L., Duchen, P., Wilches, R., Trujillo, D., Mair, M., et al. (2009). El impacto del cambio taxonómico en la conservación: ¿Mata, puede salvar o es irrelevante? Biol. Desventaja. 142, 3201–3206. doi: 10.1016 / j. biocon.2009.07.019
Texto completo de CrossRef | Google Scholar
Murphy, N. P., Adams, M., Guzik, M. T., and Austin, A. D. (2013). Microendemismo extraordinario en anfípodos de primavera del desierto australiano. Mol. Phylogenet. Evol. 66, 645–653. doi: 10.1016 / j. ympev.2012.10.013
Resumen de PubMed | Texto completo Cruzado/Google Scholar
Murphy, N. P., King, R. A., and Deean, S. (2015). ¿Especies, ESU o poblaciones? Delimitar y describir morfológicamente diversidad críptica en el desierto de Australia primavera anfípodos. Invertir. Syst. 29, 457–467. doi: 10.1071/IS14036
CrossRef Texto Completo | Google Scholar
Nater, A., Mattle-Greminger, M. P., Nurcahyo, A., Nowak, M. G., de Manual, M., Desai, T., et al. (2017). Evidencia morfométrica, conductual y genómica de una nueva especie de orangután. Curr. Cell Biol. 27, 3487–3498. doi: 10.1016 / j.cub.2017.09.047
Texto completo de CrossRef | Google Scholar
Nicotra, A. B., Chong, C., Bragg, J. G., Ong, C. R., Aitken, N. C., Chuah, A., et al. (2016). Population and phylogenomic decomposition via genotyping-by-sequencing in Australian Pelargonium. Mol. Ecol. 25, 2000–2014. doi: 10.1111 / mec.13584
Resumen de PubMed / Texto completo cruzado / Google Scholar
Nistelberger, H., Byrne, M., Coates, D., and Roberts, J. D. (2014a). Una fuerte estructura filogeográfica en un milpiés indica una vicarianza pleistocénica entre poblaciones en formaciones de hierro con bandas en Australia semiárida. PLoS ONE 9: e0093038. doi: 10.1371 / journal.ponga.0093038
Resumen de PubMed / Texto completo cruzado/Google Scholar
Nistelberger, H., Gibson, N., Macdonald, B., Tapper, S.-L., and Byrne, M. (2014b). Evidencia filogeográfica de dos refugios de mesic en un hotspot de biodiversidad. Heredity 113, 454-463. doi: 10.1038 / hdy.2014.46
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Ogilvie, H. A., Heled, J., Xie, D., y Drummond, A. J. (2016). Rendimiento computacional y precisión estadística de beast y comparaciones con otros métodos. Syst. Biol. 65, 381–396. doi: 10.1093/sysbio/syv118
Resumen de PubMed | Texto completo cruzado/Google Scholar
Oliver, P. M., Keogh, J. S., and Moritx, C. (2015). New approaches to cataloguing and understanding evolutionary diversity: a perspective from Australian herpetology (en inglés). Aust. J. Zool. 62, 417–430. doi: 10.1071 / ZO14091
Texto completo cruzado / Google Scholar
Oliver, P. M., Laver, R. J., Demello Martins, F., Pratt, R. C., Hunjan, S., and Moritz, C. (2017). A novel hotspot of vertebrate endemism and an evolutionary refugium in tropical Australia (en inglés). Buceador. Distrib. 23, 53–66. doi: 10.1111 / ddi.12506
CrossRef Full Text/Google Scholar
Oliver, P. M., and Lee, M. S. Y. (2010). Los códigos botánicos y zoológicos impiden la investigación de la biodiversidad al desalentar la publicación de nuevas especies sin nombre. Taxon 59, 1201-1205. doi: 10.2307 / 20773990
Texto completo cruzado / Google Scholar
Padial, J., Miralles, A., De la Riva, I., and Vences, M. (2010). The integrative future of taxonomy (en inglés). Delantero. Zool. 7:16. doi: 10.1186/1742-9994-7-16
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Palsbøll, P. J., Bérubé, M., and Allendorf, F. W. (2007). Identificación de unidades de manejo utilizando datos genéticos de población. Tendencias Ecol. Evol. 22, 11–16. doi: 10.1016 / j.tree.2006.09.003
Resumen de PubMed | Texto completo cruzado/Google Scholar
Patton, J. L., and Smith, M. F. (1994). Paraphyly, polyphyly, and the nature of species boundaries in Pocket Gophers (Genus Thomomys). Syst. Biol. 43, 11–26. doi: 10.1093 / sysbio / 43.1.11
Texto completo cruzado/Google Scholar
Pierson, J.C., Coates, D. J., Oostermeijer, J. G. B., Beissinger, S. R., Bragg, J. G., Sunnucks, P., et al. (2016). Factores genéticos en planes de recuperación de especies amenazadas en tres continentes. Delantero. Ecol. Environ. 14, 433–440. doi: 10.1002 / tarifa.1323
Texto completo de CrossRef/Google Scholar
Potter, S., Bragg, J. G., Peter, B. M., Bi, K., and Moritz, C. (2016). Phylogenomics at the tips: inferring lineages and their demographic history in a tropical lizard, Carlia amax. Mol. Ecol. 25, 1367–1380. doi: 10.1111 / mec.13546
Resumen de PubMed | Texto completo cruzado/Google Scholar
Potter, S., Xue, A. T., Bragg, J. G., Rosauer, D. F., Roycroft, E. J. y Moritz, C. (2018). Los cambios climáticos del pleistoceno impulsan la diversificación a través de una sabana tropical. Mol. Ecol. 27, 520–532. doi: 10.1111 / mec.14441/full
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Rabosky, D. L., Hutchinson, M. N., Donnellan, S. C., Talaba, A. L., y Lovette, I. J. (2014). Phylogenetic disassembly of species boundaries in a widespread group of Australian skinks (Scincidae: Ctenotus). Mol. Phylogenet. Evol. 77, 71–82. doi: 10.1016 / j. ympev.2014.03.026
Resumen de PubMed | Texto completo Cruzado | Google Scholar
Ralls, K., Ballou, J. D., Dudash, M. R., Eldridge, M. D. B., Fenster, C. B., Lacy, R. C., et al. (2018). Pedir un cambio de paradigma en el manejo genético de poblaciones fragmentadas. Conserv. Lett. 11, 1–6. doi: 10.1111 / conl.124126
CrossRef Full Text | Google Scholar
Rannala, B. (2015). The art and science of species delimitation (en inglés). Curr. Zool. 61, 846–853. doi: 10.1093 / czoolo / 61.5.846
Texto completo de CrossRef / Google Scholar
Riedel, A., Sagata, K., Suhardjono, Y. R., Tänzler, R., and Balke, M. (2013). Taxonomía integradora en la vía rápida-hacia una mayor sostenibilidad en la investigación de la biodiversidad. Delantero. Zool. 10:15. doi: 10.1186 / 1742-9994-10-15
Resumen de PubMed | Texto completo cruzado/Google Scholar
Rieseberg, L. H., and Willis, J. H. (2007). Especiación de plantas. Science 317, 910-914. doi: 10.1126 / ciencia.1137729
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Rosauer, D. F., Blom, M. P. K., Bourke, G., Catalano, S., Donnellan, S., Mulder, E., et al. (2016). Phylogeography, hotspots and conservation priorities: an example from the Top End of Australia (en inglés). Biol. Desventaja. 204, 83–93. doi: 10.1016 / j. biocon.2016.002
Texto completo de CrossRef | Google Scholar
Rosauer, D. F., Byrne, M., Blom, M. P. K., Coates, D. J., Donnellan, S., Doughty, P., et al. (2018). Real-world conservation planning for evolutionary diversity in the Kimberley, Australia, soidesteps uncertain taxonomy Conserv. Lett. 11: e12438. doi: 10.1111 / conl.12438
Texto completo de CrossRef | Google Scholar
Rosenblum, E. B., Sarver, B. A. J., Brown, J. W., Des Roches, S., Hardwick, K. M., Hether, T. D., et al. (2012). Ricitos de oro se encuentra con Santa Rosalía: un modelo de especiación efímera explica los patrones de diversificación a través de escalas de tiempo. Evol. Biol. 39, 255–261. doi: 10.1007 / s11692-012-9171-x
Resumen de PubMed / Texto completo cruzado / Google Scholar
Roux, C., Fraïsse, C., Romiguier, J., Anciaux, Y., Galtier, N., and Bierne, N. (2016). Arrojando luz sobre la zona gris de especiación a lo largo de un continuo de divergencia genómica. PLoS Biol. 14: e2000234. doi: 10.1371 / journal.pbio.2000234
Resumen de PubMed | Texto completo de CrossRef/Google Scholar
Ryder, O. A. (1986). Species conservation and systematics – the dilemma of subspecies (en inglés). Tendencias Ecol. Evol. 1, 9–10. doi: 10.1016/0169-5347 (86)90059-5
Texto completo de CrossRef | Google Scholar
Sampson, J. F., Byrne, M., Yates, C. J., Gibson, N., Thavornkanlapachai, R., Stankowski, S., et al. (2014). La inmigración genética contemporánea mediada por el polen refleja el aislamiento histórico de un arbusto raro en un paisaje fragmentado. Heredity 112, 172-181. doi: 10.1038 / hdy.2013.89
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Santamaría, L. y Méndez, P. F. (2012). Evolution in biodiversity policy: current gaps and future needs. Evol. Appl. 5, 202–218. doi: 10.1111 / j. 1752-4571. 2011. 00229.x
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Schlick-Steiner, B. C., Steiner, F. M., Seifert, B., Stauffer, C., Christian, E., y Crozier, R. H. (2010). Integrative taxonomy: a multisource approach to exploring biodiversity. Annu. Apo. Entomol. 55, 421–438. doi: 10.1146 / annurev-ento-112408-085432
Resumen de PubMed | Texto completo cruzado | Google Scholar
Seehausen, O, Takimoto, G., Roy, D., and Jokela, J. (2008). Inversión de especiación y dinámica de biodiversidad con hibridación en ambientes cambiantes. Mol Ecol. 17, 30–44. doi: 10.1111 / j. 1365-294X. 2007. 03529.x
Resumen de PubMed / Texto completo cruzado / Google Scholar
Shepherd, K. A., Thiele, K. R., Sampson, J., Coates, D. J. y Byrne, M. (2015). A rare, new species of Atriplex (Chenopodiaceae) comprising two genetically distinct but morphologically críptic populations in arid Western Australia: implications for taxonomy and conservation. Aust. Syst. Robot. 28, 234–245. doi: 10.1071/SB15029
Texto completo de CrossRef/Google Scholar
Singhal, S., and Bi, K. (2017). History cleans up messes: The impact of time in driving divergence and introgression in a tropical suture zone (en inglés). Evolution 71, 1888-1899. doi: 10.1111 / evo.13278
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Singhal, S., Hoskin, C., Couper, P., Potter, S., y Moritz, C. (2018). Framework for resolving cryptic species: a case study from the lizards of the Australian wet tropics (en inglés). Syst. Biol. . doi: 10.1093/sysbio/syy
Resumen PubMed | Texto completo Cruzado/Google Scholar
Singhal, S., and Moritz, C. (2013). Aislamiento reproductivo entre escalas de linajes filogeográficos con divergencia. Proc. Roy. Soc B 280: 20132246. doi: 10.1098 / rspb.2013.2246026
PubMed Abstract | CrossRef Texto Completo | Google Scholar
Steane, D. A., Potts, B. M., McLean, E., Collins, L., Prober, S. M., Stock, W. D., et al. (2015). Las exploraciones de todo el genoma revelan una estructura críptica de la población en un eucalipto adaptado a seco. Genet de Árbol. Genomas 11:33. doi: 10.1007 / s11295-015-0864-z
Texto completo cruzado / Google Scholar
Struck, T. H., Feder, J. L., Bendiksby, M., Birkeland, S., Cerca, J., Gusarov, V. I., et al. (2017). Encontrar procesos evolutivos ocultos en especies crípticas. Tendencias Ecol. Evol. 33, 153–163. doi: 10.1016 / j.tree.2017.11.007
Resumen de PubMed / Texto completo cruzado / Google Scholar
Sukumaran, J., and Knowles, L. L. (2017). La coalescente multiespecie delimita la estructura, no las especies. Proc. NAT. Aca. Sci. U. S. A. 114, 1607-1612. doi: 10.1073 / pnas.1607921114
Resumen de PubMed | Texto completo cruzado/Google Scholar
Swarts, N. D., Clements, M. A., Bower, C. C., and Miller, J. T. (2014). Definiendo unidades de conservación en un complejo de orquídeas morfológicamente similares, sexualmente engañosas y en peligro de extinción. Biol. Desventaja. 174, 55–64. doi: 10.1016 / j. biocon.2014.03.017
Texto completo de CrossRef | Google Scholar
Tautz, D., Arctander, P., Minelli, A., Thomas, R. H., and Vogler, A. P. (2003). Una petición de taxonomía de ADN. Tendencias Ecol. Evol. 18, 70–74. doi: 10.1016/S0169-5347(02)00041-1
Texto completo cruzado/Google Scholar
Taylor, H. R., Dussex, N., and van Heezik, Y. (2017). Cerrar la brecha genética de la conservación mediante la identificación de barreras para la implementación para los profesionales de la conservación. Glob. Ecol. Conserv. 10, 231–242. doi: 10.1016 / j. gecco.2017.04.001
CrossRef Texto completo | Google Scholar
Thomson, S.A., Pyle, R. L., Ahyong, S. T., Alonso-Zarazaga, M., Ammirati, J., Araya, J. F., et al. (2018). La taxonomía basada en la ciencia es necesaria para la conservación global. PLoS Biol. 16: e2005075. doi: 10.1371 diario.pbio.2005075
Resumen de PubMed | Texto completo Cruzado | Google Scholar
Vieites, D. R., Wollenberg, K. C., Andreonec, F., Köhler, J., Glaw, F., y Vences, M. (2009). Gran subestimación de la biodiversidad de Madagascar evidenciada por un inventario integrado de anfibios. Proc. Natl. Acad. Sci. U. S. A. 106, 8267-8272. doi: 10.1073 / pnas.0810821106
Texto completo cruzado
Vogler, A. P., and de Salle, R. (1994). Diagnóstico de Unidades de Gestión de la Conservación. Desventaja. Biol. 8, 354–363. doi: 10.1046 / j. 1523-1739. 1994. 08020354.x
CrossRef Full Text/Google Scholar
Waples, R. S. (1991). Salmón del Pacífico, Oncorhynchus spp., y la definición de» especie » en la Ley de Especies en Peligro de Extinción. Estropear. Pescado. Apo. 53, 11-22.
Google Scholar
Waples, R. S. (1995). «Evolutionarily significant units and the conservation of biological diversity under the Endangered Species Act», en Evolution and the Aquatic Ecosystem: Defining Unique Units in Population Conservation, eds J. L. Nielsen (Bethesda, MD:American Fisheries Society), 8-27.
Google Scholar
Waples, R. S., Gustafson, R. G., Weitkamp, L. A., Myers, J. M., Jjonson, O. W., Busby, P. J., et al. (2001). Characterizing diversity in salmon from the Pacific Northwest. J. Fish Biol. 59, 1–41. doi: 10.1111 / j. 1095-8649. 2001.tb01376.x
CrossRef Full Text / Google Scholar
Waples, R. S., Nammack, M., Cochrane, J. F., and Hutchings, J. A. (2013). A tale of two acts: endangered species listing practices in Canada and the United States (en inglés). BioScience 63, 723-734. doi: 10.1093 / bioscience / 63.9.723
Texto completo de CrossRef / Google Scholar
Waples, R. S., Punt, A. E., and Cope, J. M. (2008). Integración de los datos genéticos en la gestión de los recursos marinos: ¿cómo podemos hacerlo mejor? Fish 9, 423-449. doi: 10.1111 / j. 1467-2979. 2008. 00303.x
Texto completo cruzado / Google Scholar
Wege, J. A., Thiele, K. R., Shepherd, K. A., Butcher, R., Macfarlane, T. D., and Coates, D. J. (2015). Taxonomy Strategic in a biodiverse landscape: a novel approach to maximizing conservation outcomes for rare and poorly known flora. Biodivers. Conserv. 24, 17–32. doi: 10.1007 / s10531-014-0785-4
CrossRef Full Text/Google Scholar
Whitlock, R. (2014). Relationships between adaptive and neutral genetic diversity and ecological structure and functioning: a meta-analysis. J. Ecol. 102, 857–872. doi: 10.1111/1365-2745.12240
Resumen PubMed | Texto completo cruzado/Google Scholar
Wiley, E. O. (1978). El concepto de especie evolutiva reconsiderado. Syst. Biol. 27, 17–26. doi: 10.2307 / 2412809
Texto completo de CrossRef/Google Scholar
Wilson, E. O. (2017). La investigación de la biodiversidad requiere más recursos en el terreno. NAT. Ecol. Evol. 1, 1590–1591. doi: 10.1038 / s41559-017-0360-y
Resumen de PubMed / Texto completo cruzado / Google Scholar
Yang, Z. H., and Rannala, B. (2014). Delimitación de especies no guiada utilizando datos de secuencias de ADN de múltiples loci. Mol. Biol. Evol. 31, 3125–3135. doi: 10.1093 / molbev / msu279
Resumen PubMed / Texto completo cruzado / Google Scholar
Yeates, D. K., Seago, A., Nelson, L., Cameron, S.L., Joseph, L., and Trueman, W. H. (2011). Taxonomía integrativa, o taxonomía iterativa? Sys. Ent. 36, 209–217. doi: 10.1111/j.1365-3113.2010.00558.x
CrossRef Full Text | Google Scholar