No início da década de 1960, os biólogos começaram a investigar como as proteínas de diferentes espécies evoluem na sequência de nível (Zuckerkandl 1962, Margoliash de 1963, Doolittle & Blombäck 1964). As proteínas analisadas incluíram a hemoglobina (Zuckerkandl & Pauling 1962), citocromo C (Margoliash 1963), e fibrinopeptides (Doolittle & Blombäck 1964). Estas primeiras investigações levaram a uma descoberta notável: parecia que o número de diferenças entre sequências de proteínas de diferentes espécies eram aproximadamente proporcionais ao tempo desde a divergência das espécies (Figura 1).
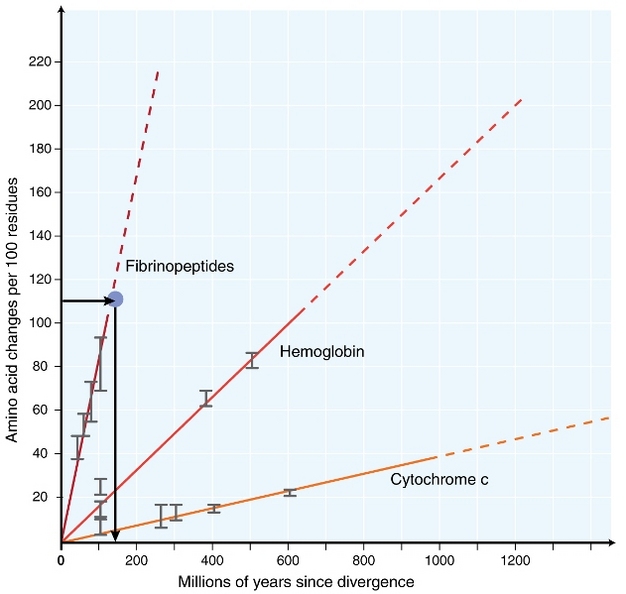
Zuckerkandl & Pauling (1965) comparou a acumulação constante de aminoácidos substituições ao longo do tempo para regular ‘ticks’ de relógios, e afirmou que “não pode existir molecular relógio evolutivo’. Assim, o termo “relógio molecular” foi inicialmente inventado para descrever mudanças nos aminoácidos que ocorrem em proporção ao tempo desde a divergência das espécies.
desde o seu primeiro uso, o termo “relógio molecular” tem sido usado em muitos contextos diferentes. Hoje em dia, é frequentemente utilizado para se referir simplesmente ao número de alterações, ou ‘substituições’, acumuladas na sequência de ADN ou proteínas numa determinada linhagem. O número de substituições por uma unidade de tempo definida pode ser descrito como a “taxa” do relógio molecular, que neste contexto é equivalente a “taxa evolutiva”. Note que a conotação inicial ainda se mantém em alguns casos. Por exemplo, quando as pessoas afirmam “nós assumimos um relógio molecular” (particularmente em análises filogenéticas), isso significa que os números de substituições foram assumidos para ocorrer constantemente ao longo do tempo.
O conceito de um relógio molecular constante tem implicações extraordinárias para biólogos evolucionários. Se um relógio molecular constante como inicialmente proposto realmente existisse, inferir o tempo dos eventos evolutivos se tornaria um problema bastante simples (Figura 1). No entanto, tornou-se bastante claro que as substituições não ocorrem constantemente ao longo do tempo em diferentes linhagens (Kumar 2005). No entanto, o conceito de relógio molecular tem sido extremamente influente no campo da evolução molecular. Uma das ideias mais importantes inspiradas pelo conceito do relógio molecular é a teoria neutra da evolução molecular.
O Relógio Molecular e Neutralidade
Quando a idéia de uma constante do relógio molecular surgiu pela primeira vez, pensava-se que o predominante evolutiva força subjacente de aminoácidos ou nucleotídeos substituições foi a seleção natural. Seguindo esta linha de pensamento, um relógio molecular constante indicaria que substituições adaptativas em diferentes espécies ocorrem constantemente ao longo do tempo. No entanto, é difícil explicar como substituições adaptativas ocorreriam de forma semelhante a um relógio. Teoricamente, os destinos das mutações adaptativas são determinados por vários parâmetros evolutivos, tais como a força da vantagem seletiva dessa mutação, o tamanho da população efetiva e as taxas de mutação adaptativa (Kimura 1983). Estes parâmetros são susceptíveis de diferir entre espécies, e mesmo dentro de uma espécie, dependendo de mutações específicas e suas interações com ambientes. em vez disso, Kimura (1968, 1969) propôs que a maioria das mudanças a nível molecular têm poucas consequências funcionais, ou são “neutras”. Se uma mutação não tem consequência de aptidão, o seu destino na população é determinado completamente por acaso Aleatório. Isto significa que não podemos prever se uma mutação neutra específica será eventualmente fixada na população. No entanto, a taxa a que substituições neutras ocorrem na população pode ser prevista porque depende de um único parâmetro, a saber, a taxa de mutação (Kimura 1968). vamos imaginar uma população com N número de indivíduos haplóides. Se ocorrerem mutações neutras a taxa u por indivíduo por geração, o número total de mutações numa geração será N vezes u. Uma vez que todas estas novas mutações são neutras, os seus destinos são completamente determinados por acaso. Em outras palavras, todas as mutações têm a mesma chance de alcançar a fixação (o que leva a uma “substituição”). A probabilidade de cada nova mutação neutra atingir a fixação, dado que ocorreu uma substituição, é simplesmente 1 / N. A taxa de substituições é calculado como o número de novas mutações em cada geração (Nu) multiplicada pela probabilidade de cada nova mutação atinge fixação (1/N), que é igual a u. Em outras palavras, para neutras as mutações, a taxa de substituição é igual à taxa de mutação! portanto, se a maioria das mutações são neutras (como proposto na teoria neutra) e se as taxas de mutação são constantes ao longo do tempo, as substituições também devem ocorrer constantemente ao longo do tempo. Devemos então observar taxas regulares de substituições de clock a nível molecular. Kimura (1969) thus considered the observation of relatively constant molecular clock in protein sequences as strong support for the neutral theory of molecular evolution.
Testing How the Neutral Molecular Clock Run
According to the neutral theory, the question of whether substitution rates are constant over time or not is equivalent to whether neutral mutation rates are constant over time. Por esta razão, muitos estudos subsequentes focaram na análise de dados de locais neutros para determinar se as taxas de mutação neutra são realmente constantes ao longo do tempo. Vamos rever brevemente a forma como estes estudos são implementados, antes de discutir debates teóricos sobre relógios moleculares. Análises de relógios moleculares proteicos também continuaram, mas os debates em torno da variação de relógios moleculares proteicos são muito diferentes dos relativos aos relógios moleculares neutros, e não serão incluídos neste artigo. Os leitores interessados devem consultar Gillespie (1991), Kumar (2005), Kim and Yi (2008) e Bedford et al. (2008).
A maioria das análises empíricas de relógios moleculares neutros baseiam-se no teorema de que as taxas de mutação neutra podem ser inferidas a partir de taxas de substituição neutras (Kimura 1968, 1969). Na prática, cada estudo define um determinado tipo de sítios no genoma como sítios neutros e compara as taxas de substituição desses sítios entre linhagens.
Quais os sítios no genoma são verdadeiramente neutros não podem ser completamente determinados, mas os cientistas apresentaram vários proxies úteis. Antes da era da sequenciação do genoma, a maioria dos dados de sequência disponíveis eram os de sequências de ADN codificantes de proteínas. Os estudos dividem frequentemente as sequências de ADN codificadoras de proteínas em dois tipos de locais (Wu & Li 1985). O primeiro tipo de sítios inclui aqueles para os quais qualquer alteração levaria a substituições de aminoácidos, ou “sítios não sinónimos”. O segundo tipo de sítios, “sítios sinónimos”, inclui os que codificam posições “degeneradas” na tabela de códons, onde uma alteração não conduz a uma substituição de aminoácidos. Por exemplo, TCT e TCC ambos codificam serina. Se a terceira posição destes codões mudar, eles ainda produzirão os mesmos aminoácidos. Tais substituições seriam menos visíveis para a seleção natural. Consequentemente, relógios moleculares em locais sinônimos devem estar mais perto do relógio molecular neutro do que Relógios Não sinônimos.
Como técnicas de sequenciamento avançadas, alguns estudos usaram introns como proxies para locais neutros, uma vez que eles não são incorporados no mRNAs maduro, e, portanto, são mais propensos a ser neutros (Yi et al. 2002). Sequências de elementos transposíveis inactivos que foram inseridos muito antes de a divergência de espécies também ser frequentemente utilizada (estas são frequentemente referidas como “repetições ancestrais”, por exemplo, Thomas et al., 2003). Finalmente, alguns estudos utilizaram sequências de ADN não codificantes (todas as sequências após a remoção das sequências de ADN codificador de proteínas) extraídas de alinhamentos de genoma inteiros para testar relógios moleculares neutros (Elango et al. 2006, Huttley et al. 2007).
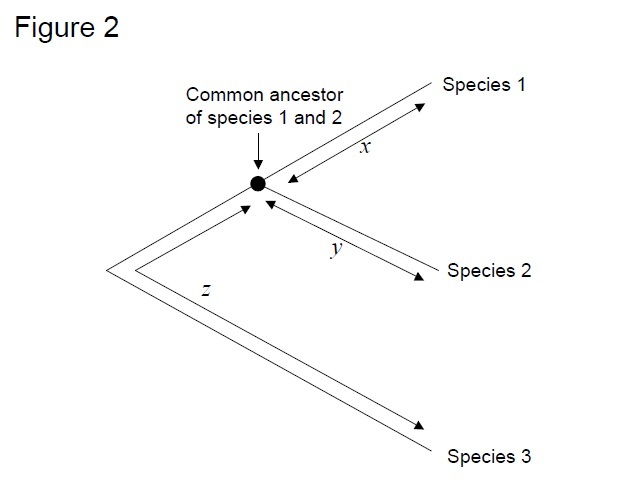
O teste mais frequentemente utilizado é o chamado “teste da taxa relativa”(Sarich & Wilson 1973). Inicialmente, as taxas de substituição por unidade de tempo foram estimadas dividindo o número total de diferenças (substituições) entre proteínas de diferentes espécies pelo tempo de divergência, estimado a partir de registros fósseis (Figura 1). No entanto, registros fósseis não estão disponíveis para muitas comparações, e estão associados com grandes margens de erro. O ensaio da taxa relativa supera a necessidade de registos fósseis (Figura 2). Enquanto existir uma sequência de grupos para as duas linhagens de interesse, podemos determinar se os dois ramos seguem o mesmo ou diferentes relógios moleculares, sem o conhecimento do tempo absoluto de divergência (Figura 2).
What are the Determinants of Neutral Molecular Clocks?
quase todas as controvérsias no centro dos debates sobre relógios moleculares neutros derivam da questão de quais são as principais fontes de mutações. Esta questão é diretamente relevante para a compreensão de padrões de mutação, que são a principal fonte de mudança evolutiva e doença genética. Além disso, compreender como as taxas de mutação variam entre linhagens e dentro dos genomas é uma questão fundamental na genômica comparativa, que visa usar comparações de seqüências para identificar regiões genômicas que são funcionalmente importantes. o que determina as taxas de mutação neutra? Um dos mais importantes contribuintes para Relógios moleculares neutros é a variação específica da linhagem em tempos de geração. Desde o início, a ideia de um relógio molecular neutro constante foi percebida como estando em desacordo com os mecanismos moleculares da mutação germinal. Há muito tempo que se considera que a maioria das mutações resultam de erros na replicação do ADN nas linhagens (Haldane 1947, Muller 1954). Uma vez que as mutações ocorrem quando o DNA germinal é replicado para a próxima geração, elas devem acumular-se em proporção ao número de gerações, em vez da quantidade absoluta de tempo. Portanto, se compararmos os números de substituições que se acumularam em duas linhagens desde a sua divergência, a linhagem com tempo de geração mais longo, tendo sofrido menos eventos de replicação de DNA, abrigaria menos substituições em comparação com a linhagem com o tempo de geração mais curto. Consequentemente, o relógio molecular deve correr mais lentamente em espécies com tempos de geração mais longos. Esta ideia é referida como o “efeito geração-tempo”.de facto, o efeito tempo de geração foi observado pela primeira vez em estudos de primatas, mesmo antes do debate sobre o relógio molecular. Morris Goodman, que estava usando métodos imunológicos para investigar a relação de espécies na época, observou que a taxa a que algumas proteínas divergem parecia ser diminuída em macacos, em particular humanos, em comparação com os macacos do Velho Mundo (Goodman 1961, 1962, 1963). Este efeito é referido como “abrandamento da taxa hominóide”. Uma vez que os hominóides têm tempos de geração mais longos do que os macacos do Velho Mundo, esta observação pode ser explicada pelo efeito geração-tempo.
Wu& Li (1985) foram os primeiros a testar a hipótese do efeito de geração-tempo usando dados de sequência de ADN. Eles usaram dados de 11 genes de primatas e roedores. Uma vez que os primatas têm um tempo de geração muito mais longo do que os roedores, o relógio molecular deve ser mais rápido nos roedores em comparação com os primatas. Na verdade, eles descobriram que para sites sinônimos, roedores mostram aproximadamente duas vezes a taxa de evolução molecular quando comparados com primatas (Wu & Li 1985). No entanto, para sites não sinônimos, tal efeito não foi encontrado. Em outras palavras, o relógio molecular neutro, mas não o relógio molecular de aminoácidos, funciona mais rápido na linhagem de roedores em comparação com a linhagem de primatas, que se encaixa bem com a idéia de um efeito de geração-tempo. estudos posteriores contribuíram para o abrandamento da taxa de hominóides (Li &
Li 1992, Huttley et al. 2007). Além disso, observou-se uma diferença de taxa em escalas filogenéticas ainda menores, especialmente em primatas.: por exemplo, o relógio molecular humano é mais lento que o relógio molecular chimpanzé (Elango et al. 2006); as taxas nos macacos do Novo Mundo são mais rápidas que as taxas nos hominóides e nos macacos do Velho Mundo (Steiper & jovem 2006). As diferentes taxas de relógios moleculares observadas nestes estudos são qualitativamente consistentes com o efeito geração-tempo. no entanto, as diferenças reais entre as linhagens não são quantitativamente consistentes com a diferença nos tempos de geração. Por exemplo, Kumar & Subramanian (2002) mostrou que, embora a diferença em tempos de geração entre os primatas e os roedores é muito maior do que entre os humanos e os macacos do Velho Mundo, as diferenças observadas nos relógios moleculares são semelhantes nas duas comparações. Vale a pena notar que Kumar & Subramanian (2002) usou filtros estatísticos específicos para remover dados que mostravam padrões de substituição “heterogéneos”, o que poderia ter causado um viés em direção a sequências em evolução lenta (Yi et al. 2002). No entanto, a diferença nos relógios moleculares de primatas e roedores parece muito inferior à originalmente proposta por Wu & Li (1985). Por exemplo, Huttley et al. (2007) analisou alinhamentos de genoma inteiro de várias espécies, incluindo o opossum, e mostrou que a diferença de taxa entre linhagens euterianas e a linhagem opossum (~30%) é muito maior do que a diferença de taxa entre linhagens humanas e mouse (~14%). Estes exemplos demonstram que o grau de diferenças nos relógios moleculares varia significativamente entre os diferentes estudos devido a diferenças nos conjuntos de dados e métodos estatísticos. Eles também mostram que as diferenças de taxa entre linhagens não podem ser completamente contabilizadas pela diferença apenas nos tempos de geração. Claramente, há outros contribuintes para Relógios moleculares neutros.
de fato, traços de história de vida que não os tempos de geração parecem co-variar com relógios moleculares. Martin & Palumbi (1993) demonstrou que os relógios moleculares do ADN são mais rápidos em espécies com pequeno tamanho corporal. Esta observação levou à hipótese de que as taxas metabólicas são determinantes importantes dos relógios moleculares. Uma elevada taxa metabólica produz um grande número de radicais mutagénicos de oxigénio, o que aumentaria as taxas de mutação (Rand 1994). Como as taxas metabólicas e o tamanho do corpo geralmente variam com os tempos de geração, tem sido difícil distinguir qual deles constitui o principal determinante das taxas de clock molecular. Tsantes & Steiper (2009) propuseram, com base em dados de primatas, que a idade na primeira reprodução, ao invés do tamanho do corpo, é o principal determinante dos relógios moleculares. Uma vez que a idade na primeira reprodução reflete o efeito tempo de geração, este estudo apoia a ideia de que o efeito tempo de geração é o principal determinante do relógio molecular. No entanto, este estudo baseia-se ainda num número limitado de linhagens (foram utilizados quatro pares de espécies). Assim, a distinção entre Tamanho do corpo, efeito de geração-tempo, e taxas metabólicas ainda permanece como uma questão importante na generalização e compreensão de relógios moleculares neutros.
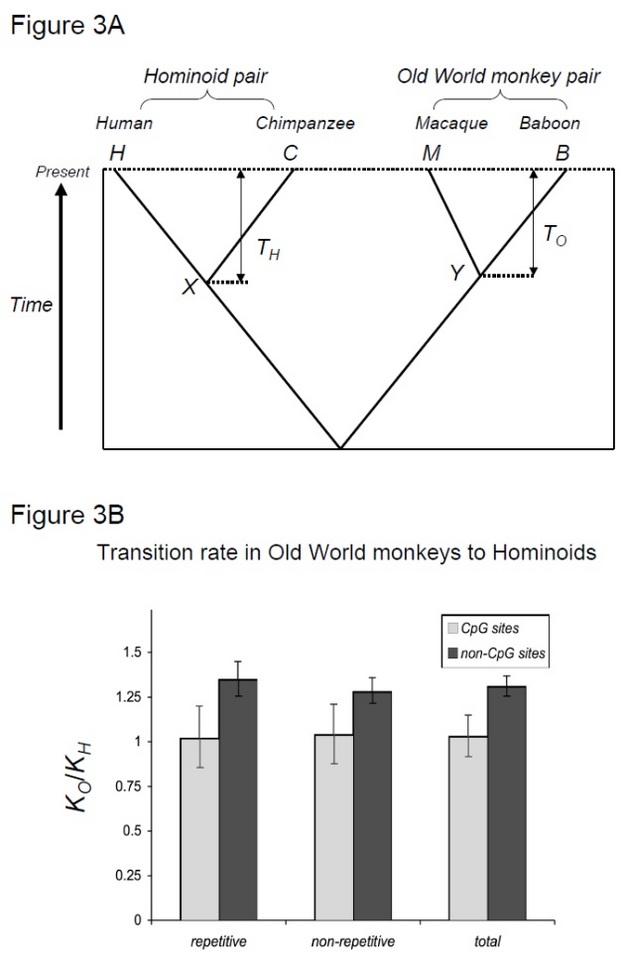
além disso, a importância de factores que não co-variam com o tempo de geração, como a metilação do ADN, tem sido cada vez mais apreciada. A metilação do DNA é uma modificação química do DNA genômico encontrado em diversos táxons. Nos genomas animais, a metilação do ADN ocorre quase exclusivamente nas citosina seguidas de guaninas (chamadas “CpG”). Citosinas metiladas, por sua vez, tendem a sofrer mutações rápidas para as timinas devido à instabilidade química (Bird 1980). Na verdade, no genoma humano, as mutações causadas pela metilação do ADN ocorrem mais do que uma ordem de magnitude mais frequentemente do que outras mutações (Nachman & Crowell 2000, Elango et al. 2008). Como as mutações causadas pela metilação do ADN ocorrem em grande parte independentemente da replicação do ADN, tais mutações podem seguir relógios moleculares diferentes dos outros. Especificamente, em vez de dependência do tempo de geração, as mutações causadas pela metilação do ADN podem seguir um relógio molecular dependente do tempo, que é semelhante ao que foi inicialmente proposto por Zuckerkandl & Pauling (Kim et al. 2006)! para testar esta hipótese, Kim et al. (2006) compared human-chimpanzee divergence to macaque-baboon divergence, two species pairs that share similar divergence times but with different generation times (Steiper et al. 2004). O par homem-chimpanzé (o par hominóide) tem tempos de geração muito mais longos em comparação com o par macaco-babuíno (o par macaco do Velho Mundo). Este estudo mostrou que para sites não-CpG, o par de macacos do Velho Mundo acumulou aproximadamente 30% mais substituições, o que pode ser explicado pelo efeito de desaceleração da taxa hominóide acima mencionado. Em contraste, relógios moleculares em locais de CpG mostraram números semelhantes de substituições em pares hominóides e macacos do Velho Mundo (Figura 3). Assim, relógios moleculares dependentes do tempo e da geração dependentes do tempo coexistem dentro dos mesmos genomas. A suposição de que um único relógio molecular pode existir para uma determinada linhagem não é mais válida, porque as forças mutacionais predominantes variam entre as regiões genômicas.
Conclusions
the concept of a constant molecular clock was initially proposed based on a limited amount of protein sequence data. Embora estudos posteriores tenham mostrado que tal observação não é um padrão geral em aminoácidos, ela teve influência significativa no campo da Evolução molecular, em particular no desenvolvimento da teoria neutra da Evolução molecular para dados de sequência de DNA. Seguindo a teoria neutra, estudos focaram em elucidar padrões de variação nas taxas de mutação neutra. Durante as últimas décadas, observámos que os relógios moleculares funcionam a taxas diferentes entre linhagens. Além disso, o grau de variação pode variar em função dos diferentes tipos de dados e dos métodos estatísticos específicos utilizados. O efeito tempo de geração continua a manter-se a um nível qualitativo, mas é insuficiente para explicar a variação quantitativa das taxas de mutação neutra entre as linhagens. Traços de história de vida e mutações dependentes da não-replicação, tais como as causadas pela metilação do DNA, também são importantes contribuintes para Relógios moleculares genômicos. Na verdade, diferentes tipos de relógios moleculares são observados mesmo dentro de um genoma, porque as entradas mutacionais predominantes variam entre diferentes regiões genômicas. Assim, ao invés de assumir um único relógio molecular neutro para cada genoma, estudos futuros devem ter como objetivo revelar a variação de relógios moleculares neutros genômicos, para aprender sobre paisagens mutacionais genômicas. Essa informação não é apenas útil para compreender a matéria-prima que rege a evolução molecular e a doença genética, mas também constitui um componente crítico que influencia as análises genómicas comparativas e funcionais para identificar as regiões genómicas funcionais.