we wczesnych latach 60.biolodzy zaczęli badać, w jaki sposób białka u różnych gatunków ewoluują na poziomie sekwencji (Zuckerkandl 1962, margoliash 1963, Doolittle & blombäck 1964). Badane białka obejmowały hemoglobinę (Zuckerkandl & Pauling 1962), cytochrom C (Margoliash 1963) i fibrynopeptydy (Doolittle & Blombäck 1964). Te wczesne badania doprowadziły do niezwykłego odkrycia: okazało się, że liczba różnic między sekwencjami białek różnych gatunków była w przybliżeniu proporcjonalna do czasu od dywergencji gatunków (ryc. 1).
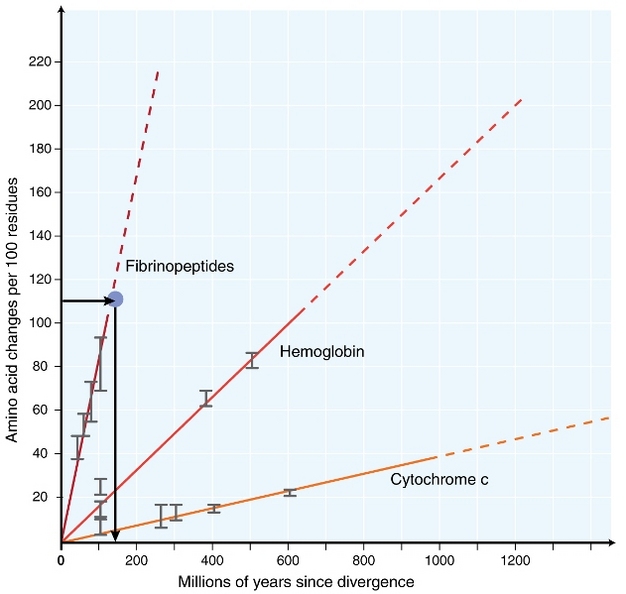
Zuckerkandl &Pauling (1965) porównał stałą akumulację podstawień aminokwasów w czasie do regularnych „kleszczy” zegarów i stwierdził, że „może istnieć zegar ewolucyjny molekularny”. Tak więc termin „zegar molekularny” został początkowo ukuty w celu opisania zmian w aminokwasach zachodzących proporcjonalnie do czasu od dywergencji gatunków.
od pierwszego użycia termin „zegar molekularny” był używany w wielu różnych kontekstach. Obecnie często używa się go po prostu w odniesieniu do liczby zmian lub „podstawień”, nagromadzonych w sekwencji DNA lub białek w danej linii. Liczbę podstawień na określoną jednostkę czasu można opisać jako „szybkość” zegara molekularnego, która w tym kontekście jest równoważna „szybkości ewolucji”. Zauważ, że początkowa konotacja nadal utrzymuje się w niektórych przypadkach. Na przykład, gdy ludzie stwierdzają „założyliśmy zegar molekularny” (szczególnie w analizach filogenetycznych), oznacza to, że zakłada się, że liczba substytucji występuje stale w czasie.
koncepcja stałego zegara molekularnego ma niezwykłe implikacje dla biologów ewolucyjnych. Gdyby rzeczywiście istniał stały zegar molekularny, jak pierwotnie zaproponowano, wnioskowanie o czasie zdarzeń ewolucyjnych stałoby się raczej prostym problemem(Rysunek 1). Jednak stało się jasne, że podstawienia nie występują stale w czasie w różnych liniach (Kumar 2005). Niemniej jednak koncepcja zegara molekularnego miała ogromny wpływ na dziedzinę ewolucji molekularnej. Jedną z najważniejszych idei zainspirowanych koncepcją zegara molekularnego jest neutralna teoria ewolucji molekularnej.
Zegar molekularny i neutralność
Kiedy idea stałego zegara molekularnego pojawiła się po raz pierwszy, uważano, że dominującą siłą ewolucyjną leżącą u podstaw podstawienia aminokwasów lub nukleotydów jest dobór naturalny. Zgodnie z tą linią myślenia, stały zegar molekularny wskazywałby, że adaptacyjne podstawienia u różnych gatunków występują stale w czasie. Trudno jednak wyjaśnić, w jaki sposób adaptacyjne podstawienia następowałyby w sposób podobny do zegara. Teoretycznie losy mutacji adaptacyjnych są determinowane przez kilka parametrów ewolucyjnych, takich jak siła selektywnej przewagi tej mutacji, wielkość efektywnej populacji i wskaźniki mutacji adaptacyjnych (Kimura 1983). Parametry te mogą różnić się między gatunkami, a nawet w obrębie gatunku, w zależności od specyficznych mutacji i ich interakcji ze środowiskiem.
zamiast tego Kimura (1968, 1969) zaproponował, że większość zmian na poziomie molekularnym ma niewielkie konsekwencje funkcjonalne lub jest „neutralna”. Jeśli mutacja nie ma konsekwencji przydatności, jej los w populacji jest determinowany całkowicie przez przypadek. Oznacza to, że nie możemy przewidzieć, czy konkretna mutacja neutralna zostanie ostatecznie ustalona w populacji. Jednak tempo, w jakim występują neutralne podstawienia w populacji, można przewidzieć, ponieważ zależy to od jednego parametru, a mianowicie szybkości mutacji (Kimura 1968).
wyobraźmy sobie populację z N liczbą haploidalnych osobników. Jeśli mutacje neutralne występują z szybkością u na osobę na pokolenie, całkowita liczba mutacji w jednym pokoleniu będzie N razy U. ponieważ wszystkie te nowe mutacje są neutralne, ich losy są całkowicie określone przez przypadek. Innymi słowy, wszystkie mutacje mają równe szanse na osiągnięcie fiksacji (co prowadzi do „podstawienia”). Prawdopodobieństwo, że każda nowa neutralna mutacja osiągnie fiksację, biorąc pod uwagę, że nastąpiła podstawienie, wynosi po prostu 1/N. Szybkość podstawień jest obliczana jako liczba nowych mutacji w każdym pokoleniu (Nu) pomnożona przez prawdopodobieństwo, że każda nowa mutacja osiągnie fiksację (1/N), co jest równe u. innymi słowy, dla mutacji neutralnych szybkość podstawienia jest równa szybkości mutacji!
dlatego, jeśli większość mutacji jest neutralna (jak zaproponowano w teorii neutralnej) i jeśli szybkość mutacji jest stała w czasie, substytucje powinny również występować stale w czasie. Następnie powinniśmy obserwować podobne do zegara regularne stawki podstawień na poziomie molekularnym. Kimura (1969) uznał więc obserwację względnie stałego zegara molekularnego w sekwencjach białkowych za silne wsparcie dla neutralnej teorii ewolucji molekularnej.
testowanie, jak działa neutralny Zegar molekularny
zgodnie z teorią neutralną, pytanie, czy stopy substytucji są stałe w czasie, czy nie, jest równoważne z tym, czy neutralne stopy mutacji są stałe w czasie. Z tego powodu wiele późniejszych badań koncentrowało się na analizie danych z neutralnych miejsc w celu ustalenia, czy neutralne wskaźniki mutacji są rzeczywiście stałe w czasie. Przed omówieniem teoretycznych debat nad zegarami molekularnymi omówimy krótko sposób realizacji tych badań. Analizy molekularnych zegarów białkowych również kontynuowano, ale debaty wokół zmienności molekularnych zegarów białkowych są bardzo różne od tych dotyczących neutralnych zegarów molekularnych i nie zostaną uwzględnione w tym artykule. Zainteresowani czytelnicy powinni skonsultować Gillespie (1991), Kumar (2005), Kim and Yi (2008) oraz Bedford et al. (2008).
Większość empirycznych analiz neutralnych zegarów molekularnych opiera się na twierdzeniu, że neutralne wskaźniki mutacji można wywnioskować z neutralnych wskaźników substytucji (Kimura 1968, 1969). W praktyce, każde badanie definiuje pewien typ miejsc w genomie jako neutralne miejsca, i porównuje stopy podstawienia tych miejsc między liniami.
które miejsca w genomie są naprawdę neutralne nie mogą być całkowicie określone, ale naukowcy wymyślili kilka przydatnych proxy. Przed erą sekwencjonowania genomu, większość dostępnych danych sekwencyjnych były te z kodujących białka sekwencji DNA. Badania często dzielą kodujące białka sekwencje DNA na dwa typy miejsc (Wu & Li 1985). Pierwszy typ miejsc obejmuje te, dla których jakakolwiek zmiana prowadziłaby do podstawienia aminokwasów, lub „miejsca niesynonimiczne”. Drugi typ miejsc, „miejsca synonimiczne”, obejmuje te, które kodują „zdegenerowane” pozycje w tabeli kodonowej, gdzie zmiana nie prowadzi do podstawienia aminokwasu. Na przykład TCT i TCC kodują serynę. Jeśli trzecia pozycja tych kodonów zmieni się, nadal będą produkować te same aminokwasy. Takie substytucje byłyby mniej widoczne dla doboru naturalnego. W związku z tym Zegary molekularne w miejscach synonimicznych powinny być bliżej neutralnego zegara molekularnego niż Zegary niesynonimiczne.
w miarę zaawansowania technik sekwencjonowania niektóre badania wykorzystywały introny jako proxy dla miejsc neutralnych, ponieważ nie są one włączane do dojrzałych mRNA, a zatem są bardziej prawdopodobne, że będą neutralne (Yi et al. 2002). Sekwencje nieaktywnych elementów transponowalnych, które zostały wstawione na długo przed dywergencją gatunków, były również często stosowane (są one często określane jako „powtórzenia przodków”, np. Thomas et al., 2003). Wreszcie, w niektórych badaniach wykorzystano niekodujące sekwencje DNA (wszystkie sekwencje po usunięciu kodujących białko sekwencji DNA) wyekstrahowane z całych dopasowań genomu w celu przetestowania neutralnych zegarów molekularnych (Elango et al. 2006, Huttley et al. 2007).
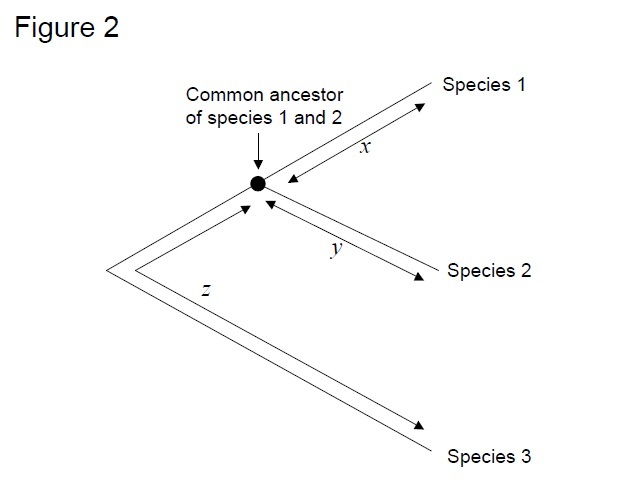
najczęściej stosowanym testem jest tzw.”test szybkości względnej”(Sarich& Wilson 1973). Początkowo, stopy substytucji na jednostkę czasu były szacowane przez podzielenie całkowitej liczby różnic (podstawień) między białkami różnych gatunków przez czas dywergencji, oszacowany na podstawie zapisów kopalnych (Fig.1). Jednak zapisy kopalne nie są dostępne dla wielu porównań i wiążą się z dużymi marginesami błędów. Test szybkości względnej pokonuje konieczność zapisów kopalnych (Rys. 2). Dopóki istnieje Sekwencja pozagrupowa do dwóch interesujących linii, możemy określić, czy dwie gałęzie podążają za tymi samymi lub różnymi zegarami molekularnymi, bez znajomości absolutnego czasu rozbieżności(ryc. 2).
jakie są wyznaczniki neutralnych zegarów molekularnych?
prawie wszystkie kontrowersje w centrum debat nad neutralnymi zegarami molekularnymi wynikają z Pytania, Jakie są główne źródła mutacji. To pytanie ma bezpośrednie znaczenie dla zrozumienia wzorców mutacji, które są ostatecznym źródłem zmian ewolucyjnych i chorób genetycznych. Ponadto, zrozumienie, jak mutacje zmieniają się między liniami i w genomach jest fundamentalnym pytaniem w genomice porównawczej, która ma na celu wykorzystanie porównań sekwencji do identyfikacji regionów genomowych, które są funkcjonalnie ważne.
więc co determinuje neutralne tempo mutacji? Jednym z najważniejszych czynników wpływających na neutralne Zegary molekularne jest specyficzna dla linii zmienność czasów generacji. Od samego początku idea stałego neutralnego zegara molekularnego była postrzegana jako sprzeczna z molekularnymi mechanizmami mutacji linii zarodkowej. Od dawna uważa się, że większość mutacji wynika z błędów w replikacji DNA w liniach zarodkowych (Haldane 1947, Muller 1954). Ponieważ mutacje występują, gdy dna linii zarodkowej jest replikowany dla następnego pokolenia, powinny one gromadzić się proporcjonalnie do liczby pokoleń, a nie bezwzględnej ilości czasu. Dlatego, jeśli porównamy liczbę podstawień, które nagromadziły się w dwóch liniach od czasu ich rozbieżności, linia z dłuższym czasem generacji, po przejściu mniejszej liczby zdarzeń replikacji DNA, zawierałaby mniej podstawień w porównaniu do linii z krótszym czasem generacji. W związku z tym zegar molekularny powinien działać wolniej u gatunków o dłuższych czasach generacji. Idea ta jest określana jako „efekt generowania czasu”.
w rzeczywistości efekt czasu generacji został po raz pierwszy zaobserwowany w badaniach naczelnych, jeszcze przed debatą na temat zegara molekularnego. Morris Goodman, który w tym czasie stosował metody immunologiczne do badania pokrewieństwa gatunków, zauważył, że szybkość, z jaką niektóre białka się rozchodzą, wydaje się zmniejszać u małp, w szczególności u ludzi, w porównaniu z małpami Starego Świata (Goodman 1961, 1962, 1963). Efekt ten jest określany jako „spowolnienie szybkości u małp człekokształtnych”. Ponieważ małpy człekokształtne mają dłuższy czas generacji niż małpy Starego Świata, obserwację tę można wyjaśnić efektem czasu generacji.
Wu & Li (1985) byli pierwszymi, którzy przetestowali hipotezę efektu czasu generacji przy użyciu danych sekwencji DNA. Wykorzystali dane z 11 genów naczelnych i gryzoni. Ponieważ naczelne mają znacznie dłuższy czas generacji niż gryzonie, zegar molekularny powinien być szybszy u gryzoni w porównaniu z naczelnymi. W rzeczywistości odkryli, że dla synonimicznych miejsc gryzonie wykazują około dwa razy większą szybkość ewolucji molekularnej w porównaniu z naczelnymi (Wu & Li 1985). Jednak w przypadku witryn niesynonimicznych taki efekt nie został znaleziony. Innymi słowy, neutralny zegar molekularny, ale nie Zegar molekularny aminokwasów, działa szybciej w linii gryzoni w porównaniu z linią naczelnych, co dobrze pasuje do idei efektu czasu generacji.
kolejne badania dostarczyły dalszego wsparcia dla spowolnienia liczby małp człekokształtnych (Li & Tanimura 1987, Bailey et al. 1991) oraz różnica w stawkach między rodami gryzoni i naczelnych (gu & Li 1992, Huttley et al. 2007). Ponadto zaobserwowano różnicę w częstości występowania w jeszcze mniejszych skalach filogenetycznych, zwłaszcza u naczelnych: na przykład, ludzki zegar molekularny działa wolniej niż zegar molekularny szympansa (Elango et al. 2006); stawki u małp nowego świata są szybsze niż stawki u małp człekokształtnych i małp Starego Świata (Steiper & Young 2006). Różne szybkości zegarów molekularnych obserwowane w tych badaniach są jakościowo zgodne z efektem czasu generacji.
jednak rzeczywiste różnice między liniami nie są ilościowo zgodne z różnicą czasów generacji. Na przykład Kumar & Subramanian (2002) wykazał, że chociaż różnica w czasie generacji naczelnych i gryzoni jest znacznie większa niż różnica między ludźmi a małpami Starego Świata, obserwowane różnice w zegarach molekularnych są podobne w tych dwóch porównaniach. Warto zauważyć, że Kumar & Subramanian (2002) użył specyficznych filtrów statystycznych do usunięcia danych, które wykazały „heterogeniczne” wzorce substytucji, które mogły spowodować stronniczość w kierunku powoli ewoluujących sekwencji (Yi et al. 2002). Niemniej jednak różnica w zegarach molekularnych naczelnych i gryzoni wydaje się znacznie mniejsza niż pierwotnie zaproponowana przez Wu & Li (1985). Na przykład Huttley et al. (2007) przeanalizował całe dopasowanie genomu kilku gatunków, w tym oposa, i wykazał, że różnica w tempie między liniami euteriańskimi a liniami oposa (~30%) jest znacznie większa niż różnica w tempie między liniami ludzkimi a mysimi (~14%). Przykłady te pokazują, że stopień różnic w zegarach molekularnych różni się znacząco w różnych badaniach ze względu na różnice w zestawach danych i metodach statystycznych. Pokazują one również, że różnice w stawkach między liniami nie mogą być w pełni uwzględnione jedynie przez różnicę czasów generacji. Oczywiście są też inni współtwórcy neutralnych zegarów molekularnych.
rzeczywiście, cechy historii życia inne niż czasy generacji wydają się współgrać z zegarami molekularnymi. Martin & Palumbi (1993) wykazał, że zegary molekularne DNA działają szybciej u gatunków o niewielkich rozmiarach ciała. Ta obserwacja doprowadziła do hipotezy, że tempo przemiany materii jest ważnym wyznacznikiem zegarów molekularnych. Wysoki metabolizm wytwarza dużą liczbę mutagennych rodników tlenowych, które zwiększają szybkość mutacji (Rand 1994). Ponieważ tempo metabolizmu i wielkość ciała na ogół zmieniają się wraz z czasem generacji, trudno było odróżnić, który z nich stanowi główny wyznacznik zegarów molekularnych. Tsantes & Steiper (2009) zaproponował, na podstawie danych naczelnych, że wiek przy pierwszej reprodukcji, a nie rozmiar ciała, jest głównym wyznacznikiem zegarów molekularnych. Ponieważ wiek w pierwszej reprodukcji odzwierciedla efekt czasu generacji, badanie to popiera ideę, że efekt czasu generacji jest głównym wyznacznikiem zegara molekularnego. Jednak badania te nadal opierają się na ograniczonej liczbie linii (wykorzystano cztery pary gatunków). Tak więc rozróżnianie między wielkością ciała, efektem czasu generacji i tempem metabolizmu nadal pozostaje ważną kwestią w uogólnianiu i zrozumieniu neutralnych zegarów molekularnych.
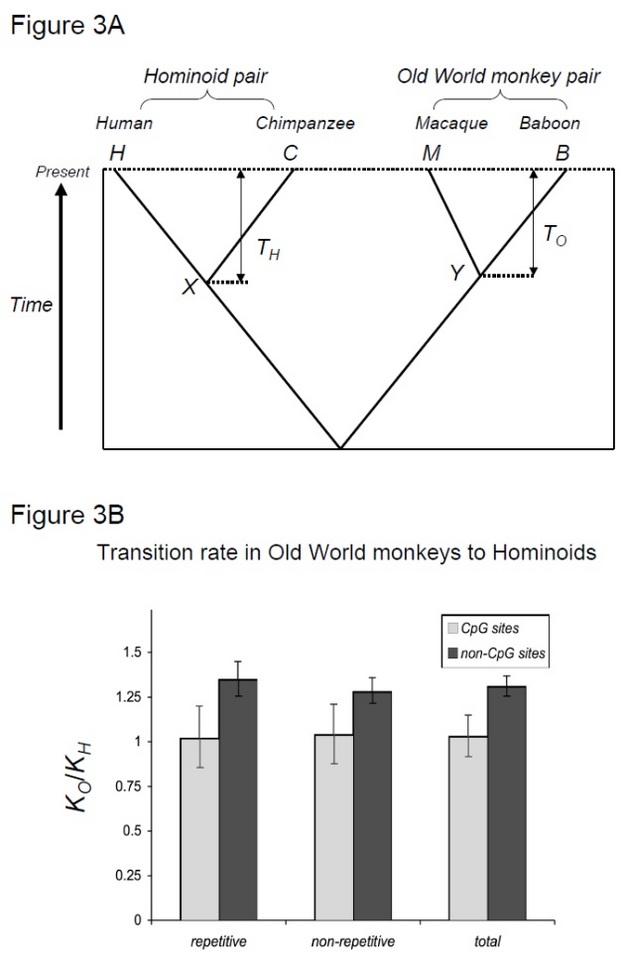
ponadto coraz częściej doceniane jest znaczenie czynników, które nie zmieniają się wraz z czasem generacji, takich jak metylacja DNA. Metylacja DNA jest chemiczną modyfikacją genomowego DNA występującą w różnych taksonach. W genomach zwierzęcych metylacja DNA zachodzi prawie wyłącznie w cytozynach, a następnie guaninach (tzw. „CpG”). Z kolei metylowane cytozyny mają tendencję do szybkiej mutacji w tyminach z powodu niestabilności chemicznej (Bird 1980). Rzeczywiście, w ludzkim genomie mutacje spowodowane metylacją DNA występują częściej niż o rząd wielkości niż inne mutacje (Nachman& Crowell 2000, Elango et al. 2008). Ponieważ mutacje spowodowane metylacją DNA występują w dużej mierze niezależnie od replikacji DNA, takie mutacje mogą następować po innych zegarach molekularnych niż inne. W szczególności, zamiast zależności od czasu generacji, mutacje spowodowane metylacją DNA mogą następować po zależnym od czasu zegarze molekularnym, który jest podobny do tego, co pierwotnie zaproponował Zuckerkandl & Pauling (Kim et al. 2006)!
aby przetestować tę hipotezę, Kim et al. (2006) porównał dywergencję człowieka-szympansa do dywergencji makaka-pawiana, dwóch par gatunków, które mają podobne czasy dywergencji, ale z różnymi czasami generacji (Steiper et al. 2004). Para człowiek-szympans (para małp człekokształtnych) ma znacznie dłuższy czas generacji w porównaniu do pary Makak-pawian (para małp Starego Świata). Badanie to wykazało, że w przypadku miejsc innych niż CpG, para małp Starego Świata zgromadziła około 30% więcej podstawień, co można wyjaśnić wspomnianym wcześniej efektem spowolnienia szybkości u małp człekokształtnych. W przeciwieństwie do tego, Zegary molekularne w miejscach CpG wykazały podobną liczbę podstawień w parach małp człekokształtnych i małp Starego Świata(ryc. 3). Tak więc Zegary molekularne zależne od czasu i zależne od czasu generacji współistnieją w tych samych genomach. Założenie, że dla danej linii może istnieć pojedynczy zegar molekularny, nie jest już aktualne, ponieważ dominujące siły mutacyjne różnią się między regionami genomu.
wnioski
koncepcja stałego zegara molekularnego została początkowo zaproponowana w oparciu o ograniczoną ilość danych o sekwencji białek. Mimo że późniejsze badania wykazały, że taka obserwacja nie jest ogólnym wzorcem w aminokwasach, miała znaczący wpływ na dziedzinę ewolucji molekularnej, w szczególności na rozwój neutralnej teorii ewolucji molekularnej dla danych o sekwencji DNA. Zgodnie z teorią neutralną, badania koncentrowały się na wyjaśnieniu wzorców zmienności w szybkości mutacji neutralnych. W ciągu ostatnich kilku dekad zaobserwowaliśmy, że zegary molekularne działają w różnym tempie między liniami. Ponadto stopień zmienności może się różnić w zależności od różnych rodzajów danych i konkretnych stosowanych metod statystycznych. Efekt czasu generacji utrzymuje się na poziomie jakościowym, ale jest niewystarczający do wyjaśnienia ilościowej zmienności neutralnych szybkości mutacji wśród linii. Cechy historii życia i niezaleĺźne od replikacji mutacje, takie jak te spowodowane metylacjä … DNA, sÄ … rĂłwnieĹź waĺźnymi przyczynkami genomowych zegarăłw molekularnych. Rzeczywiście, różne typy zegarów molekularnych są obserwowane nawet w obrębie genomu, ponieważ dominujące wejścia mutacyjne różnią się między różnymi regionami genomu. Tak więc, zamiast zakładać pojedynczy neutralny zegar molekularny dla każdego genomu, przyszłe badania powinny mieć na celu ujawnienie zmienności genomowych neutralnych zegarów molekularnych, aby dowiedzieć się o genomowych krajobrazach mutacyjnych. Takie informacje są nie tylko przydatne do zrozumienia surowca regulującego ewolucję molekularną i choroby genetyczne, ale również stanowią krytyczny Składnik wpływający na porównawcze i funkcjonalne analizy genomu w celu identyfikacji funkcjonalnych regionów genomowych.