wprowadzenie
międzynarodowa polityka ochrony uznaje różnorodność biologiczną na trzech poziomach: ekosystem, gatunek i genetyka, i że zarządzanie powinno zmierzać do zachowania wszystkich trzech (Konwencja o różnorodności biologicznej, 2007). Znajduje to wyraźne odzwierciedlenie w Konwencji o różnorodności biologicznej, Aichi Biodiversity Targets, uzgodnionej w 2010 r. (https://www.cbd.int/sp/targets/), gdzie w celach i celach znajduje się szczególne odniesienie nie tylko do ekosystemów i gatunków, ale także do różnorodności genetycznej. Jednak obecne podejście do ochrony bioróżnorodności opiera się w dużej mierze na obszarach geograficznych, ekosystemach, społecznościach ekologicznych i gatunkach, z mniejszą uwagą na różnorodność genetyczną i kontinuum gatunkowo-populacyjne. W rzeczywistości większość praktyków zajmujących się ochroną przyrody skupia się na gatunkach jako metryce różnorodności biologicznej, z pewnym uwzględnieniem jednostek wewnątrzspecyficznych w ramach intensywnie zarządzanych gatunków zagrożonych. W tym kontekście naturalne jest dążenie do stosowania prostych kategoryzacji-nazwanych gatunków i innych nazwanych taksonów (tj. podgatunków), a czasami jednostek ochrony w obrębie gatunków. Gatunki w szczególności zapewniają wspólną miarę różnorodności biologicznej, która stanowi podstawę wielu przedsięwzięć naukowych i zarządzania (Mace, 2004; Wilson, 2017). Jako podstawowa jednostka ochrony, gatunki są często wykorzystywane do określenia wartości bioróżnorodności poprzez wykazy gatunków na obszarach chronionych, identyfikację gatunków zagrożonych w ramach jurysdykcji oraz jako podstawa procedur bezpieczeństwa biologicznego. Ponadto gatunki są zazwyczaj podmiotami, z którymi komunikujemy społeczeństwu kwestie ochrony.
ten impuls do prostego identyfikowania jednostek dyskretnych w przyrodzie jest sprzeczny z kilkoma, pokrewnymi rzeczywistościami. Po pierwsze, specjacja nie jest natychmiastowa. Raczej jest to typowo długotrwały proces (Dynesius and Jansson, 2014; Roux et al., 2016), przechodząc od izolowanych populacji, być może podlegających różnym presjom selekcji, do jednoznacznych gatunków w długich skalach czasowych, z częstymi odwróceniami spowodowanymi zmianą środowiska lub prostym połączeniem genetycznym w miarę ponownego łączenia się zakresów (Rysunek 1; Seehausen et al., 2008; Rosenblum et al., 2012). Po drugie, najbardziej uznane gatunki nie są genetycznie jednolite. Rozbieżność (dryf i przedłużająca się izolacja) i retikulacja (przepływ genów, hybrydyzacja, introgresja i rekombinacja) skutecznie kształtują zróżnicowanie geograficzne i genomowe w obrębie gatunków (Cutter and Payseur, 2013; Edwards et al., 2016), a niektóre gatunki mogą być zorganizowane w długie izolowane populacje, z których niektóre mogą uzasadniać uznanie za pełne, ale być może morfologicznie tajemnicze gatunki (Bickford et al., 2007; Jorger i Schrödl, 2013). Po trzecie, tak długo uznawane, nazwane gatunki nie są równoważne pod względem tego, jak wiele ewolucyjnego Dziedzictwa reprezentują-mówiąc prościej, dziobak (jako przedstawiciel starożytnie rozbieżnej linii) może być uznany za wartego więcej niż niedawno rozbieżny gryzoń(Isaac et al., 2007). Po czwarte, oznaczenia gatunków nie są odlewane w kamieniu, a raczej są hipotezami, które należy przetestować, gdy nowe dowody staną się dostępne (Hey et al., 2003). Każda z tych kwestii stwarza złożoność i niejednoznaczność w wytyczaniu gatunków w ogóle i przypisywaniu im lub ich składowym populacjom uwagi ochronnej. Ponadto wytyczanie gatunków podlega również różnym pojęciom gatunkowym i sposobom ich diagnozowania, co wywołało wielką konsternację wśród biologów ochrony (Mace, 2004; Frankham et al., 2012; Garnett and Christidis, 2017).
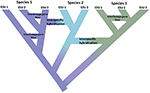
Rysunek 1. Specjacja jako kontinuum przechodzące od izolowanych populacji do jednoznacznych gatunków pokazanych przez zmianę koloru z kolorami reprezentującymi różne gatunki. Niektóre linie, które powstają w wyniku izolacji populacji, rozwijają się w prawdziwe gatunki z wewnętrznymi węzłami drzewa reprezentującymi inicjację procesu specjacji. Inne linie nie rozwijają się w prawdziwe gatunki, ale z czasem się rozchodzą, co wskazuje, że gatunki nie są genetycznie jednolite. Linie te są pokazane jako ewolucyjnie znaczące jednostki (ESUs). Przepływ genów międzyliniowych jest wskazany dla gatunku 1 I gatunku 3. W tym przypadku niezależność ewolucyjna może być przejściowa, ponieważ dwie linie należące do tego samego gatunku nie są reprodukcyjnie izolowane. Podczas gdy pozostają one jako odrębne linie (gatunki), międzygatunkowa hybrydyzacja może wystąpić z ograniczoną wymianą genów (zmiana koloru) między gatunkami.
wdrożenie skutecznych strategii ochrony, które mają na celu ochronę zarówno gatunków, jak i różnorodności genetycznej, przyniosłoby korzyści zarówno dzięki bardziej spójnemu spojrzeniu na sposób, w jaki ograniczamy gatunki i inne jednostki w kontinuum populacji-gatunków, jak i w jaki sposób jednostki te są interpretowane w różnych przepisach na całym świecie. Nowe możliwości genomiki stwarzają zarówno szanse, jak i wyzwania dla osiągnięcia tych celów. Poniżej rozważamy: (i) w jaki sposób analizy w skali genomu można pogodzić z konserwatywnym podejściem do wytyczania granic i nazewnictwa gatunków, jednocześnie identyfikując główne składniki różnorodności genetycznej w obrębie gatunków; (ii) w jaki sposób istniejące prawodawstwo dotyczące ochrony na kilku kontynentach, które definiuje „gatunki” jako szeroki zakres kategorii specyficznych dla podczerwieni, może być stosowane, biorąc pod uwagę wysoką rozdzielczość oferowaną przez ekrany genomu; oraz (iii) w jaki sposób wgląd w procesy specjacji i strukturyzację zmienności z genomiki jest zgodny z prawodawstwem, a następnie z zarządzaniem. Następnie proponujemy kilka sposobów dostosowania jednostek ochrony do podejść legislacyjnych w celu ochrony pełnego spektrum różnorodności od populacji po gatunki.
genomika, specjacja i taksonomia
podobnie jak wiele dziedzin nauk biologicznych, Systematyka, praktyka nazywania taksonów, rozumienia ich związków i sposobu ich formowania, jest znacznie zaawansowana przez genomikę i modele statystyczne. Czy przez całe sekwencjonowanie genomu (np Nater et al., 2017) lub próbkowanie genomu (SNPs, target capture-sequencing; Jones and Good, 2016; Leache and Oaks, 2017), mamy teraz bezprecedensową rozdzielczość wzorców różnorodności genetycznej (Funk et al., 2012). W połączeniu z coraz silniejszymi modelami statystycznymi do wnioskowania historii dywergencji genomowej i gatunkowej, dane te dostarczają nowych wglądów w procesy ewolucyjne, które generują gatunki i zmienność genetyczną.
zastosowanie tych nowych narzędzi generuje dwa przeciwstawne spostrzeżenia. Z jednej strony wiele badań ujawnia wysoce rozbieżne populacje genetyczne w obrębie nazwanych gatunków, tak zwanych gatunków tajemniczych(Bickford et al., 2007; Jorger i Schrödl, 2013; Struck i in., 2017). Nie jest to nic nowego, ponieważ opiera się na długiej historii genetyki przestrzennej populacji i filogeografii molekularnej, która była produktywnym przedsięwzięciem przez trzy dekady (Avise et al., 1987). To, co genomika wnosi do tego przedsięwzięcia, to zarówno zwiększona rozdzielczość struktury populacji, jak i dane wystarczające do statystycznie wnioskowania historii populacji i testowania alternatywnych modeli rozbieżności (Degnan and Rosenberg, 2009; Sukumaran and Knowles, 2017). Z drugiej strony, analizy w skali genomu ujawniają również, że (tak długo jak sądzono w przypadku roślin) wymiana genetyczna (introgresja) wśród gatunków zwierząt jest bardziej powszechna niż wcześniej sądzono, zarówno podczas specjacji, jak i po specjacji, a nawet może napędzać nowe promieniowanie adaptacyjne (Mallet, 2007; Rieseberg and Willis, 2007; Arnold and Kunte, 2017) Rysunek 1. Podsumowując, te spostrzeżenia dodatkowo podkreślają, że specjacja jest przedłużona (Roux et al., 2016) i ujawniają przeciwstawne siły działające poprzez filogeografię-kontinuum filogenetyki (Edwards et al., 2016).
Jak to wszystko ma się do celów taksonomii i ochrony? Istnieje silna i uzasadniona obawa, że zwiększona rozdzielczość zapewniana przez ekrany genomu może prowadzić do szalejącego nadmiernego podziału gatunków(Isaac et al., 2004), potencjalnie ograniczając elastyczność zarządzania i wysyłając małe genetycznie rozbieżne populacje do chowu wsobnego i ostatecznego wymierania (Frankham et al., 2012). Odwrotnie, Gippoliti et al. (2018) argumentują, że bezwładność taksonomiczna, skutkująca wcześniejszym brakiem rozpoznania wielu gatunków afrykańskich kopytnych, doprowadziła do depresji outbredowej po wymieszaniu i braku ochrony geograficznie ograniczonych taksonów. Ich widok (Zobacz także Morrison et al., 2009) jest to, że nie ma dowodów na to, że inflacja taksonomiczna ma negatywny wpływ na ochronę kopytnych (Gippoliti et al., 2018). Niepokojące jest to, że niestabilna Taksonomia opóźnia zarówno praktykę ochrony, jak i postrzeganie publiczne (Garnett and Christidis, 2017). Obawy te są częściowo napędzane przez długotrwałą debatę wokół pojęć gatunków i metod diagnostycznych w biologii systematycznej, w szczególności zastosowanie koncepcji gatunków filogenetycznych, która utrzymuje, że gatunki są jednostkami minimalnie diagnozowalnymi (Cracraft, 1983). Jednak większość taksonomistów jest z natury konserwatywna, nazywając gatunki tylko wtedy, gdy istnieje zgodność między wieloma liniami dowodów (Dayrat, 2005; Padial et al., 2010; Schlick-Steiner et al., 2010: Yeates et al., 2011). Uważamy, że gatunki powinny reprezentować solidne, niezależnie rozwijające się linie, które pozostają w dużej mierze nienaruszone w kontakcie z bliskimi krewnymi—zgodnie z intencją koncepcji ewolucyjnych gatunków (Wiley, 1978) i uogólnionej koncepcji gatunków Lineage (de Queiroz, 1998, 2007). W praktyce wymaga to bezpośrednich dowodów izolacji reprodukcyjnej lub rozsądnych surogatów tego samego, jednocześnie pozwalając na ograniczoną wymianę genetyczną(Singhal et al., 2018).
dane genomowe zapewniają większą niż wcześniej moc identyfikacji różnicowania i dywergencji w obrębie i między gatunkami, wymagając większego rozważenia tego, jak wpływa to na wyznaczanie jednostek taksonomicznych. W przypadku, gdy populacje są zarówno silnie różniące się genetycznie, jak i fenotypowo w stosunku do już nazwanych gatunków, istnieje silny argument, aby nazwać je odrębnymi gatunkami. Jeśli populacje są silnie rozbieżne fenotypowo z niewielką separacją genomową, istnieje wezwanie do oceny, czy te różnice fenotypowe są (i) dziedziczne i (ii) mogą powodować materialną izolację rozrodczą. Skany genomu mogą rozwiązać takie sytuacje, testując ograniczony przepływ genów. Trudniejszym zagadnieniem jest to, gdzie morfologicznie podobne, ale genetycznie rozbieżne populacje są wykrywane. Takie populacje mogą być łatwo diagnozowane jako oddzielne linie ewolucyjne poprzez zastosowanie statystycznych metod delimitacji do danych multilocus (Carstens et al., 2013; Rannala, 2015), ale czy są to efemeryczne populacje czy trwałe gatunki (Sukumaran and Knowles, 2017)? Z pewnością jest możliwe, w ramach zasad nomenklatury, rozpoznawanie gatunków na podstawie samych sekwencji DNA, zapewniając diagnostykę opartą na sekwencjach (Tautz et al., 2003; Cook et al., 2010). Na przykład Murphy et al. (2015) Wytypuj i opisz sześć Australijskich gatunków amfipodów Desert spring w oparciu o dane sekwencyjne DNA, pomimo braku wyraźnych różnic morfologicznych. Jest to jednak ogólny obszar do poprawy, w którym systematycy muszą stosować termin „tajemnicze gatunki” z większą konsekwencją i rygorem (Struck et al., 2017).
w sytuacjach, w których dane o sekwencji DNA stanowią potencjalną kluczową cechę rozpoznawania gatunku, sugerujemy, że szczególnie ważne jest zastosowanie innych linii dowodów, aby uniknąć nadmiernego rozdzielania (Oliver et al., 2015). Jednym z podejść jest zbadanie znacznej izolacji reprodukcyjnej, w której takie populacje tworzą naturalne strefy kontaktu. Na przykład, w porównawczej analizie genomicznej stref kontaktu między parami linii jaszczurek Carlia, które są fenotypowo nie do odróżnienia, wskaźniki izolacji genetycznej były silnie skorelowane z czasem dywergencji (Singhal i Moritz, 2013; Singhal i Bi, 2017) (Rysunek 2). Bardziej rozbieżne z tych par linii zostały obecnie formalnie uznane za oddzielne gatunki, podczas gdy bliżej spokrewnione (jeszcze statystycznie diagnozowalne) linie z dowodami genomicznie rozległej wymiany genetycznej w różnych strefach kontaktu nie były (Singhal et al., 2018). Biorąc pod uwagę silny empiryczny związek między rozbieżnością a izolacją reprodukcyjną u tych taksonów, autorzy ci dodatkowo oddzielili dwie allopatyczne i tajemnicze Izolaty (blisko spokrewnionych gatunków), które miały jeszcze większą rozbieżność genomową niż te określone powyżej. Rozszerzenie tej izolacji rozrodczej na inne gatunki o głęboko rozbieżnych, ale morfologicznie podobnych liniach filogeograficznych (Potter et al., 2016, 2018; Afonso Silva et al., 2017a) sugeruje, że do opisania pozostaje więcej gatunków, zwłaszcza na wyspach (Rysunek 2, Carlai amax i C. rufilatus), a nawet niektóre z tych gatunków zostały opisane po komplementarnej analizie fenotypowej (Afonso Silva et al.,2017b). Inne linie z nowszymi rozbieżnościami, ale nadal statystycznie rozdzielone jako oddzielne linie przy użyciu dużego zestawu genów jądrowych, mogą być reprezentowane jako ewolucyjnie znaczące jednostki (ESUs) w obrębie gatunku.
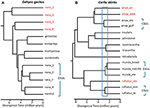
Rysunek 2. Phylogenomic resolution of species boundaries in lizards from the Australian Monsoonal Tropics (AMT). W każdym przypadku relacje i względne czasy rozbieżności są szacowane na 100 egzonów przy użyciu wielogatunkowego koalescencji zaimplementowanej w StarBEAST2, ale skale czasowe między tymi dwoma przykładami niekoniecznie są takie same. A) wyniki dla grupy Nana Gehyra geckos (Moritz et al., 2018). Różnorodność filogenetyczna linii w obrębie G. nana, jak wcześniej stwierdzono, obejmowała cztery inne już opisane i morfologicznie wyróżniające się gatunki. Późniejsze analizy pokrywania się zasięgu i morfologii doprowadziły do uznania każdej z najbardziej zróżnicowanych filogenetycznie linii (czerwone etykiety taksonów) za odrębne gatunki (Doughty et al., 2018). (Morfologicznie nierozłączne) linie pozostające u G. nana w obecnej formie można uznać za ESUs. B) bieżące ustalanie granic gatunków w Carlia skinks z AMT. Niektóre linie zostały już uznane za gatunki na podstawie kombinacji genetycznej rozgraniczenia gatunków i post hoc identyfikacji diagnostycznych różnic morfologicznych (C. insularis i C. isostriacantha; Afonso Silva et al.,2017b). Dla innych kompleksów (Potter et al., 2016, 2018), trwają rewizje taksonomiczne. Niebieski prostokąt wskazuje zakres czasów dywergencji, w których zaobserwowano znaczną izolację rozrodczą, skorelowaną z dywergencją, w strefach kontaktu między morfologicznie tajemniczymi liniami pokrewnych gatunków (Singhal and Bi, 2017). Zachowawczo i po (Singhal et al., 2018), uznajemy jako gatunek kandydujący trzy linie genetyczne o rozbieżnościach większych niż ten próg empiryczny. Pozostawiłoby to genetycznie odrębny ESUs w obrębie każdej z Carlia amax, Carlia munda i Carlia rufilatus (niebieskie nawiasy). Paski skali przedstawiają szacunki czasu rozbieżności w milionach lat.
w skrajnych przypadkach metody genomiczne ujawniają, że pojedyncze gatunki taksonomiczne nie tylko są heterogeniczne genetycznie, ale w sensie filogenetycznym zawierają również inne nazwane gatunki. Taka „parafilia” może powstać poprzez niedawną specjację z jednego, genetycznie podzielonego gatunku (Patton and Smith, 1994), ale generuje również hipotezę, że genetycznie heterogeniczny takson może reprezentować wiele gatunków. Ponownie, więcej dowodów jest wymagane. Jako przykład, analiza filogenomiczna kompleksu jaszczurek Gehyra gecko z Północnej Australii wytyczyła dziewięć linii ewolucyjnych, z czterema już nazwanymi gatunkami filogenetycznie osadzonymi wśród nich (Moritz et al., 2018; Rys. 2). Późniejsze dowody nakładania się geograficznego (przy zachowaniu podziału filogenetycznego)i subtelnych różnic morfologicznych wspierały nazewnictwo czterech najbardziej rozbieżnych filogenetycznie linii (Doughty et al., 2018). Odwrotnie, bliżej spokrewnione (ale statystycznie rozdzielne) linie miały raczej sąsiednie niż nakładające się dystrybucje, ale nie były morfologicznie diagnozowalne i w związku z tym zostały zachowane jako pojedynczy gatunek (G. nana sensu stricto), aczkolwiek z wieloma ESUs, do czasu dalszych badań (ryc. 2). Kolejne skany genomu próbek przekraczających granice tych ESUs wykazały, że przynajmniej niektóre mają znaczną wymianę genetyczną, potwierdzającą oryginalne, konserwatywne podejście do rozpoznawania gatunków.
w innych przypadkach, badania genomu roślin, nawet poprzez prostą ordynację SNP, mogą ujawnić stark i nieprzewidziane rozbieżności między próbkami populacji, sugerując obecność tajemniczych taksonów, które mogą być następnie analizowane pod kątem diagnostycznych znaków fenotypowych. Na przykład, oparte na SNP dowody na rozbieżność genomową między populacjami i różnice ekotypowe w Eucalyptus salubris ujawniły dwie odrębne linie molekularne, które utrzymywały swoją odrębność w obszarze pokrywania się geograficznego, z adaptacją ekotypową uważaną za ważny czynnik w minimalizowaniu przepływu genów między liniami. (Steane et al., 2015; rys. 3). W innym przypadku, w australijskim pelargonie, analiza genomiczna, w połączeniu z badaniami morfologicznymi i reprodukcyjnymi, doprowadziła do istotnych zmian w taksonomii, zarówno podziału, jak i liftingu taksonów, zwłaszcza tam, gdzie wcześniej używane cechy morfologiczne okazały się labilne i niezgodne z filogenezą (Nicotra et al., 2016).
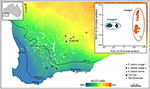
Rysunek 3. Analiza genomiczna z wykorzystaniem danych SNP zidentyfikowała tajemnicze linie u rozpowszechnionego eukaliptusa salubrisa. Lokalizacja próbek populacji każdej linii pokazano w gradiencie wskaźnika suchości w południowo-zachodniej Australii Zachodniej. PCoA oparty na binarnych odległościach genetycznych pochodzących z 16 122 markerów dartseq, wykazuje dwie odrębne linie. Adaptacja z Steane et al. (2015).
powyższe przypadki ilustrują progresywne podejście do testowania hipotez, które zostało wykorzystane w taksonomii integracyjnej (Padial et al., 2010; Yeates et al., 2011). Dalej, te skrzynki używać jakaś forma Genom redukcja sekwencjonowanie uzyskiwać wysokiej jakości DANE od 1000 ’ s niezależny geny, dawać dużo większy zaufanie w rezultat niż jest w przypadku używać tylko plastid loci lub kilka jądrowy geny. Przykładem tego, jak zastosowanie integracyjnej taksonomii może radykalnie zmienić postrzeganie różnorodności, jest połączone badanie genetyczne, akustyczne i morfologiczne żab z Madagaskaru, które prawie podwoiło znaną różnorodność gatunkową (Vieites et al., 2009). Autorzy Ci zaproponowali szereg pojęć odzwierciedlających stan uporządkowania taksonomicznego i zbieżności: Potwierdzone gatunki kandydujące do genetycznie i fenotypowo rozbieżnych, ale jeszcze nie nazwanych taksonów; niepotwierdzone gatunki kandydujące do genetycznie rozbieżnych taksonów, w których morfologia i akustyka nie zostały jeszcze zbadane; oraz głęboko spokrewnione linie dla Bytów, o których wiadomo, że są genetycznie rozbieżne, ale fenotypowo tajemnicze. Analizy genomowe nie zostały jeszcze zastosowane do tych taksonów, ale z pewnością poinformowałyby o decyzjach dotyczących linii spokrewnionych w porównaniu z kandydującymi gatunkami.
podejście genomowe, nawet przy zastosowaniu zachowawczym, bez wątpienia doprowadzi do dalszego podziału morfologicznie konserwatywnych taksonów. Ale odwrotnie, istnieją wyraźne przykłady, w których te same podejścia ujawniają nadmiernie podzielone lub nieprawidłowo zdiagnozowane gatunki(Nicotra et al., 2016). Oczywiście istnieje już wiele badań molekularnych nad kompleksami gatunków roślin i zwierząt w oparciu o mniejsze zbiory danych (np., microsatellite, mtDNA i cpDNA), które zidentyfikowały, gdzie gatunki zostały błędnie zdiagnozowane lub gdzie wiele gatunków zostało określonych taksonomicznie, ale są obecnie uważane za jeden gatunek. Na przykład, Rabosky et al. (2014) wykorzystał mitochondrialne i jądrowe filogenezy genów do rewizji kompleksu scincid Ctenotus inornatus, powodując redukcję netto gatunków i dostosowanie granic gatunków; cechy morfologiczne stosowane wcześniej, podczas gdy wygodne dla biologów polowych, były wysoce labilne i w dużej mierze nie pasowały do granic gatunków zdiagnozowanych genetycznie. W roślinach niektóre z najbardziej znanych przykładów znajdują się w storczykach, takich jak Europejski rodzaj orchidei Ophrys i australijski rodzaj orchidei Caladenia, gdzie badania molekularne wskazują na znacznie mniej osobników, które byłyby rozpoznawane jako gatunki, niż te oparte na cechach morfologicznych i pseudopulacyjnych zespołach zapylania (patrz Devey et al., 2008; Swarts et al., 2014).
dane w skali genomu, interpretowane zachowawczo i idealnie z równoległymi analizami fenotypów, będą nadal prowadzić do zmian taksonomicznych, zwłaszcza w taksonach morfologicznie konserwatywnych (Fišer et al., 2018). Jednak, w przeciwieństwie do obaw wyrażanych przez niektórych (Garnett and Christidis, 2017), uważamy, że jest to właściwe dążenie do ewolucyjnej systematyki, nawet jeśli wyniki są chwilowo niewygodne.
Genomics and Intraspecific Conservation Units
chociaż mniej kontrowersyjne niż taxonomic delimitation and the species level debate, ale pod wieloma względami tak samo złożone, było znaczącą dyskusją nad definicją i wytyczeniem jednostek ochrony jako ważnych elementów różnorodności wewnątrzgatunkowej, które wymagają uwzględnienia w działaniach ochronnych (Ryder, 1986; Waples, 1991; Dizon et al., 1992; Moritz, 1994; Vogler and de Salle, 1994; Crandall et al., 2000; Fraser and Bernatchez, 2001; Avise, 2004; Funk et al., 2012) (rysunek 4). Biorąc pod uwagę ostrożne podejście do rozpoznawania gatunków na podstawie dowodów genomicznych, które opowiadamy się powyżej (patrz także Oliver et al., 2015; Singhal et al., 2018), jest nieuniknione, że niektóre gatunki taksonomiczne nadal będą zawierać długie izolowane populacje w ich obrębie. Koncepcje takie jak Evolutionarly Significant Units (ESUs) lub Designatable Units (DUs) zostały zaproponowane w celu uzupełnienia istniejącej taksonomii (Ryder, 1986; Waples, 1991; Moritz, 1994; Mee et al., 2015). Podobnie jak w przypadku gatunków, interpretacje i kryteria są różne, ale wspólnym zamiarem jest rozpoznanie głównych elementów różnorodności wewnątrzgatunkowej dla działań ochronnych (Funk et al., 2012). Moritz (1994, 2002) opowiadał się za uproszczonym, ale praktycznym podejściem molekularnym do identyfikacji niezależnie ewoluujących segmentów w obrębie gatunków, „historycznej” osi różnorodności; podczas gdy inni kładą większy nacisk na „adaptacyjną” oś obejmującą różnorodność funkcjonalną (Ryder, 1986; Crandall et al., 2000; Fraser and Bernatchez, 2001; Waples et al., 2001; Mee et al., 2015) (Rysunek 4). Niezależnie od względnych zalet tych alternatyw, oczywiste jest, że techniczne i analityczne narzędzia genomiki populacji mogą teraz solidnie zidentyfikować oba składniki różnorodności wewnątrzgatunkowej(Funk et al., 2012; Hoban et al., 2013; Whitlock, 2014; Yang and Rannala, 2014).
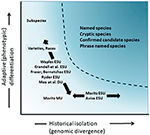
Rysunek 4. Podział różnorodności genetycznej na dwa składniki: zmienność adaptacyjna, która powstaje poprzez dobór naturalny, i assayed choć analiza fenotypów i historycznej izolacji lub neutralnej dywergencji oceniane przez dywergencji genomowej. Ciemniejszy zacieniony obszar powyżej krzywej wskazuje warunki, w których populacje są prawdopodobnie uważane za odrębne gatunki w większości pojęć. W odniesieniu do dwóch osi różnorodności genetycznej (za Moritzem, 2002) przedstawiono różne koncepcje jednostek konserwatorskich i kategorie taksonomiczne podrzędne.
To prowadzi do pytania: dlaczego po prostu nie nazwać Esusa podgatunkiem? Podgatunki tradycyjnie były definiowane i postrzegane przez botaników i zoologów jako fenotypowo odrębne, allopatyczne zestawy populacji, które mogą przenikać się ze sobą w granicach geograficznych (Mayr, 1942; Grant, 1981). Jako takie potencjalnie odzwierciedlają rozbieżność adaptacyjną, niekoniecznie będąc historycznie odizolowanymi. Nazwy podgatunków są szeroko stosowane u roślin, z różnicami geograficznymi i ekologicznymi podstawowymi środkami, za pomocą których są wyróżniane (Hamilton and Reichard, 1992). W zakresie, w jakim podgatunki wykazują lokalną adaptację, z lub bez ciągłego przepływu genów, sugerujemy, że fenotypowo zdefiniowane podgatunki powinny być nadal rozpoznawane w ochronie, a nie zestawiane z ESUs, a przynajmniej nie ESUs, jak zdefiniowano jako reprezentujące historycznie izolowane populacje.
w taki sam sposób, jak genomika populacji zapewnia znacznie większą precyzję w identyfikacji ESUs poprzez wykorzystanie zarówno neutralnych, jak i adaptacyjnych loci, zapewnia również środki do wykorzystania dużej liczby neutralnych loci do łatwiejszego wyznaczania jednostek zarządzania (MUs) jako demograficznie niezależnych jednostek charakteryzujących się ograniczonym przepływem genów (Funk et al., 2012). Podczas gdy większość naszej uwagi skupia się tutaj na ESU i analogicznych jednostkach, takich jak DUs, które mogą być uznane za główne wewnątrz-specyficzne jednostki ochrony i zostały przyjęte do ochrony w niektórych jurysdykcjach, mus zostały również zidentyfikowane jako niezależne demograficznie zestawy populacji (patrz Palsbøll et al., 2007), które można utrzymać w większym ESU (Moritz, 1994; Funk et al., 2012). Jednostki zarządzania mogą być ważne w monitorowaniu i zarządzaniu populacjami gatunków i były również określane jako „stada”, szczególnie istotne dla zarządzania rybołówstwem (Avise, 2004; Laikre et al., 2005). Definicja MUs przez Moritza (1994) jako „populacje ze znaczną rozbieżnością częstotliwości alleli w loci nuklearnych lub mitochondrialnych, niezależnie od filogenetycznej odrębności alleli” jest ogólnie powszechnie akceptowana, chociaż, jak wskazali Palsbøll et al. (2007), łączność demograficzna jest kluczowym czynnikiem Nie poziom historycznego przepływu genów.
wyznaczenie jednostek ochrony jako ważnych elementów różnorodności wewnątrzgatunkowej ma również znaczące implikacje dla ratowania genetycznego małych populacji wsobnych poprzez przywrócenie przepływu genów lub augmentację i staje się coraz bardziej jasne, że ratowanie genetyczne musi być rozważane szerzej, jeśli Należy zapobiec zwiększonemu wymieraniu populacji (Love Stowell et al., 2017; Ralls et al., 2018). Moritz (1999) zaproponował, że mieszanie MUs, ale nie ESUs, można uznać za odpowiednią strategię ratowania genetycznego. Jak podkreślają Frankham et al. (2012) mieszanie taksonomicznie uznanych gatunków nie byłoby ogólnie uważane za akceptowalne przez menedżerów, w którym to przypadku taksonomiczne rozdzielanie mogłoby ograniczyć możliwości ratowania małych populacji wsobnych. Sugerujemy, że podczas gdy bardziej rygorystyczne podejście do wyznaczania jednostek wewnątrzgatunkowych, takich jak MUs, może pomóc złagodzić ten problem, oczywiste jest również, że genomiczne szacowanie szybkości migracji i rozbieżności adaptacyjnych może zapewnić znacznie bardziej solidne wskazówki nie tylko w definiowaniu granic gatunków i zmienności wewnątrzgatunkowych, ale także w projektowaniu strategii ratowania genetycznego.
Filogenomika i Ochrona
genomika w połączeniu z metodami filogenetycznymi może łatwo rozwiązać relacje i ewolucyjne odległości między taksonami, a także rozgraniczać gatunki. Najlepiej jest to osiągnąć przy użyciu metod drzewa gatunków, a nie konkatenacji, ponieważ te pierwsze uwzględniają heterogeniczność drzewa genowego i unikają nadmiernej inflacji długości końcówek z powodu polimorfizmu przodków(Edwards et al., 2016; Ogilvie et al., 2016). Podczas gdy masowe liczby loci często nie są niezbędne, w tym więcej loci często poprawia rozdzielczość zarówno relacji i długości gałęzi (np. Blom et al., 2016). Znaczenie dla ochrony przyrody jest dwojakie. Po pierwsze, oceny filogenetyczne umożliwiają rozważenie odrębności gatunków lub Esusów, a nie tylko tego, czy są one opisane. Tak więc, program krawędzi (Isaac et al., 2007) opowiada się za ważeniem gatunków do ochrony w oparciu o to, jak wiele unikalnej historii ewolucyjnej reprezentują, oprócz zwykłych kryteriów IUCN. Po drugie, same długości gałęzi mogą reprezentować wartości bioróżnorodności, niezależnie od taksonomii (Faith, 1992), co wykazało wartość w priorytetyzacji obszarów dla ochrony (Laity et al., 2015; Rosauer et al., 2016, 2018). W tym sensie przyjęcie metryk filogenetycznych przy użyciu środków takich jak różnorodność filogenetyczna i endemizm filogenetyczny umożliwia podkreślenie obszarów o wysokiej różnorodności ewolucyjnej, które mogą być ukierunkowane na ochronę i mieć wysoki priorytet w każdym procesie planowania dla lepszej ochrony różnorodności biologicznej. Takie podejście może zwiększyć ochronę różnorodności genetycznej i procesów ewolucyjnych dla różnych grup organizmów w różnych skalach geograficznych(Laity et al., 2015) podejmowanie decyzji o obszarach do priorytetyzacji bardziej odporne na kaprysy jak Genomic dowody przekłada się na rozpoznawanie gatunków lub wewnątrz konkretnych jednostek.
świat rzeczywisty—definicje legislacyjne
polityka i ramy prawne dotyczące ochrony różnorodności biologicznej obejmują konwencje i strategie międzynarodowe, strategie i prawodawstwo krajowe oraz Ustawodawstwo i strategie państwowe lub regionalne w poszczególnych krajach. Główne międzynarodowe instrumenty ochrony biologicznej obejmują Konwencję o różnorodności biologicznej wdrażaną poprzez Konwencję stron (COP), Konwencję Światowego Dziedzictwa, globalną strategię różnorodności biologicznej, proces Montrealski w zakresie ochrony lasów, Konwencję Ramsarską w sprawie Mokradeł, Konwencję o gatunkach migrujących, Konwencję o międzynarodowym handlu gatunkami zagrożonymi (CITES), Międzynarodową Unię Ochrony Przyrody (IUCN) w sprawie Czerwonej Listy gatunków zagrożonych oraz ramową Konwencję Narodów Zjednoczonych w sprawie zmian klimatu. Wszystkie te karty uosabiają zasadę zachowania gatunków i różnorodności genetycznej jako podstawowy cel zachowania bioróżnorodności, przy czym gatunki stanowią szeroko rozumianą jednostkę ochrony. Definicje gatunków na mocy ustawodawstwa i dyrektyw międzynarodowych są jednak bardzo zróżnicowane. Obejmują one ujęcie taksonomiczne gatunków i taksonów wewnątrzgatunkowych, ale mogą również obejmować koncepcje genetyczne / ewolucyjne, które często są objęte prawną definicją „gatunku” (Tabela 1).

Tabela 1. Światowe Ustawodawstwo i dyrektywy, które definiują” gatunki ” do notowania i ochrony.
biorąc pod uwagę szeroki zakres poziomów, które mogą być chronione w podczerwieni (Tabela 1), Ustawodawstwo i związane z nim procesy listowania w krajach powinny ułatwiać zachowanie różnorodności genetycznej. Na przykład definicja „gatunku”, jeśli weźmiemy pod uwagę prawodawstwo na całym świecie (Tabela 1), może obejmować: gatunki, podgatunki, odmiany i geograficznie i / lub genetycznie odrębne populacje, podczas gdy w ustawodawstwie południowoafrykańskim może to również obejmować kultywary, rasy geograficzne, szczepy i mieszańce. Tak więc, zgodnie z większością przepisów na całym świecie „gatunek” obejmuje podmioty, które odzwierciedlają różne poziomy specyficzne dla danego gatunku, które są częścią szerszego rozmieszczenia gatunków i odzwierciedlają w różnym stopniu kontinuum populacji-gatunku. Jest to równoznaczne z nieuniknioną trudnością, z jaką spotykają się taksonomowie, biolodzy ewolucyjni i genetycy zajmujący się ochroną przyrody w znalezieniu bardziej spójnego i jednolitego sposobu określania jednostek w tym kontinuum.
niektórzy twierdzą, że obawy dotyczące implikacji politycznych gatunków i niepewności w zakresie podczerwieni są niewłaściwie umiejscowione (Hey et al., 2003), ale nadal jesteśmy zdania, że chociaż prawodawstwo może zapewnić mechanizm ochrony różnorodności genetycznej na różnych poziomach, to na biologach spoczywa obowiązek zapewnienia większej jasności i spójności w zakresie tego, w jaki sposób poziomy te powinny być rozdzielane od gatunków do poszczególnych populacji. Obecnie menedżerowie i ustawodawcy pozostawiają oszałamiający zestaw pojęć gatunkowych i jednostek INFRA-specyficznych dostarczanych przez naukowców przy wyznaczaniu jednostek w kontinuum populacji-gatunków. Na przykład wszystkie jurysdykcje uznają gatunki uznane przez taksonomów za kluczowe jednostki ochrony (Tabela 1), jednak wśród taksonomów nadal toczy się szeroka debata na temat koncepcji gatunków, a sposób, w jaki gatunki są diagnozowane w praktyce, różni się w obrębie i w poszczególnych grupach. Doprowadziło to do niedawnego wezwania do bardziej regulowanego podejścia do taksonomii (Garnett and Christidis, 2017), na które inni przeciwstawili się, że regulacja taksonomii nie jest odpowiedzią (Thomson et al., 2018). Chociaż zgadzamy się z niektórymi punktami Thomson et al. (2018) sugerujemy, że raczej naiwne jest proponowanie, że sama legislacja musi ulec zmianie, aby wyraźnie odnieść się do konkretnego pojęcia taksonu sugerowanego przez nazwę. Te i poprzednie takie debaty podkreślają potrzebę, aby biolodzy ewolucyjni i taksonomiści przejęli przewodnictwo w zapewnianiu jasności (przejrzystości) w sposobie, w jaki przekładają dowody genomiczne i inne na rozgraniczenie gatunków lub jednostek wewnątrzgatunkowych, oraz w jaki sposób te taksony odnoszą się do odpowiednich przepisów dotyczących ochrony i ochrony.
tłumaczenie na zarządzanie
jak omówiono powyżej, wszystkie przepisy i dyrektywy uznają poziom gatunków za podstawową jednostkę ochrony, ale także przewidują uznanie różnych jednostek specyficznych dla infra. Wiele z nich ma również domniemany wymóg formalnego nazewnictwa i opisu taksonomicznego gatunków, gdy rozważa się priorytet ochrony i formalną ochronę. Na przykład, zgodnie z wytycznymi IUCN Red Listing Guidelines nie zaleca się umieszczania w wykazie nienazwanych gatunków i istnieje wymóg, aby nowy opis gatunku został opublikowany w ciągu czterech lat od umieszczenia gatunku na Czerwonej Liście IUCN lub został usunięty. Podkreśla to znaczenie nazewnictwa taksonomicznego i opisu jednostek, zarówno na poziomie gatunków, jak i na poziomie INFRA-specyficznym, ograniczone informacje taksonomiczne dla wielu grup roślin i zwierząt (Mace, 2004; Oliver and Lee, 2010; Wilson, 2017) oraz potrzebę przyspieszenia wysiłków taksonomicznych, szczególnie na obszarach bogatych w bioróżnorodność (Riedel et al., 2013; Wege et al., 2015; Wilson, 2017).
taksonomicznie nazwane gatunki i wewnątrzspecyficzne kategorie taksonomiczne, takie jak podgatunki i odmiany, na ogół stanowią podstawowe jednostki do formalnej listy, priorytetyzacji ochrony i ochrony, ale w niektórych jurysdykcjach inne kategorie wewnątrzspecyficznych jednostek ochrony są formalnie uznawane. Są one na ogół identyfikowane na podstawie połączonych danych genomicznych i fenotypowych. Na przykład USA Endangered Species Act rozpoznaje odrębne segmenty populacji (DPSs), które są szeroko oparte na ESUs (Waples, 1991), podczas gdy kanadyjski Species at Risk Act rozpoznaje DUs. Operacyjna interpretacja „odrębnych populacji”zgodnie z kanadyjską Ustawą o gatunkach zagrożonych, dus ma na celu identyfikację dyskretnych zestawów populacji, które stanowią ważne elementy ewolucyjnego Dziedzictwa gatunków i które prawdopodobnie nie zostaną zastąpione przez naturalną dyspersję (Mee et al., 2015).
zarówno DUs, jak i DPSs pokrywają się koncepcyjnie z ESUs (Waples et al., 2013) i były najszerzej stosowane do ryb północnoamerykańskich, w tym łososiowatych z północno-zachodnich Stanów Zjednoczonych (Waples, 1995) i kanadyjskich koregonidów (Mee et al., 2015) Co ciekawe DPSs w USA stosuje się tylko do kręgowców, podczas gdy w Kanadzie DUs może być stosowany do każdego organizmu, który może być wymieniony na podstawie jego ustawy(patrz Waples et al., 2013), z Dusem obecnie notowanym na stawonogach, mięczakach i roślinach. W Australii, podobnie jak w Kanadzie, odrębne populacje mogą być wymienione dla każdego organizmu uznanego w ustawie o ochronie środowiska i różnorodności biologicznej z 1999 r. (ustawa EPBC), chociaż nie są one specjalnie nazwaną kategorią, taką jak DUSs i DPSs. Jednak do tej pory zostały one uznane tylko dla kręgowców. W USA jest 127 kręgowców DPSS wymienionych do ochrony (https://ecos.fws.gov/ecp/); w Kanadzie 70 DUs zostały wymienione obejmujące wszystkie organizmy (http://www.registrelep-sararegistry.gc.ca/species/schedules_e.cfm?id=1); a w Australii populacje 7 gatunków kręgowców obejmujących Ssaki, ryby i ptaki zostały uznane za posiadające szczególny status ochronny na mocy ustawy EPBC (http://www.environment.gov.au/cgi-bin/sprat/public/publicthreatenedlist.pl). Podczas gdy oceny tych populacji w USA i Kanadzie na ogół używają informacji genetycznej (Waples et al., 2013), w Australii są one zwykle oparte na identyfikacji geograficznej i jurysdykcyjnej, chociaż w niektórych z tych przypadków zostały poparte analizą różnicowania genetycznego ssaków (Firestone et al., 1999; Armstrong, 2006) i fish (Colgan and Paxton, 1997; Ahonen et al., 2009).
lista jednostek ochrony roślin i bezkręgowców na podczerwień w Kanadzie (DUs), ale nie w USA czy Australii, podkreśla inną kwestię nie związaną specjalnie z wytyczaniem granic jednostek ochrony, ale szerzej z istotnymi niespójnościami w priorytetyzacji ochrony różnorodności biologicznej; kultowe kręgowce, takie jak ptaki, ssaki i ryby, są przedmiotem zainteresowania w wielu jurysdykcjach, podczas gdy inne organizmy nie są. Ta preferencyjna lista populacji niektórych organizmów odzwierciedla szereg kwestii, które są mniej związane z nauką o wytyczaniu jednostek ochrony, a bardziej związane z listą populacji opartą na świadomości społecznej, celowości politycznej i charyzmatycznej naturze organizmu, który ma zostać wymieniony (patrz Waples et al., 2013).
podczas gdy w Australii niektóre populacje znanych ssaków, ptaków i ryb są chronione na mocy ustawy EPBC Act, istnieje obecnie znaczny zbiór informacji opartych na badaniach molekularnych gadów, bezkręgowców i roślin, w których populacje można rozpoznać jako ESUs lub MUs, a wiele z nich ma znaczenie ochronne ze względu na ich wąski zasięg geograficzny, niewielką wielkość populacji, tempo spadku i podatność na procesy zagrażające. Na przykład uderzające przykłady wysoce ustrukturyzowanych populacji i głęboko rozbieżnych linii w obrębie gatunków, które z łatwością spełniałyby wymagania ESU, można znaleźć w szeregu kręgowców o niskim rozproszeniu z tropików (Rysunek 2) i gdzie indziej. Podobnie u Australijskich bezkręgowców, takich jak amfipody i stonogi, znaczna struktura genetyczna i wzorce filogeograficzne wspierają wytyczanie odrębnych klastrów populacji jako ESUs, które są geograficznie zróżnicowane, o wąskich zakresach geograficznych, ograniczone do konkretnych siedlisk i reprezentowane tylko przez kilka populacji (Finston et al., 2007; Murphy et al., 2013; Nistelberger et al., 2014a). Często te endemiczne gatunki są taksonami zagrożonymi ochroną (Harvey et al., 2011) i współwystępują w endemitach (np. Oliver et al., 2017), które same w sobie są wysokimi priorytetami w zakresie ochrony wielu taksonów (Rosauer et al., 2018), m.in. w regionie Kimberley i Stirling Range w południowo-zachodniej Australii Zachodniej.
w Australijskich roślinach istnieje również znaczna liczba badań genetycznych i filogeograficznych populacji identyfikujących różne grupy populacji w zakresie gatunków z różnych rodzajów, odzwierciedlających nieujawnione i historycznie izolowane systemy populacji, złożoność geologiczną i edaficzną oraz zajmujące kontrastujące siedliska pod względem roślinności i klimatu (patrz Broadhurst et al., 2017). Genetycznie odrębne populacje są szczególnie widoczne u gatunków z południowo-zachodniej Australii Zachodniej (Coates, 2000; Byrne et al., 2014) z szeregiem badań podkreślających znaczącą strukturę genetyczną i wysoce rozbieżne linie w zakresie rodzajów roślin i rodzin, w tym: Acacia (Mimosaceae; Coates, 1988), Atriplex (Amaranthaceae; Shepherd et al., 2015), Banksia (Proteaceae; Coates et al., 2015), Lambertia (Proteaceae; Byrne et al., 2002), Rodzina hakea (Proteaceae; Sampson et al., 2014), Eremophila (Scrophulariaceae; Llorens et al., 2015), Pultanaea (Fabaceae; Millar and Byrne, 2013) i Calothamnus (Myrtaceae; Nistelberger et al., 2014b). W wielu przypadkach populacje te są uznawane w planowaniu ochrony przez państwowe agencje rządowe i brane pod uwagę w ocenach oddziaływania na środowisko (patrz Shepherd et al., 2015), mimo że nie są one objęte ochroną na mocy przepisów. Przyznajemy jednak, że formalne uznanie tych jednostek poprawiłoby ich zachowanie w dłuższej perspektywie.
te Australijskie przykłady podkreślają rosnącą potrzebę rozpoznania tej warstwy różnorodności genetycznej poniżej poziomu gatunku. Wyzwaniem jest zapewnienie ochrony wysoce ustrukturyzowanych populacji i głęboko rozbieżnych linii, które stanowią zagrożenie dla ochrony, w obliczu konkurujących ze sobą wymagań dotyczących ochrony. Poza kultowymi gatunkami wyzwanie to jest widoczne również na całym świecie, z wyjątkiem Kanady. Brak ochrony wyraźnie wytyczonych jednostek ochronnych prawdopodobnie zwiększy ryzyko tajemniczego wymarcia i utraty znacznej różnorodności genetycznej, która w wielu przypadkach może obejmować linie i historycznie izolowane populacje, które przetrwały w obrębie gatunku przez miliony lat (Moritz, 2002).
wnioski i zalecenia
wykaz gatunków i różnych kategorii w ramach przepisów dotyczących gatunków zagrożonych lub organizacji międzynarodowych, takich jak IUCN, wskazuje na zaangażowanie w ochronę różnorodności genetycznej w całym kontinuum populacji gatunku. Jednak obecne podejścia do ochrony różnorodności biologicznej w dużej mierze koncentrują się na końcu gatunku tego kontinuum, z mniejszą uwagą na różnorodność genetyczną i zmienność w podczerwieni. To bardziej ograniczone skupienie się na zmienności genetycznej na poziomie wewnątrzgatunkowym sugeruje, że podczas gdy menedżerowie ochrony coraz częściej uznają wartość wyraźnego uwzględnienia informacji genetycznej w działaniach zarządzania i priorytetyzacji, często brakuje im przydatnych informacji od naukowców zajmujących się bioróżnorodnością (Waples et al., 2008; Laikre et al., 2010; Cook i Sgrò, 2017; Taylor et al., 2017). Ta przepaść między nauką a praktyką ochrony przyrody utrudnia bardziej skuteczne globalne podejście do ochrony i zarządzania różnorodnością genetyczną.
sugerujemy, że brak jasności ze strony naukowców zajmujących się ochroną różnorodności biologicznej w stosunku do jednostek ochrony jest w dużej mierze oparty na niespójności na dwóch poziomach. Pierwsza dotyczy koncepcji wielu gatunków i różnych metod diagnostycznych stosowanych do wyodrębnienia gatunków, co powoduje niespójność między grupami taksonomicznymi. Drugi odnosi się do braku spójności i zgody taksonomów, biologów ewolucyjnych i biologów ochrony, w jaki sposób traktować taksony podczerwone i inne jednostki, takie jak ESUs, DUs i MUs.
chociaż uważamy, że prawodawstwo jest często wystarczająco elastyczne pod względem sposobu definiowania taksonów (lub „gatunków”), uważamy, że istnieje potrzeba systematycznego uwzględniania kontinuum gatunków populacji i wyraźniejszego definiowania jednostek ochrony, z większym uznaniem dla pojęć takich jak ESU i ich powiązania z odpowiednimi przepisami. Podkreślamy, że kwestia ta bez wątpienia stanie się bardziej krytyczna, biorąc pod uwagę moc genomiki w wykrywaniu różnicowania w coraz drobniejszych skalach i jej coraz większe wykorzystanie do wyznaczania gatunków i jednostek podczerwonych.
na podstawie poprzedniej analizy proponujemy następujące zalecenia:
(i) Dane genomowe powinny być interpretowane zachowawczo przy wyznaczaniu gatunków, przy użyciu integracyjnego podejścia taksonomicznego, które zazwyczaj obejmuje dowody wykraczające poza same dane genetyczne.
(ii) w przypadku gdy genetycy identyfikują wiele linii lub odrębne populacje w obrębie gatunku, a te nie uzasadniają podniesienia do poziomu gatunku i nie są uznawane taksonomicznie, że istnieje silniejszy impuls i bardziej jednolite podejście do identyfikacji wyraźnie uzasadnionych wewnątrzspecyficznych jednostek do praktycznej oceny stanu ochrony lub oceny wpływu, oraz że są one wyraźnie uznawane za jednostki do ochrony w kontekście prawodawstwa właściwego dla tej jurysdykcji.
(iii) IUCN, poprzez swoją grupę specjalistów ds. genetyki konserwatorskiej i podgrupy regionalne, rozważa opracowanie wytycznych dotyczących rozpoznawania i znakowania jednostek wewnątrzgatunkowych, które są zgodne z najlepszą praktyką naukową.
(iv) bardziej niuansowy widok wyłaniający się z ewolucyjnej genomiki gatunków i kontinuum między populacjami a gatunkami powinien być skuteczniej przekazywany przez naukowców kierownikom ochrony i decydentom politycznym.
szerzej echo zalecenia wykonane niedawno w wielu recenzjach (Frankham, 2010; Santamaría and Méndez, 2012; Hoban et al., 2013; Moritz and Potter, 2013; Laity et al., 2015; Pierson et al., 2016; Cook i Sgrò, 2017; Taylor et al., 2017), które podkreślają potrzebę poprawy komunikacji naukowców i podnoszenia świadomości z decydentami politycznymi, agencjami rządowymi, organizacjami pozarządowymi i praktykami, że wyniki z systematyki molekularnej, taksonomii molekularnej, filogeografii i badań genetycznych populacji mogą dostarczyć krytycznych informacji na temat zachowania różnorodności genetycznej i zarządzania procesami ewolucyjnymi.
wkład autora
DC, MB i CM przyczyniły się do przygotowania i rewizji manuskryptu.
Oświadczenie o konflikcie interesów
autorzy oświadczają, że badanie zostało przeprowadzone przy braku jakichkolwiek relacji handlowych lub finansowych, które mogłyby być interpretowane jako potencjalny konflikt interesów.
podziękowania
Ten projekt był wspierany przez Australian Research Council (Arc Linkage Grant No. LP120200063), Australian National University i Western Australian Department of Biodiversity, Conservation, and Attractions.
Afonso Silva, A. C., Bragg, J. G., Potter, S., Fernandes, C., Coelho, M. M., and Moritz, C. (2017a). Tropical specialist versus climate generalist: diversification and demographic history of sister species of Carlia skinks from northwestern Australia. Mol. Ecol. 26:4045–4058. doi: 10.1111 / mec.14185
CrossRef Pełny tekst | Google Scholar
Afonso Silva, A. C., Santos, N., Ogilvie, H. A., and Moritz, C. (2017b). Walidacja i opis dwóch nowych północno-zachodnich Australijskich skórek tęczowych z wielogatunkowymi metodami koalescencji i morfologią. PeerJ 5: e3724 doi: 10.7717 / peerj.3724
PubMed Abstrakt | CrossRef Pełny tekst | Google Scholar
Ahonen, H., Harcourt, R. G., and Stow, A. J. (2009). DNA jądrowe i mitochondrialne ujawniają izolację populacji rekina szarego (Carcharias taurus). Mol. Ecol. 18, 4409–4421. doi: 10.1111 / j. 1365-294X. 2009. 04377.x
PubMed Abstract / CrossRef Full Text / Google Scholar
Phylogeographic structure in Rhinonicteris aurantia( Chiroptera: Hipposideridae): implications for conservation. Acta Chiropterologica 8, 63-81. doi: 10.3161/1733-5329(2006)82.0.CO
CrossRef Full Text / Google Scholar
, and Kunte, K. (2017). Adaptive genetic exchange: a tangled history of domieszka and evolutionary innovation. Trendy Ecol. Evol. 32, 601–611. doi: 10.1016 / j.drzewo.2017.05.007
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Avise, J. C. (2004). Markery molekularne, Historia naturalna i ewolucja. 2.Edn. Sunderland, MA; Oxford: Oxford University Press.
Google Scholar
Avise, J. C., Arnold, J., Ball, R. M., Bermingham, E., Lamb, T., Neigel, J. E., et al. (1987). Filogeografia wewnątrzgatunkowa: mitochondrialny most DNA między genetyką populacji a systematyką. Ann. Ks. Ekol. Sys. 18, 489–522. doi: 10.1146/annurev.es.18.110187.002421
CrossRef Pełny tekst/Google Scholar
Bickford, D., Lohman, D. J., Sodhi, N. S, Ng, P. K., Meier, R., Winker, K., et al. (2007). Zagadkowe gatunki jako okno na różnorodność i ochronę. Trendy Ecol. Evol. 22, 148–155. doi: 10.1016 / j.drzewo.2006.11.004
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Blom, M. P. K., Bragg, J. G., Potter, S., and Moritz, C. (2016). Accounting for uncertainty in gene tree estimation: summary-coalescent species tree in a challenging radiation of Australian lizards. Syst. Biol. 66, 352–366. doi: 10.1093/sysbio/syw089
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Broadhurst, L., Breed, M., Lowe, A., Bragg, J., Catullo, R., Coates, D. J., et al. (2017). Różnorodność genetyczna i struktura flory australijskiej. Nurków. Distrib. 23, 41–52. doi: 10.1111 / ddi.12505
CrossRef Pełny tekst / Google Scholar
, Krauss, S. L., Sniderman, J. M. K., et al. (2014). „A diverse flora-species and genetic relationships,” in Plant Life on the Sandplains in Southwest Australia, a Global Biodiversity Hotspot, editors H. Lambers (Crawley, WA: University of Western Australia Publishing), 81-99.
PubMed Abstract
Byrne, M., Macdonald, B., and Coates, D. J. (2002). Wzorce filogeograficzne i duża różnorodność cpDNA w obrębie Acacia acuminata (Leguminosae: Mimosoideae). kompleks w Australii Zachodniej. J. Evol. Biol. 15, 576–587. doi: 10.1046 / j.1420-9101.2002.00429.x
CrossRef Pełny tekst | Google Scholar
Carstens, B. C., Pelletier, T. A., Reid, N. M., and Satler, J. D. (2013). Jak zawieść w wytyczaniu gatunków. Mol. Ecol. 22, 4369–4383. doi: 10.1111 / mec.12413
PubMed Abstract / CrossRef Full Text / Google Scholar
Różnorodność genetyczna i struktura genów populacji w rzadkiej trawie Chittering wattle, Acacia anomala Court. Aust. J. Bot. 36:273–286. doi: 10.1071 / BT9880273
CrossRef Full Text / Google Scholar
Definiowanie jednostek ochrony w bogatej i fragmentarycznej florze: implikacje dla zarządzania zasobami genetycznymi i procesami ewolucyjnymi w roślinach południowo-zachodniej Australii. Aust. J. Bot. 48, 329–339. doi: 10.1071/BT99018
CrossRef Pełny tekst/Google Scholar
Coates, D. J., McArthur, S. L., and Byrne, M. (2015). Znaczna utrata różnorodności genetycznej w wyniku wymierania populacji wywołanego przez patogen w rzadkiej endemicznej Banksia brownii (Proteaceae). Biol. Conserv. 192, 353–360. doi: 10.1016 / j.biocon.2015.10.013
CrossRef Full Text | Google Scholar
Colgan, D. J., and Paxton, J. R. (1997). Genetyka biochemiczna i uznanie zachodniego stada klejnotów pospolitych, Rexea solandri (Scombroidea: Gempylidae)., w Australii. Mar. Freshw. Res. 48, 103-118. doi: 10.1071 / MF96048
CrossRef Full Text/Google Scholar
Konwencja o różnorodności biologicznej (2007). Dostępne na stronie internetowej: https://www.cbd.int/doc/meetings/cop-bureau/cop-bur-2007/cop-bur-2007-10-14-en.pdf
Cook, C. N., and Sgrò, C. M. (2017). Dostosowanie nauki i polityki w celu osiągnięcia ewolucyjnie oświeconej ochrony. Conserv. Biol. 31, 501–512. doi: 10.1111 / cobi12863
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Cook, L. G., Edwards, R. D., Crisp, M. D., and Hardy, N. B. (2010). Czy morfologia musi być zawsze wymagana dla nowych opisów gatunków? Odwróć. Sys. 24, 322–332. doi: 10.1071 / IS10011
CrossRef Full Text / Google Scholar
Cracraft, J. (1983). Pojęcia gatunkowe i analiza specjacji. Curr. Ornithol. 1, 159–187. doi: 10.1007/978-1-4615-6781-3_6
CrossRef Full Text / Google Scholar
Crandall, K. A., Bininda-Emonds, O. R., Mace, G. M., and Wayne, R. K. (2000). Rozważanie procesów ewolucyjnych w biologii ochrony. Trendy Ecol. Evol. 15, 290–295. doi: 10.1016/S0169-5347(00)01876-0
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Cutter, A. D., and Payseur, B. A. (2013). Genomic signatures of selection at linked sites: unifying the disparity among species. Nat. Wielebny Genet. 14, 262–274. doi: 10.1038/nrg3425
PubMed Abstract | CrossRef Full Text/Google Scholar
Dayrat, B. (2005). W kierunku taksonomii integracyjnej. Biol. J. Linn. Soc. 85, 407–415. doi: 10.1111 / j.1095-8312.2005.00503.x5
CrossRef Full Text / Google Scholar
de Queiroz, K. (1998). „The general lineage concept of species, species criteria, and the process of speciation,” in Forms: Species and Speciation, D. J. Howard, r S. H. Berlocher (New York, NY: Oxford University Press), 57-75.
Google Scholar
de Queiroz, K. (2007). Pojęcia gatunkowe i delimitacja gatunków. Syst. Biol. 56, 879–886. doi: 10.1080/10635150701701083
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Degnan, J. H., and Rosenberg, N. A. (2009). Niezgodność drzewa genowego, wnioskowanie filogenetyczne i wielogatunkowe koalescencje. Trendy Ecol. Evol. 24, 332–340. doi: 10.1016 / j.drzewo.2009.01.009
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Devey, D. S., Bateman, R. M., Fay, M. F., And Hawkins, J. A. (2008). Przyjaciele czy krewni? Filogenetyka i rozgraniczenie gatunków w kontrowersyjnym europejskim rodzaju orchidei Ophrys. Ann. Bot. 101, 385–402. DOI: 10.1093/AOB | mcm299
PubMed Abstract/CrossRef Full Text/Google Scholar
Dizon, A. E., Lockyer, C, Perrin, W. F., Demaster, D. P., and Sisson, J. (1992). Przemyślenie koncepcji stada-podejście filogeograficzne. Conserv. Biol. 6, 24–36. doi: 10.1046 / j.1523-1739.1992.610024.x
CrossRef Pełny tekst | Google Scholar
Doughty, P., Bourke, G., Tedeschi, L., Pratt, R., Oliver, P., Palmer, R., et al. (2018). Gatunek zaliczany do rodziny Gehyra nana (Squamata: Gekkonidae). complex: cryptic and divergent morphological evolution in the Australian Monsoonal Tropics, with the description of four new species. Zootaxa, 4403: 201-244. doi: 10.11646 / 4403.2.1
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Dynesius, M., and Jansson, R. (2014). Trwałość linii wewnątrzgatunkowych: zaniedbana kontrola szybkości specjacji. Evolution 68, 923-934. doi: 10.1111 / evo.12316
PubMed Abstrakt / CrossRef Pełny tekst / Google Scholar
, Potter, S., Schmitt, C. J., Bragg, J. G., and Moritz, C. (2016). Reticulation, divergence, and the phylogeography-phylogenetics continuumc. Proc. Natl. Acad. Sci. U. S. A. 113, 8025-8032. doi: 10.1073 / pnas.1601066113
CrossRef Full Text / Google Scholar
Ocena zachowania i różnorodność filogenetyczna. Biol. Minusy. 61, 1–10. doi: 10.1016/0006-3207(92)91201-3
CrossRef Pełny tekst / Google Scholar
Finston, T. L., Johnson, M. S., Humphreys, W. F., Eberhard, S. M., and Halse, S. A. (2007). Tajemnicza specjacja w dwóch rozległych podziemnych rodzajach amfipodów odzwierciedla historyczne wzorce drenażu w starożytnym krajobrazie. Mol. Ecol. 16, 355–365. doi: 10.1111 / j. 1365-294X. 2006. 03123.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Firestone, K. B., Elphinstone, M. S., Sherwin, W. B., and Houlden, B. A. (1999). Phylogeographic population structure of tiger quolls Dasyurus maculatus( Dasyuridae: Marsupalia), an endangered carnivorous torbacz. Mol. Ecol. 8, 1613–1625. doi: 10.1046 / j. 1365-294x. 1999. 00745.x
CrossRef Pełny tekst / Google Scholar
Fišer, C., Robinson, C. T., and Malard, F. (2018). Zagadkowe gatunki jako okno do zmiany paradygmatu koncepcji gatunku. Mol. Ecol. 27, 613–635. doi: 10.1111 / mec.14486
PubMed Abstract / CrossRef Full Text / Google Scholar
Wyzwania i możliwości genetycznego podejścia do ochrony biologicznej. Biol. Conserv. 143,1919–1927. doi: 10.1016 / j.biocon.2010.05.011
CrossRef Pełny tekst / Google Scholar
, Eldridge, M. D., Fenster, C. B., Lacy, R. C., et al. (2012). Implikacje różnych koncepcji gatunków dla zachowania bioróżnorodności. Biol. Conserv. 153, 25–31. doi: 10.1016 / j.biocon.2012.04.034
CrossRef Full Text / Google Scholar
Adaptive evolutionary conservation: towards a unified concept for defining conservation units. Mol. Ecol. 10, 2741–2752. doi: 10.1046 / j. 1365-294X. 2001.t01-1-01411.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Funk, W. C., Mckay, J. K., Hohenlohe, P. A., and Allendorf, F. W. (2012). Wykorzystanie genomiki do wyznaczania jednostek ochrony. Trendy Ecol. Evol. 27, 489–496. doi: 10.1016 / j.drzewo.2012.05.012
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Garnett, S. T., and Christidis, L. (2017). Anarchia taksonomiczna utrudnia ochronę. Nature 546, 25-27. DOI: 10.1038/546025a
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Gippoliti, S., Cotterill, F. P. D., Zinner, D., and Groves, C. P. (2018). Wpływ bezwładności taksonomicznej na zachowanie różnorodności afrykańskich zwierząt kopytnych: przegląd. Biol. Rev. 93, 115-130. doi: 10.1111 / brv.12335
PubMed Abstract / CrossRef Full Text / Google Scholar
Grant, V. (1981). Specjacja Roślin. New York, NY: Columbia University Press.
PubMed Abstract | Google Scholar
Hamilton, C. W., and Reichard, S. H. (1992). Obecna praktyka stosowania podgatunków, odmian i formy w klasyfikacji roślin dziko rosnących. Takson 41, 485-498. doi: 10.2307/1222819
CrossRef Pełny tekst/Google Scholar
Harvey, M. S., Rix, M. G., Framenau, V. W., Hamilton, Z. R., Johnson, M. S., Teale, R. J., et al. (2011). Ochrona niewinnych: badanie taksonów endemicznych krótkiego zasięgu zwiększa wyniki ochrony. Odwróć. Syst. 25, 1–10. doi: 10.1071/IS11011
CrossRef Full Text/Google Scholar
Hey, J., Waples, R. S., Arnold, M. L., Butlin, R. K., and Harrison, R. G. (2003). Zrozumienie i konfrontacja z niepewnością gatunków w biologii i ochronie. Trendy Ecol. Evol. 18, 597–603. doi: 10.1016 / j.drzewo.2003.08.014
CrossRef Pełny tekst | Google Scholar
Hoban, S. M., Hauffe, H. C., Perez-Espona, S., Arntzen, J. W., Bertorelle, G., Bryja, J., et al. (2013). Wprowadzanie różnorodności genetycznej na czele polityki ochrony i zarządzania. Conserv. Genet. Resour. 5, 593–598. doi: 10.1007 / s12686-013-9859-y
CrossRef Pełny tekst | Google Scholar
Isaac, N. J., Mallet, J., and Mace, G. M. (2004). Inflacja taksonomiczna: jej wpływ na makroekologię i konserwację. Trendy Ecol. Evol. 19, 464–469. doi: 10.1016 / j.drzewo.2004.06.004
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Isaac, N. J., Turvey, S. T., Collen, B., Waterman, C., and Baillie, J. E. (2007). Mammals on the EDGE: conservation priorities based on threat and phylogeny. PLoS ONE 2: e296. doi: 10.1371 / dziennik.pone.0000296
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Ukierunkowane wychwytywanie w genomice ewolucyjnej i ekologicznej. Mol. Ecol. 25, 185–202. doi: 10.1111 / mec.13304
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Jorger, K. M., and Schrödl, M. (2013). Jak opisać tajemniczy gatunek? Praktyczne wyzwania taksonomii molekularnej. Przód. Zool. 10:59. doi: 10.1186/1742-9994-10-59
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Laikre, L., Allendorf, F., Aroner, L. C., Baker, S., Gregovich, D. P., Hansen, M. M., et al. (2010). Zaniedbanie różnorodności genetycznej przy wdrażaniu Konwencji o różnorodności biologicznej. Conserv. Biol. 24, 86–88. doi: 10.1111 / j. 1523-1739. 2009. 01425.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Laikre, L., Palm, S., and Ryman, N. (2005). Genetic population structure of fishes: implications for coastal zone management. Ambio 34.111–119. doi: 10.1579/0044-7447-34.2.111
PubMed Abstrakt | CrossRef Pełny tekst | Google Scholar
Laity, T., Laffan, S. W., González-Orozco, C. E., Faith, D. P., Rosauer, D. F., Byrne, M., et al. (2015). Phylodiversity to inform conservation policy: an Australian example. Sci. Total Environ. 534, 131–143. doi: 10.1016 / j.scitotenv.2015.04.113
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Leache, A. D., and oaks, J. R. (2017). Użyteczność polimorfizmu pojedynczego nukleotydu (SNP). data in phylogenetics. Ann. Ks. Ekol. Syst. 48, 69–84. doi: 10.1146/annurev-ecolsys-110316-022645
CrossRef Full Text | Google Scholar
Llorens, T. M., Macdonald, B., McArthur, S., Coates, D. J., and Byrne, M. (2015). Disjunct, highly divergent genetic lineages within two rare Eremophila (Scrophulariaceae: Myoporeae). species in a biodiversity hotspot: implications for taxonomy and conservation. Biol. J. Linn. Soc. 177, 96–111. doi: 10.1111/boj.12228
CrossRef Full Text | Google Scholar
Love Stowell, S. M., Pinzone, C. A., and Martin, A. P. (2017). Pokonywanie barier w aktywnej interwencji na rzecz różnorodności genetycznej. Bioróżnorodności. Conserv. 26, 1753–1765. doi: 10.1007 / s10531-017-1330-z
CrossRef Full Text / Google Scholar
Rola taksonomii w ochronie gatunków. Philos. Trans. R. Soc. B: Biol. Sci. 359, 711–719. doi: 10.1098 / rstb.2003.1454
PubMed Abstract / CrossRef Full Text / Google Scholar
Mallet, J. (2007). Specjacja hybrydowa. Nature 446, 279-283. doi: 10.1038 / nature05706
PubMed Abstract | CrossRef Full Text | Google Scholar
Mayr, E. (1942). Systematyka i Pochodzenie gatunków. New York, NY: Columbia University Press.
Google Scholar
Mee, J. A., Bernatchez, L., Reist, J. D., Rogers, S. M., and Taylor, E. B. (2015). Identyfikacja jednostek przeznaczonych do wyznaczania priorytetów ochrony wewnątrzgatunkowej: podejście hierarchiczne stosowane do kompleksu gatunków siejących jezioro (Coregonus spp.). Evol. Appl. 8, 423–441. 10.1111 / Ewa12247
PubMed Abstract | CrossRef Full Text | Google Scholar
Millar, M. A., and Byrne, M. (2013). Cryptic divergent lineages of Pultenaea pauciflora M.B. Scott (Fabaceae: Mirbelieae) exhibit different evolutionary history. Biol. J. Linn. Soc. 108, 871–881. doi: 10.1111/bij.12002
CrossRef Full Text | Google Scholar
Moritz, C. (1994). Defining evolutionarily significant units for conservation. Trends Ecol. Evol. 9, 373–375. doi: 10.1016/0169-5347(94)90057-4
CrossRef Full Text | PubMed Abstract
Moritz, C. (1999). Conservation units and translokations: strategies for conserving evolutionary processes. Hereditas 130, 217-228. doi: 10.1111 / j.1601-5223.1999.00217.x
CrossRef Full Text / Google Scholar
Moritz, C. (2002). Strategie ochrony różnorodności biologicznej i procesów ewolucyjnych, które ją podtrzymują. Syst. Biol. 51, 238–254. doi: 10.1080/10635150252899752
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Moritz, C., and Potter, S. (2013). Znaczenie perspektywy ewolucyjnej w planowaniu polityki ochrony przyrody. Mol. Ecol. 22, 5969–5971. doi: 10.1111 / mec.12565
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Moritz, C., Pratt, R. C., Bank, S., Bourke, G., Bragg, J. G., Doughty, P., et al. (2018). Tajemnicza różnorodność linii, rozbieżność wielkości ciała i sympatria w kompleksie gatunkowym Australijskich jaszczurek (Gehyra). Ewolucja 27, 54-66. doi: 10.1111 / evo.13380
CrossRef Pełny tekst | Google Scholar
Morrison, W. R., Lohr, J. L., Duchen, P., Wilches, R., Trujillo, D., Mair, M., et al. (2009). Wpływ zmian taksonomicznych na ochronę: Czy zabija, czy może uratować, czy jest to po prostu nieistotne? Biol. Minusy. 142, 3201–3206. doi: 10.1016 / j.biocon.2009.07.019
CrossRef Pełny tekst | Google Scholar
Murphy, N. P., Adams, M., Guzik, M. T., and Austin, A. D. (2013). Niezwykły mikro-endemizm u Australijskich amfipodów desert spring. Mol. Filogenet. Evol. 66, 645–653. doi: 10.1016 / j. ympev.2012.10.013
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Murphy, N. P., King, R. A., and Delean, S. (2015). Gatunki, ESUs czy populacje? Wytyczanie i opisywanie morfologicznie zagadkowej różnorodności Australijskich amfipodów desert spring. Odwróć. Syst. 29, 457–467. doi: 10.1071/IS14036
CrossRef Pełny tekst/Google Scholar
Nater, A., Mattle-Greminger, M. P., Nurcahyo, A., Nowak, M. G., de Manual, M., Desai, T., et al. (2017). Morfometryczne, behawioralne i genomowe dowody nowego gatunku orangutana. Curr. Cell Biol. 27, 3487–3498. doi: 10.1016 / j.cub.2017.09.047
CrossRef Pełny tekst | Google Scholar
Nicotra, A. B., Chong, C., Bragg, J. G., ONG, C. R., Aitken, N. C., Chuah, A., et al. (2016). Population and phylogenomic decomposition through genotyping-by-sequencing in Australian Pelargonium. Mol. Ecol. 25, 2000–2014. doi: 10.1111 / mec.13584
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Nistelberger, H., Byrne, M., Coates, D., and Roberts, J. D. (2014a). Silna struktura filogeograficzna w stonogu wskazuje na wikariancję plejstocenu między populacjami na prążkowanych formacjach żelaza w półpustynnej Australii. PLoS ONE 9: e0093038. doi: 10.1371 / dziennik.pone.0093038
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Nistelberger, H., Gibson, N., Macdonald, B., Tapper, S.-L., and Byrne, M. (2014b). Phylogeographic evidence for two mesic refugia in a biodiversity hotspot. 113, 454-463. doi: 10.1038 / hdy.2014.46
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Ogilvie, H. A., Heled, J., Xie, D., and Drummond, A. J. (2016). Wydajność obliczeniowa i dokładność statystyczna bestii oraz porównania z innymi metodami. Syst. Biol. 65, 381–396. doi: 10.1093/sysbio/syv118
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Oliver, P. M., Keogh, J. S., and Moritx, C. (2015). New approaches to cataloguing and understanding evolutionary diversity: a perspective from Australian herpetology. Aust. J. Zool. 62, 417–430. Doi: 10.1071/ZO14091
CrossRef Pełny tekst/Google Scholar
Oliver, P. M., Laver, R. J., Demello Martins, F., Pratt, R. C., Hunjan, S., and Moritz, C. (2017). Nowy punkt zwrotny kręgowców endemitów i ewolucyjne refugium w tropikalnej Australii. Nurek. Distrib. 23, 53–66. doi: 10.1111 / ddi.12506
CrossRef Pełny tekst | Google Scholar
Oliver, P. M., and Lee, M. S. Y. (2010). Kodeksy botaniczne i zoologiczne utrudniają badania nad bioróżnorodnością, zniechęcając do publikacji nienazwanych nowych gatunków. Takson 59, 1201-1205. doi: 10.2307/20773990
CrossRef Pełny tekst/Google Scholar
Padial, J., Miralles, A., De la Riva, I., and Vences, M. (2010). Integracyjna przyszłość taksonomii. Przód. Zool. 7:16. doi: 10.1186/1742-9994-7-16
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Palsbøll, P. J., Bérubé, M., and Allendorf, F. W. (2007). Identyfikacja jednostek zarządzających z wykorzystaniem populacyjnych danych genetycznych. Trendy Ecol. Evol. 22, 11–16. doi: 10.1016 / j.drzewo.2006.09.003
PubMed Abstract | CrossRef Full Text | Google Scholar
Patton, J. L., and Smith, M. F. (1994). Parafilię, polifilię i charakter granic gatunkowych u susłów kieszonkowych (Rodzaj Thomomys). Syst. Biol. 43, 11–26. doi: 10.1093 / sysbio / 43.1.11
CrossRef Pełny tekst | Google Scholar
Pierson, J. C., Coates, D. J., Oostermeijer, J. G. B., Beissinger, S. R., Bragg, J. G., Sunnucks, P., et al. (2016). Czynniki genetyczne w planach odnowy gatunków zagrożonych na trzech kontynentach. Przód. Ecol. Environ. 14, 433–440. doi: 10.1002 / fee.1323
CrossRef Pełny tekst | Google Scholar
Potter, S., Bragg, J. G., Peter, B. M., Bi, K., and Moritz, C. (2016). Phylogenomics at the tips: inferring lineages and their demographic history in a tropical lizard, Carlia amax. Mol. Ecol. 25, 1367–1380. doi: 10.1111 / mec.13546
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Potter, S., Xue, A. T., Bragg, J. G., Rosauer, D. F., Roycroft, E. J., and Moritz, C. (2018). Plejstoceńskie zmiany klimatyczne prowadzą do zróżnicowania tropikalnej sawanny. Mol. Ecol. 27, 520–532. doi: 10.1111 / mec.14441/pełny
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Rabosky, D. L., Hutchinson, M. N., Donnellan, S. C., Talaba, A. L., and Lovette, I. J. (2014). Filogenetyczny demontaż granic gatunków w szeroko rozpowszechnionej grupie skór Australijskich( Scincidae: Ctenotus). Mol. Filogenet. Evol. 77, 71–82. doi: 10.1016 / j. ympev.2014.03.026
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Ralls, K., Ballou, J. D., Dudash, M. R., Eldridge, M. D. B., Fenster, C. B., Lacy, R. C., et al. (2018). Apel o zmianę paradygmatu w zarządzaniu genetycznym rozproszonymi populacjami. Conserv. Lett. 11, 1–6. 10.1111 / conl.124126
CrossRef Pełny tekst | Google Scholar
Rannala, B. (2015). Sztuka i nauka o delimitacji gatunków. Curr. Zool. 61, 846–853. doi: 10.1093/czoolo/61.5.846
CrossRef Pełny tekst/Google Scholar
Riedel, A., Sagata, K., Suhardjono, Y. R., Tänzler, R., and Balke, M. (2013). Integracyjna Taksonomia na szybkiej ścieżce-w kierunku bardziej zrównoważonego rozwoju w badaniach nad różnorodnością biologiczną. Przód. Zool. 10:15. Doi: 10.1186/1742-9994-10-15
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Rieseberg, L. H., and Willis, J. H. (2007). Specjacja roślin. Nauka 317, 910-914. 10.1126 / nauka1137729
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Rosauer, D. F., Blom, M. P. K., Bourke, G., Catalano, S., Donnellan, S., Mulder, E., et al. (2016). Phylogeography, hotspots and conservation priorities: an example from the Top End of Australia. Biol. Minusy. 204, 83–93. doi: 10.1016 / j.biocon.2016.002
CrossRef Pełny tekst | Google Scholar
Rosauer, D. F., Byrne, M., Blom, M. P. K., Coates, D. J., Donnellan, S., Doughty, P., et al. (2018). Real-world conservation planning for evolutionary diversity in the Kimberley, Australia, sidesteps uncertain taxonomy Conserv. Lett. 11: e12438 10.1111 / conl.12438
CrossRef Pełny tekst/Google Scholar
Rosenblum, E. B., Sarver, B. A. J., Brown, J. W., Des Roches, S., Hardwick, K. M., Hether, T. D., et al. (2012). Złotowłosa spotyka się z Santa Rosalią: efemeryczny model specjacji wyjaśnia wzorce dywersyfikacji w różnych skalach czasowych. Evol. Biol. 39, 255–261. doi: 10.1007 / s11692-012-9171-x
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Roux, C., Fraïsse, C., Romiguier, J., Anciaux, Y., Galtier, N., and Bierne, N. (2016). Rzuca światło na szarą strefę specjacji wzdłuż kontinuum dywergencji genomowej. PLoS Biol. 14: e2000234 doi: 10.1371 / dziennik.pbio.2000234
PubMed Abstract / CrossRef Full Text / Google Scholar
Ochrona i systematyka gatunków-dylemat podgatunków. Trendy Ecol. Evol. 1, 9–10. doi: 10.1016/0169-5347(86)90059-5
CrossRef Pełny tekst | Google Scholar
Sampson, J. F., Byrne, M., Yates, C. J., Gibson, N., Thavornkanlapachai, R., Stankowski, S., et al. (2014). Współczesna imigracja genów za pośrednictwem pyłków odzwierciedla historyczną izolację rzadkiego krzewu w rozdrobnionym krajobrazie. 112, 172-181. doi: 10.1038 / hdy.2013.89
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Santamaría, L., and Méndez, P. F. (2012). Ewolucja polityki w zakresie różnorodności biologicznej: obecne luki i przyszłe potrzeby. Evol. Appl. 5, 202–218. doi: 10.1111 / j. 1752-4571. 2011. 00229.x
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Schlick-Steiner, B. C., Steiner, F. M., Seifert, B., Stauffer, C., Christian, E., and Crozier, R. H. (2010). Integrative taxonomy: a multisource approach to exploring biodiversity. Annu. Wielebny Entomol. 55, 421–438. doi: 10.1146/annurev-ento-112408-085432
PubMed Abstract / CrossRef Full Text / Google Scholar
Seehausen, O, Takimoto, G., Roy, D., and Jokela, J. (2008). Odwrócenie specjacji i dynamika bioróżnorodności z hybrydyzacją w zmieniających się środowiskach. Mol Ecol. 17, 30–44. doi: 10.1111 / j. 1365-294X. 2007. 03529.x
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Shepherd, K. A., Thiele, K. R., Sampson, J., Coates, D. J., and Byrne, M. (2015). Rzadki, nowy gatunek Atriplex (Chenopodiaceae) obejmujący dwie genetycznie odrębne, ale morfologicznie tajemnicze populacje w suchej Australii Zachodniej: implikacje dla taksonomii i ochrony. Aust. Syst. Bot. 28, 234–245. doi: 10.1071/Sb15029
CrossRef Pełny tekst / Google Scholar
Singhal, S., and bi, K. (2017). Historia oczyszcza bałagan: wpływ czasu na dywergencję i introgresję w strefie tropikalnej. Evolution 71, 1888-1899. doi: 10.1111 / evo.13278
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Singhal, S., Hoskin, C., Couper, P., Potter, S., and Moritz, C. (2018). Framework for resolving Cryptic species: a case study from the lizards of the Australian wet tropics. Syst. Biol. . doi: 10.1093/sysbio/syy
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Singhal, S., and Moritz, C. (2013). Izolacja rozrodcza między liniami filogeograficznymi Skala się z rozbieżnością. Proc. Roy. Soc B 280: 20132246. 10.1098/2013.2246026
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Steane, D. A., Potts, B. M., McLean, E., Collins, L., Prober, S. M., Stock, W. D., et al. (2015). Skany genomu ujawniają tajemniczą strukturę populacji w suchym eukaliptusie. Drzewo Genet. Genomy 11: 33. doi: 10.1007 / s11295-015-0864-z
CrossRef Pełny tekst | Google Scholar
Struck, T. H., Feder, J. L., Bendiksby, M., Birkeland, S., Cerca, J., Gusarov, V. I., et al. (2017). Odnajdywanie procesów ewolucyjnych ukrytych w tajemniczych gatunkach. Trendy Ecol. Evol. 33, 153–163. doi: 10.1016 / j.drzewo.2017.11.007
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Sukumaran, J., and Knowles, L. L. (2017). Wielogatunkowa koalescencja wyznacza strukturę, a nie Gatunek. Proc. Nat. Aca. Sci. U. S. A. 114, 1607-1612. doi: 10.1073 / pnas.1607921114
PubMed Streszczenie | CrossRef Pełny tekst | Google Scholar
Swarts, N. D., Clements, M. A., Bower, C. C., and Miller, J. T. (2014). Określenie jednostek ochrony w kompleksie morfologicznie podobnych, seksualnie zwodniczych, wysoce zagrożonych storczyków. Biol. Minusy. 174, 55–64. doi: 10.1016 / j.biocon.2014.03.017
CrossRef Pełny tekst | Google Scholar
Tautz, D., Arctander, P., Minelli, A., Thomas, R. H., and Vogler, A. P. (2003). Prośba o taksonomię DNA. Trendy Ecol. Evol. 18, 70–74. doi: 10.1016/S0169-5347(02)00041-1
CrossRef Pełny tekst / Google Scholar
Taylor, H. R., Dussex, N., And van Heezik, Y. (2017). Niwelowanie luki w genetyce ochrony poprzez identyfikowanie barier we wdrażaniu przez praktyków ochrony. Glob. Ecol. Conserv. 10, 231–242. doi: 10.1016 / j.gecco.2017.04.001
CrossRef Pełny tekst | Google Scholar
Thomson, S. A., Pyle, R. L., Ahyong, S. T., Alonso-Zarazaga, M., Ammirati, J., Araya, J. F., et al. (2018). Taksonomia oparta na nauce jest niezbędna do globalnej ochrony. PLoS Biol. 16: e2005075 doi: 10.1371 / dziennik.pbio.2005075
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Vieites, D. R., Wollenberg, K. C., Andreonec, F., Köhler, J., Glaw, F., and Vences, M. (2009). Ogromne niedoszacowanie bioróżnorodności Madagaskaru świadczy o integracyjnym inwentarzu płazów. Proc. Natl. Acad. Sci. U. S. A. 106, 8267-8272. doi: 10.1073 / pnas.0810821106
CrossRef Pełny tekst
Vogler, A. P., and de Salle, R. (1994). Diagnozowanie jednostek Zarządzania Ochroną. Minusy. Biol. 8, 354–363. doi: 10.1046 / j. 1523-1739. 1994. 08020354.x
CrossRef Full Text | Google Scholar
Waples, R. S. (1991). Łosoś pacyficzny, Oncorhynchus spp., oraz definicji „gatunków” w ustawie o gatunkach zagrożonych. Mar. Ryby. Rev. 53, 11-22.
Google Scholar
Waples, R. S. (1995). „Evolutionarily significant units and the conservation of biological diversity under the Endangered Species Act,” in Evolution and The Aquatic Ecosystem: Defining Unique Units in Population Conservation, eds J. L. Nielsen (Bethesda, MD:American Fisheries Society), 8-27.
Google Scholar
Waples, R. S., Gustafson, R. G., Weitkamp, L. A., Myers, J. M., Jjonson, O. W., Busby, P. J., et al. (2001). Charakteryzujące różnorodność łososia z Północno-Zachodniego Pacyfiku. J. Fish Biol. 59, 1–41. doi: 10.1111 / j.1095-8649.2001.tb01376x
CrossRef Pełny tekst / Google Scholar
Waples, R. S., Nammack, M., Cochrane, J. F., And Hutchings, J. A. (2013). Opowieść o dwóch aktach: zagrożonych gatunkach w Kanadzie i Stanach Zjednoczonych. BioScience 63, 723-734. doi: 10.1093 / bioscience / 63.9.723
CrossRef Full Text | Google Scholar
Waples, R. S., Punt, A. E., and Cope, J. M. (2008). Integracja danych genetycznych w zarządzaniu zasobami morskimi: jak możemy to zrobić lepiej? Rybna 9, 423-449 doi: 10.1111 / j.1467-2979.2008.00303.x
CrossRef Pełny tekst | Google Scholar
Wege, J. A., Thiele, K. R., Shepherd, K. A., Butcher, R., Macfarlane, T. D., and Coates, D. J. (2015). Strategic taxonomy in a biodiverse landscape: a novel approach to maximising conservation effects for rare and poorly known flora. Bioróżnorodności. Conserv. 24, 17–32. doi: 10.1007 / s10531-014-0785-4
CrossRef Pełny tekst | Google Scholar
Whitlock, R. (2014). Relacje między adaptacyjną i neutralną różnorodnością genetyczną a strukturą i funkcjonowaniem ekologicznym: metaanaliza. J. Ecol. 102, 857–872. doi: 10.1111/1365-2745. 12240
PubMed Abstract | CrossRef Full Text/Google Scholar
Koncepcja gatunków ewolucyjnych została ponownie rozpatrzona. Syst. Biol. 27, 17–26. doi: 10.2307/2412809
CrossRef Full Text/Google Scholar
Badania nad bioróżnorodnością wymagają większej liczby działań w terenie. Nat. Ecol. Evol. 1, 1590–1591. doi: 10.1038 / s41559-017-0360-y
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Yang, Z. H., and Rannala, B. (2014). Nieograniczona delimitacja gatunków przy użyciu danych sekwencji DNA z wielu loci. Mol. Biol. Evol. 31, 3125–3135. doi: 10.1093/molbev/msu279
PubMed Streszczenie | CrossRef Pełny tekst/Google Scholar
Yeates, D. K., Seago, A., Nelson, L., Cameron, S. L., Joseph, L., and Trueman, W. H. (2011). Taksonomia integracyjna, czy Taksonomia iteracyjna? Sys. Ent. 36, 209–217. doi: 10.1111/j.1365-3113.2010.00558.x
CrossRef Full Text | Google Scholar