Nei primi anni 1960, biologi cominciato a indagare in che modo le proteine in diverse specie evolvono a livello di sequenza (Zuckerkandl 1962, Margoliash 1963, Doolittle & Blombäck 1964). Le proteine analizzate includevano emoglobina (Zuckerkandl & Pauling 1962), citocromo C (Margoliash 1963) e fibrinopeptidi (Doolittle & Blombäck 1964). Queste prime indagini hanno portato a una scoperta notevole: è emerso che il numero di differenze tra sequenze proteiche di specie diverse era approssimativamente proporzionale al tempo trascorso dalla divergenza delle specie (Figura 1).
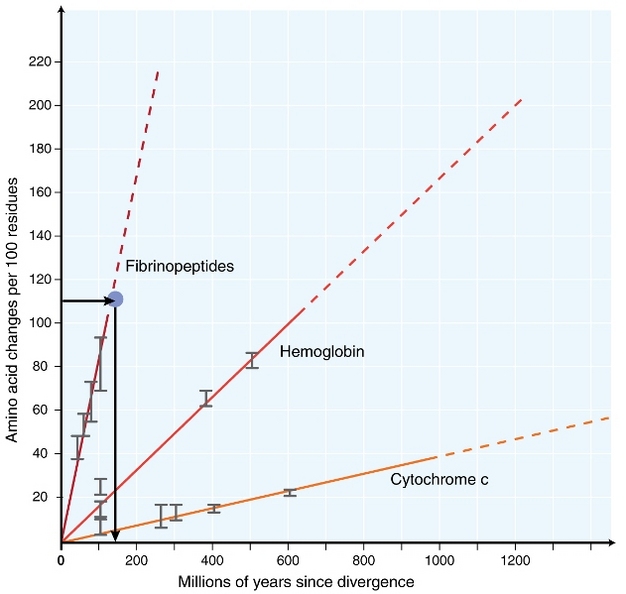
Zuckerkandl & Pauling (1965) paragonò l’accumulo costante di sostituzioni di aminoacidi nel tempo a regolari “zecche” di orologi, e affermò che “potrebbe esistere un orologio evolutivo molecolare”. Pertanto, il termine “orologio molecolare” è stato inizialmente coniato per descrivere i cambiamenti negli amminoacidi che si verificano in proporzione al tempo dalla divergenza delle specie.
Fin dal suo primo utilizzo, il termine ‘orologio molecolare’ è stato utilizzato in molti contesti diversi. Al giorno d’oggi, è spesso usato per riferirsi semplicemente al numero di cambiamenti, o “sostituzioni”, accumulati nella sequenza di DNA o proteine in un dato lignaggio. Il numero di sostituzioni per un’unità di tempo definita può essere descritto come il “tasso” dell’orologio molecolare, che in questo contesto è equivalente al “tasso evolutivo”. Si noti che la connotazione iniziale è ancora valida in alcuni casi. Ad esempio, quando le persone affermano ‘abbiamo assunto un orologio molecolare’ (in particolare nelle analisi filogenetiche), significa che il numero di sostituzioni è stato assunto per verificarsi costantemente nel tempo.
Il concetto di un orologio molecolare costante ha implicazioni straordinarie per i biologi evoluzionisti. Se un orologio molecolare costante come inizialmente proposto esistesse veramente, dedurre la tempistica degli eventi evolutivi diventerebbe un problema piuttosto semplice (Figura 1). Tuttavia, è diventato abbondantemente chiaro che le sostituzioni non si verificano costantemente nel tempo in diversi lignaggi (Kumar 2005). Tuttavia, il concetto di orologio molecolare è stato estremamente influente nel campo dell’evoluzione molecolare. Una delle idee più importanti ispirate al concetto di orologio molecolare è la teoria neutrale dell’evoluzione molecolare.
L’orologio molecolare e la neutralità
Quando l’idea di un orologio molecolare costante emerse per la prima volta, si pensava che la forza evolutiva predominante alla base delle sostituzioni di amminoacidi o nucleotidi fosse la selezione naturale. Seguendo questa linea di pensiero, un orologio molecolare costante indicherebbe che le sostituzioni adattive in diverse specie si verificano costantemente nel tempo. Tuttavia, è difficile spiegare come le sostituzioni adattive si verificherebbero in modo simile a un orologio. Teoricamente, i destini delle mutazioni adattive sono determinati da diversi parametri evolutivi, come la forza del vantaggio selettivo di quella mutazione, la dimensione della popolazione effettiva e i tassi di mutazione adattiva (Kimura 1983). È probabile che questi parametri differiscano tra le specie e anche all’interno di una specie, a seconda delle mutazioni specifiche e delle loro interazioni con gli ambienti.
Invece, Kimura (1968, 1969) ha proposto che la maggior parte dei cambiamenti a livello molecolare hanno poche conseguenze funzionali, o sono ‘neutrali’. Se una mutazione non ha conseguenze di idoneità, il suo destino nella popolazione è determinato completamente per caso casuale. Ciò significa che non possiamo prevedere se una specifica mutazione neutra alla fine sarà fissata nella popolazione. Tuttavia, la velocità con cui si verificano sostituzioni neutre nella popolazione può essere prevista perché dipende da un singolo parametro, vale a dire il tasso di mutazione (Kimura 1968).
Immaginiamo una popolazione con N numero di individui aploidi. Se le mutazioni neutre si verificano al tasso u per individuo per generazione, il numero totale di mutazioni in una generazione sarà N volte u. Poiché tutte queste nuove mutazioni sono neutre, i loro destini sono completamente determinati per caso. In altre parole, tutte le mutazioni hanno uguali possibilità di raggiungere la fissazione (che porta a una “sostituzione”). La probabilità che ogni nuova mutazione neutra raggiunga la fissazione, dato che si è verificata una sostituzione, è semplicemente 1 / N. Il tasso di sostituzioni è calcolato come il numero di nuove mutazioni in ogni generazione (Nu) moltiplicato per la probabilità che ogni nuova mutazione raggiunga la fissazione (1/N), che è uguale a u. In altre parole, per le mutazioni neutre, il tasso di sostituzione è uguale al tasso di mutazione!
Pertanto, se la maggior parte delle mutazioni sono neutre (come proposto nella teoria neutra) e se i tassi di mutazione sono costanti nel tempo, le sostituzioni dovrebbero verificarsi costantemente nel tempo. Dovremmo quindi osservare tassi regolari di sostituzioni simili a un orologio a livello molecolare. Kimura (1969) considerava quindi l’osservazione dell’orologio molecolare relativamente costante nelle sequenze proteiche come un forte supporto per la teoria neutra dell’evoluzione molecolare.
Testare come funziona l’orologio molecolare neutro
Secondo la teoria neutrale, la questione se i tassi di sostituzione siano costanti nel tempo o meno è equivalente a se i tassi di mutazione neutri siano costanti nel tempo. Per questo motivo, molti studi successivi si sono concentrati sull’analisi dei dati da siti neutri per determinare se i tassi di mutazione neutri sono effettivamente costanti nel tempo. Esamineremo brevemente come questi studi sono implementati, prima di discutere i dibattiti teorici sugli orologi molecolari. Anche le analisi degli orologi molecolari proteici sono proseguite, ma i dibattiti che circondano la variazione degli orologi molecolari proteici sono molto diversi da quelli riguardanti gli orologi molecolari neutri e non saranno inclusi in questo articolo. I lettori interessati dovrebbero consultare Gillespie (1991), Kumar (2005), Kim e Yi (2008) e Bedford et al. (2008).
La maggior parte delle analisi empiriche degli orologi molecolari neutri si basano sul teorema che i tassi di mutazione neutri possono essere dedotti dai tassi di sostituzione neutri (Kimura 1968, 1969). In pratica, ogni studio definisce un certo tipo di siti nel genoma come siti neutri e confronta i tassi di sostituzione di tali siti tra lignaggi.
Quali siti nel genoma sono veramente neutrali non possono essere completamente determinati, ma gli scienziati hanno escogitato diversi proxy utili. Prima dell’era del sequenziamento del genoma, la maggior parte dei dati di sequenza disponibili erano quelli delle sequenze di DNA codificanti proteine. Gli studi spesso dividono le sequenze di DNA codificanti proteine in due tipi di siti (Wu & Li 1985). Il primo tipo di siti include quelli per i quali qualsiasi cambiamento porterebbe a sostituzioni di amminoacidi, o “siti non sinonimi”. Il secondo tipo di siti, “siti sinonimi”, include quelli che codificano posizioni “degenerate” nella tabella dei codoni, in cui un cambiamento non porta a una sostituzione di aminoacidi. Ad esempio, TCT e TCC codificano entrambi serine. Se la terza posizione di questi codoni cambia, produrranno ancora gli stessi amminoacidi. Tali sostituzioni sarebbero meno visibili alla selezione naturale. Di conseguenza, gli orologi molecolari nei siti sinonimi dovrebbero essere più vicini all’orologio molecolare neutro rispetto agli orologi non sinonimi.
Come tecniche di sequenziamento avanzate, alcuni studi hanno utilizzato introni come proxy per siti neutri, poiché non sono incorporati negli MRNA maturi, e quindi hanno maggiori probabilità di essere neutrali (Yi et al. 2002). Sequenze di elementi trasponibili inattivi che sono stati inseriti molto prima della divergenza delle specie sono stati spesso impiegati (questi sono spesso indicati come ‘ripetizioni ancestrali’, ad esempio, Thomas et al., 2003). Infine, alcuni studi hanno utilizzato sequenze di DNA non codificanti (tutte le sequenze dopo aver rimosso sequenze di DNA codificanti proteine) estratte da allineamenti di tutto il genoma per testare orologi molecolari neutri (Elango et al. 2006, Huttley et al. 2007).
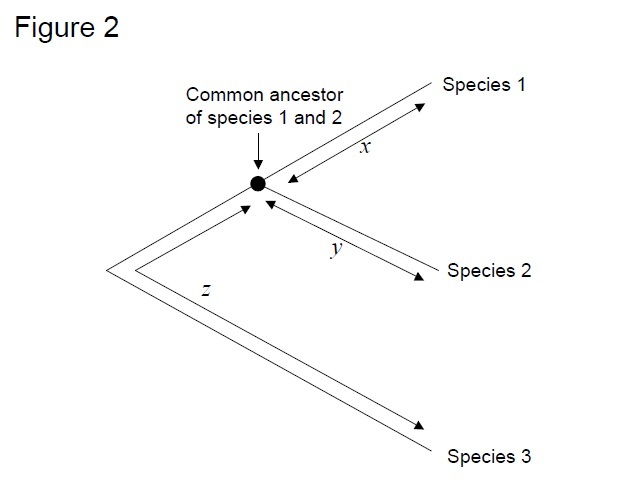
Il test più comunemente usato è il cosiddetto”test della velocità relativa”(Sarich& Wilson 1973). Inizialmente, i tassi di sostituzione per unità di tempo sono stati stimati dividendo il numero totale di differenze (sostituzioni) tra proteine di specie diverse per il tempo di divergenza, stimato dai reperti fossili (Figura 1). Tuttavia, i documenti fossili non sono disponibili per molti confronti e sono associati a grandi margini di errore. Il test della velocità relativa supera la necessità di documentazione fossile (Figura 2). Finché esiste una sequenza di outgroup alle due linee di interesse, possiamo determinare se i due rami seguono gli stessi o diversi orologi molecolari, senza la conoscenza del tempo assoluto di divergenza (Figura 2).
Quali sono i determinanti degli orologi molecolari neutri?
Quasi tutte le controversie al centro dei dibattiti sugli orologi molecolari neutri derivano dalla questione di quali siano le principali fonti di mutazioni. Questa domanda è direttamente rilevante per comprendere i modelli di mutazione, che sono la fonte ultima del cambiamento evolutivo e della malattia genetica. Inoltre, capire come i tassi di mutazione variano tra lignaggi e all’interno dei genomi è una questione fondamentale nella genomica comparativa, che mira a utilizzare i confronti di sequenze per identificare le regioni genomiche che sono funzionalmente importanti.
Quindi cosa determina i tassi di mutazione neutri? Uno dei più importanti contributori agli orologi molecolari neutri è la variazione specifica del lignaggio nei tempi di generazione. Fin dall’inizio, l’idea di un orologio molecolare neutro costante è stata percepita come in contrasto con i meccanismi molecolari della mutazione germinale. È stato a lungo considerato che la maggior parte delle mutazioni derivano da errori nella replicazione del DNA nelle linee germinali (Haldane 1947, Muller 1954). Poiché le mutazioni si verificano quando il DNA germinale viene replicato per la prossima generazione, dovrebbero accumularsi in proporzione al numero di generazioni, piuttosto che alla quantità assoluta di tempo. Pertanto, se confrontassimo il numero di sostituzioni che si sono accumulate in due lignaggi dalla loro divergenza, il lignaggio con un tempo di generazione più lungo, avendo subito meno eventi di replicazione del DNA, ospiterebbe meno sostituzioni rispetto al lignaggio con il tempo di generazione più breve. Di conseguenza, l’orologio molecolare dovrebbe funzionare più lentamente in specie con tempi di generazione più lunghi. Questa idea è indicata come “effetto generazione-tempo”.
In effetti, l’effetto generazione-tempo è stato osservato per la prima volta negli studi sui primati, anche prima del dibattito sull’orologio molecolare. Morris Goodman, che stava usando metodi immunologici per indagare sulla relazione tra le specie all’epoca, osservò che la velocità con cui alcune proteine divergono sembrava essere diminuita nelle scimmie, in particolare negli esseri umani, rispetto alle scimmie del Vecchio Mondo (Goodman 1961, 1962, 1963). Questo effetto è indicato come “rallentamento del tasso di ominoidi”. Poiché gli ominoidi hanno tempi di generazione più lunghi rispetto alle scimmie del Vecchio Mondo, questa osservazione può essere spiegata dall’effetto generazione-tempo.
Wu & Li (1985) furono i primi a testare l’ipotesi dell’effetto del tempo di generazione usando i dati della sequenza del DNA. Hanno usato i dati di 11 geni di primati e roditori. Poiché i primati hanno un tempo di generazione molto più lungo rispetto ai roditori, l’orologio molecolare dovrebbe essere più veloce nei roditori rispetto ai primati. In effetti, hanno scoperto che per i siti sinonimi, i roditori mostrano circa due volte il tasso di evoluzione molecolare rispetto ai primati (Wu & Li 1985). Per i siti nonsynonymous tuttavia, un tale effetto non è stato trovato. In altre parole, l’orologio molecolare neutro, ma non l’orologio molecolare degli aminoacidi, ticchetta più velocemente nel lignaggio dei roditori rispetto al lignaggio dei primati, che si adatta bene all’idea di un effetto generazione-tempo.
Studi successivi hanno fornito ulteriore supporto al rallentamento della velocità degli ominoidi (Li & Tanimura 1987, Bailey et al. 1991) e la differenza di tasso tra il roditore e le stirpi dei primati (Gu & Li 1992, Huttley et al. 2007). Inoltre, una differenza di velocità è stata osservata in scale filogenetiche ancora più piccole, specialmente nei primati: ad esempio, l’orologio molecolare umano funziona più lentamente dell’orologio molecolare degli scimpanzé (Elango et al. 2006); i tassi nelle scimmie del Nuovo Mondo sono più veloci dei tassi negli ominoidi e nelle scimmie del Vecchio Mondo (Steiper& Young 2006). I diversi tassi di orologi molecolari osservati in questi studi sono qualitativamente coerenti con l’effetto del tempo di generazione.
Tuttavia, le differenze effettive tra le linee non sono quantitativamente coerenti con la differenza nei tempi di generazione. Ad esempio, Kumar & Subramanian (2002) ha mostrato che anche se la differenza nei tempi di generazione tra primati e roditori è molto più grande di quella tra umani e scimmie del Vecchio Mondo, le differenze osservate negli orologi molecolari sono simili in questi due confronti. Vale la pena notare che Kumar& Subramanian (2002) ha utilizzato filtri statistici specifici per rimuovere dati che mostravano modelli di sostituzione “eterogenei”, che potrebbero aver causato un pregiudizio verso sequenze in lenta evoluzione (Yi et al. 2002). Tuttavia, la differenza negli orologi molecolari di primati e roditori appare molto meno di quanto originariamente proposto da Wu & Li (1985). Ad esempio, Huttley et al. (2007) ha analizzato gli allineamenti dell’intero genoma di diverse specie tra cui l’opossum e ha dimostrato che la differenza di tasso tra i lignaggi euteri e il lignaggio opossum (~30%) è molto maggiore della differenza di tasso tra lignaggi umani e topi (~14%). Questi esempi dimostrano che il grado di differenze negli orologi molecolari varia significativamente tra i diversi studi a causa delle differenze nei set di dati e nei metodi statistici. Essi mostrano anche che le differenze di tasso tra lignaggi non possono essere completamente contabilizzate dalla differenza nei tempi di generazione da solo. Chiaramente, ci sono altri contributori agli orologi molecolari neutri.
In effetti, i tratti della storia della vita diversi dai tempi di generazione sembrano co-variare con gli orologi molecolari. Martin& Palumbi (1993) ha dimostrato che gli orologi molecolari del DNA corrono più velocemente in specie con piccole dimensioni corporee. Questa osservazione ha portato all’ipotesi che i tassi metabolici siano determinanti importanti degli orologi molecolari. Un alto tasso metabolico produce un gran numero di radicali mutageni dell’ossigeno, che aumenterebbero i tassi di mutazione (Rand 1994). Poiché i tassi metabolici e le dimensioni corporee generalmente co-variano con i tempi di generazione, è stato difficile distinguere quale di questi costituisce il principale determinante dei tassi di clock molecolari. Tsantes& Steiper (2009) ha proposto, sulla base dei dati dei primati, che l’età alla prima riproduzione, piuttosto che le dimensioni del corpo, è il principale determinante degli orologi molecolari. Poiché l’età alla prima riproduzione riflette l’effetto del tempo di generazione, questo studio supporta l’idea che l’effetto del tempo di generazione sia il principale determinante dell’orologio molecolare. Tuttavia, questo studio si basa ancora su un numero limitato di lignaggi (sono state utilizzate quattro coppie di specie). Pertanto, la distinzione tra dimensioni corporee, effetto del tempo di generazione e tassi metabolici rimane ancora una questione importante nella generalizzazione e nella comprensione degli orologi molecolari neutri.
Inoltre, l’importanza di fattori che non co-variano con i tempi di generazione, come la metilazione del DNA, è stata sempre più apprezzata. La metilazione del DNA è una modifica chimica del DNA genomico trovato in diversi taxa. Nei genomi animali, la metilazione del DNA si verifica quasi esclusivamente a citosine seguite da guanine (cosiddette “CpG”). Le citosine metilate, a loro volta, tendono a mutare rapidamente in timine a causa dell’instabilità chimica (Bird 1980). Infatti, nel genoma umano, le mutazioni causate dalla metilazione del DNA si verificano più di un ordine di grandezza più frequentemente di altre mutazioni (Nachman& Crowell 2000, Elango et al. 2008). Poiché le mutazioni causate dalla metilazione del DNA si verificano in gran parte indipendentemente dalla replicazione del DNA, tali mutazioni possono seguire diversi orologi molecolari rispetto ad altri. Nello specifico, invece della dipendenza dal tempo di generazione, le mutazioni causate dalla metilazione del DNA possono seguire un orologio molecolare dipendente dal tempo, che è simile a quanto inizialmente proposto da Zuckerkandl & Pauling (Kim et al. 2006)!
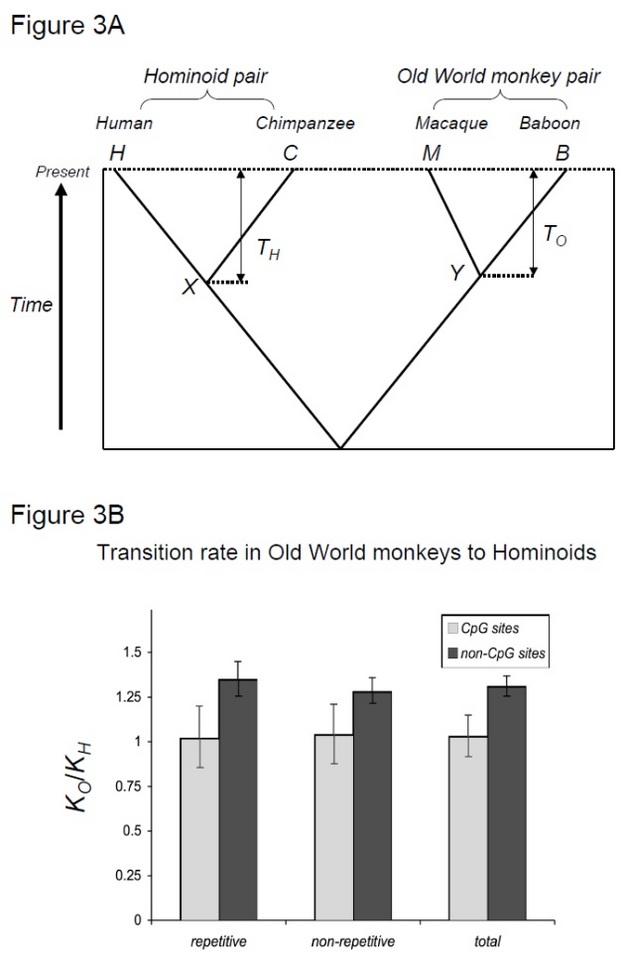
Per testare questa ipotesi, Kim et al. (2006) ha confrontato la divergenza uomo-scimpanzé con la divergenza macaco-babbuino, due coppie di specie che condividono tempi di divergenza simili ma con tempi di generazione diversi (Steiper et al. 2004). La coppia uomo-scimpanzé (la coppia ominoide) ha tempi di generazione molto più lunghi rispetto alla coppia macaco-babbuino (la coppia scimmia del Vecchio Mondo). Questo studio ha dimostrato che per i siti non CpG, la coppia di scimmie del Vecchio Mondo ha accumulato circa il 30% in più di sostituzioni, il che può essere spiegato dal suddetto effetto di rallentamento del tasso ominoide. Al contrario, gli orologi molecolari nei siti CpG hanno mostrato un numero simile di sostituzioni in coppie di scimmie ominoidi e del Vecchio Mondo (Figura 3). Pertanto, gli orologi molecolari dipendenti dal tempo e dipendenti dal tempo di generazione coesistono all’interno degli stessi genomi. L’ipotesi che un singolo orologio molecolare possa esistere per un dato lignaggio non è più valida, perché le forze mutazionali predominanti variano tra le regioni genomiche.
Conclusioni
Il concetto di un orologio molecolare costante è stato inizialmente proposto sulla base di una quantità limitata di dati di sequenza proteica. Anche se studi successivi hanno dimostrato che tale osservazione non è un modello generale negli amminoacidi, ha avuto un’influenza significativa sul campo dell’evoluzione molecolare, in particolare sullo sviluppo della teoria neutra dell’evoluzione molecolare per i dati della sequenza del DNA. Seguendo la teoria neutra, gli studi si sono concentrati sulla delucidazione dei modelli di variazione nei tassi di mutazione neutri. Durante gli ultimi decenni, abbiamo osservato che gli orologi molecolari funzionano a velocità diverse tra i lignaggi. Inoltre, il grado di variazione potrebbe variare a seconda dei diversi tipi di dati e dei metodi statistici specifici utilizzati. L’effetto del tempo di generazione continua a mantenersi a livello qualitativo, ma non è sufficiente a spiegare la variazione quantitativa dei tassi di mutazione neutri tra i lignaggi. I tratti della storia della vita e le mutazioni dipendenti dalla non replicazione, come quelle causate dalla metilazione del DNA, sono anche importanti contributori agli orologi molecolari genomici. Infatti, diversi tipi di orologi molecolari sono osservati anche all’interno di un genoma, perché gli input mutazionali predominanti variano tra le diverse regioni genomiche. Quindi, piuttosto che assumere un singolo orologio molecolare neutro per ogni genoma, gli studi futuri dovrebbero mirare a rivelare la variazione degli orologi molecolari neutri genomici, per conoscere i paesaggi mutazionali genomici. Tali informazioni non sono solo utili per comprendere la materia prima che regola l’evoluzione molecolare e la malattia genetica, ma costituiscono anche una componente critica che influenza le analisi genomiche comparative e funzionali per identificare le regioni genomiche funzionali.