- Introduzione
- Materiali e metodi
- Ceppi
- Estrazione del DNA
- stx1 e stx2 Amplificazioni PCR
- Sequenziamento del DNA
- Preparati per micropiastre Vero Cells
- Preparati batterici senza cellule
- Analisi citotossica e determinazione della LD50
- Test di blocco e neutralizzazione della tossina
- L’acquisizione di OMVs e l’estrazione del DNA da OMVs
- Risultati
- Amplificazioni PCR del gene stx1 e stx2
- Sequenziamento del DNA
- Analisi citotossica e determinazione LD50
- Neutralizzazione della tossina
- le amplificazioni PCR del gene stx1 da OMVs
- Discussione
- Contributi dell’autore
- Finanziamenti
- Dichiarazione sul conflitto di interessi
- Ringraziamenti
- Abbreviazioni
Introduzione
Per diversi anni, il significato di Aeromonas spp., come agente che causa la diarrea umana era controverso; diversi studi hanno dimostrato che il meccanismo patogeno di Aeromonas è multifattoriale perché sono coinvolti molti fattori di virulenza, inclusa la produzione di citotossine (Castro-Escarpulli et al., 2002, 2003). Queste tossine possono causare diarrea o colite emorragica e possono svolgere un ruolo importante nella sindrome emolitico-uremica (HUS) e nello sviluppo del TTP (Bogdanović et al., 1991; Fang et al., 1999; Monforte-Cirac et al., 2010).
Le citotossine implicate in queste malattie includono la tossina Shiga e la Stx strettamente correlata. Le varianti Stx sono espresse in specie batteriche Shigella, Enterobacter, Citrobacter, Acinetobacter, Campylobacter e Hamiltonella (Mauro e Koudelka, 2011). Alperi e Figueras (2010) hanno descritto la presenza di Stx1 e Stx2 in isolati clinici di Aeromonas spp., associato a gastroenterite, colite emorragica e HUS. I geni che codificano queste tossine si trovano in diversi batteriofagi lambdoidi che lisogenizzano questo ceppo. Inoltre, il genere Aeromonas ha un sistema a secrezione zero chiamato OMVs. Gli OMV potrebbero essere un mezzo attraverso il quale alcune proteine, RNA, componenti spaziali periplasmici e altri componenti associati alla virulenza, possono essere trasferiti orizzontalmente ad altri generi; pertanto, si ritiene che gli OMV giochino un ruolo importante nella patogenicità (Guerrero-Mandujano et al.,2015 bis, b).
Per questo motivo, lo scopo di questo studio era quello di valutare il danno causato dalla produzione di Stx da ceppi isolati da bambini di Città del Messico in colture cellulari Vero.
Materiali e metodi
Ceppi
Questo studio ha incluso 66 isolati clinici dall’INP, 54 ottenuti da infezioni intestinali e 12 da infezioni extra-intestinali. I ceppi sono stati isolati da campioni ottenuti per test di routine presso l’ospedale menzionato; pertanto, non è stato richiesto alcun consenso informato da parte dei genitori o dei tutori legali dei bambini. Tutti i ceppi sono stati geneticamente identificati da 16S rDNA-RFLP (Hernández-Cortez et al., 2011). Il ceppo tipizzato per Escherichia coli O157: H7 CECT 4076 è stato utilizzato come controllo positivo e il ceppo E. coli K12 (5512 ENCB) della collezione del Laboratorio di batteriologia medica (Escuela Nacional de Ciencias Biologicas, IPN) è stato utilizzato come controllo negativo per la produzione di tossine. I ceppi sono stati mantenuti per brevi periodi a temperatura ambiente su inclinazioni a base di agar del sangue; per una conservazione più lunga, sono stati congelati a -70°C in brodo di glicerolo-Todd-Hewitt al 20% (p/v) (Oxoid, Messico) o liofilizzati in 7.siero di glucosio di cavallo al 5%.
Estrazione del DNA
Tutte le colture sono state coltivate su agar di soia triptica a 30°C per 18 h. Il DNA genomico di ciascun ceppo è stato ottenuto attraverso InstaGene Matrix (BioRad®, Messico) secondo le istruzioni fornite dal produttore. La purezza e la quantità del DNA sono state determinate utilizzando lo spettrofotometro Ampli Quant AQ-07. Il DNA è stato conservato a -20°C fino all’uso.
stx1 e stx2 Amplificazioni PCR
La presenza di stx1 e stx2 dal DNA di gli om e il DNA genomico è stato rilevato da sola, le reazioni di PCR utilizzando primer STX1F/STX1R e STXF/STXR con 144 e 217 bp prodotto, rispettivamente, di questi primer sono stati progettati in base alla sequenza di subunità A. Il primer, la reazione, e condizioni di amplificazione sono stati elaborati come precedentemente descritto da Hernández-Cortez et al. (2013), con i controlli positivi (E. coli O157:H7) e negativi (E. coli K12).
Sequenziamento del DNA
I prodotti della reazione a catena della polimerasi sono stati purificati utilizzando un kit di estrazione rapida in gel PureLink (Invitrogen®, Messico) secondo le istruzioni del produttore. I prodotti sono stati sequenziati direttamente su un analizzatore genetico ABI-PRISM 310 (Applied Biosystems, Foster City, CA, USA) utilizzando i primer forward e reverse utilizzati per la PCR, secondo le istruzioni del produttore. Il sequenziamento è stato eseguito presso l’Instituto de Biología, UNAM (Messico). L’analisi della sequenza è stata eseguita con lo strumento di ricerca di allineamento locale di base (BLAST) fornito dal National Center for Biotechnology Information (NCBI).
Preparati per micropiastre Vero Cells
Questa procedura è stata eseguita in micropiastre a 96 pozzetti con monostrato di cellule Vero (ATCC CCL81) con confluenza dell ‘ 80%, aggiungendo un mezzo essenziale minimo (MEM; Invitro®, Messico) integrato con siero bovino fetale al 10% v/v (FBS; Invitro®, Messico).
La sospensione cellulare è stata omogeneizzata e regolata a 105-106 cellule / mL utilizzando una camera di Neubauer. Dopo la regolazione, la sospensione è stata depositata in un pozzetto da 200 µL. Le micropiastre sono state incubate a 37°C sotto il 5% di CO2 per 24 h (CO2 Incubator, VWR Scientific, USA) (Giono-Cerezo et al., 1994).
Preparati batterici senza cellule
Cinque colonie da ciascuna piastra di agar del sangue sono state inoculate in 3 mL di Craig medium (0,4% estratto di lievito, 3% casamino acidi, 0,05% K2HPO4). Questi sono stati incubati per 24 ore a 37°C e la densità ottica della coltura batterica utilizzata era 0,25 a 600 nm. I preparati senza cellule sono stati fatti centrifugando le colture a 14.000 g per 10 min a 4°C, seguita dalla filtrazione del surnatante attraverso un filtro a membrana (dimensione dei pori 0.45 pm, Sartorius Minisart NML). I surnatanti senza cellule sono stati conservati a -20°C. In questo modo sono stati ottenuti un totale di 66 preparati batterici senza cellule; il controllo positivo (E. coli O157:H7) e il controllo negativo (E. coli K12) sono stati ottenuti allo stesso modo (Giono-Cerezo et al., 1994).
Analisi citotossica e determinazione della LD50
Il filtrato privo di cellule (20 µL) è stato inoculato in pozzetti contenenti cellule e nel rispettivo terreno di crescita senza antibiotici. Le cellule inoculate sono state incubate per 96 ore a 37°C con il 5% di CO2 e osservate ogni 24 ore al microscopio invertito. Ci si aspettava che l’effetto citotossico apparisse come arrotondamento e restringimento delle cellule con granulazione spessa e, infine, distruzione progressiva e irreversibile del monostrato. Tutti i test sono stati eseguiti in duplice copia; controlli di fattibilità con MEM e Craig medium sono stati eseguiti anche (Giono-Cerezo et al., 1994).
La DL50 è stata determinata in ceppi di Aeromonas rilevati come positivi per causare danni citotossici nelle cellule Vero. Una micropiastra standard da 96 pozzetti con celle Vero è stata preparata come indicato in 2.6, ma il mezzo è stato modificato in 100 µL di MEM con 1% BFS. Questa preparazione è stata esposta a 100 µL di preparazione batterica senza cellule e sono state eseguite diluizioni seriali su tutta la fila di piastre. Dopo 24 ore di incubazione a 37°C con il 5% di CO2, la LD50 è stata determinata al microscopio invertito. LD50 è stato assegnato al pozzo in cui il 50% delle cellule Vero è stato danneggiato e il 50% non danneggiato (Marques et al., 1986).
Test di blocco e neutralizzazione della tossina
Il test di neutralizzazione della tossina è stato eseguito nei ceppi che hanno indotto danni citotossici nelle cellule Vero perché questo danno è indicativo di Stx. Per questo test, è stata preparata una micropiastra con cellule Vero cresciute fino a una confluenza del 90-100% con la LD50 di ciascuna preparazione batterica senza cellule. Per eseguire il test di neutralizzazione per mostrare il danno cellulare prodotto come conseguenza dell’azione Stx, sono stati utilizzati due anticorpi monoclonali (Universidad Nacional Autónoma de México) ottenuti da E. coli O157:H7.
Per ogni Ab è stata effettuata una quantificazione della proteina di Bradford secondo le istruzioni del produttore (Biorad), con un risultato di 78,2 e 74,8 µg/µL, rispettivamente, di anti-Stx1 e anti-Stx2 A. L’Ab concentrato è stato lavorato e una doppia diluizione dell’Ab. Quindi, 10 µL di Ab (anti-STX1 o anti-STX2) sono stati incubati con 190 µL del surnatante privo di cellule ad una concentrazione di LD50 per 1 h a 37°C. In seguito, 200 µL di quest’ultimo sono stati inoculati in cellule Vero al 90% della confluenza e incubati per 24 h a 37°C con il 5% di CO2 (Marques et al., 1986).
L’acquisizione di OMVs e l’estrazione del DNA da OMVs
Vescicole di membrana esterna sono state ottenute da Aeromonas hydrophila F-0050. Il protocollo è stato eseguito come precedentemente descritto da Guerrero-Mandujano et al. (2015a).
Il DNA di OMVs è stato ottenuto attraverso InstaGene Matrix (BioRad®, Messico) secondo le istruzioni fornite dal produttore. Quindi, il DNA degli OMV è stato purificato con la tecnica di estrazione del DNA fenolo-cloroformio (Guerrero-Mandujano et al., 2015b).
Risultati
Amplificazioni PCR del gene stx1 e stx2
Reazione a catena della polimerasi Lo screening di 66 ceppi clinici di Aeromonas ha mostrato che 22/66 (33,3%) ceppi contenevano il gene stx1, 42/66 (63,6%) ceppi contenevano entrambi i geni, nessun ceppo conteneva solo il gene stx2 e 2/66 (3%) ceppi erano negativi per entrambi i geni.
Sequenziamento del DNA
L’analisi BLASTn ha mostrato una somiglianza dal 79 al 99% e un valore atteso di 3e-16 / 2e-97 tra i geni stx-1/stx-2 di E. coli O157: H7 e l’amplicone di Aeromonas spp., ceppo.
Analisi citotossica e determinazione LD50
Il test di citotossicità eseguito in colture cellulari Vero ha mostrato che 17/66 (25,7%) preparati batterici privi di cellule hanno causato danni citotossici, suggerendo la produzione di un Stx attivo (Figura 1), rivelato dal danno caratteristico causato da Stx.
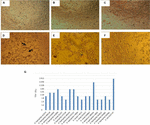
FIGURA 1. Analisi di citotossicità e determinazione della LD50 eseguita in colture cellulari Vero. (A) Coltura cellulare Vero con mezzo essenziale minimo (test di vitalità). (B) Coltura cellulare Vero con Craig Medium (test di vitalità). C) Controllo negativo con E. coli K12. (D) Controllo positivo con E. coli O157: H7, il danno citotossico è mostrato (frecce) dalla distruzione del monostrato cellulare e dall’effetto “vecchio pizzo”. (E) Danno citotossico del ceppo A. hydrophila F-0050. F) Danno citotossico del ceppo A. caviae 421423. G) Rappresentazione grafica del titolo LD50 di tutti i ceppi.
I risultati per determinare la DL50 del titolo sono state le seguenti: 6/17 (35.2%) cellula batterica preparazioni presentato un titolo di 1:16; 5/17 (il 29,4%) cellula batterica preparazioni raffigurato un titolo di 1:8; 3/17 (17.6%) cellula batterica preparazioni aveva un titolo di 1:64; 2/17 (11.7%) cellula batterica preparati con un titolo di 1:32; 1/17 (5.8%) senza cellula batterica preparati con un titolo di 1:256 (Figura 1G).
Neutralizzazione della tossina
L’applicazione di anti-STX1 Ab in 11/17 (64 .7%) ceppi il danno alle cellule nel surnatante è stato completamente inibito a 39,1 µg / µL di concentrazione Ab; in 4/17 (23,5%) preparati batterici senza cellule, è stato osservato un danno cellulare ridotto alla stessa concentrazione; e in 2/17 (11,7%) preparati batterici senza cellule, il danno cellulare è stato completamente inibito a una concentrazione 78,2 µg/µL. L’utilizzo dell’Ab anti-STX2 ha mostrato che in 9/17 (52,9%) preparati batterici senza cellule, il danno cellulare è stato totalmente inibito a 37,4 µg/µL e 6/17 (35,2%) preparati batterici senza cellule hanno mostrato un danno cellulare ridotto alla stessa concentrazione; e in 2/17 (11.7%) preparati batterici senza cellule, il danno cellulare è stato completamente inibito con una concentrazione Ab di 74,8 µg / µL (Figure 2 e 3).

FIGURA 2. Inibizione dell’effetto citotossico indotto nella coltura di cellule Vero da preparati batterici senza cellule di Aeromonas spp. ceppi portatori del gene stx1 o stx2.
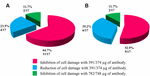
FIGURA 3. Percentuale di ceppi che hanno mostrato inibizione o riduzione del danno cellulare. (A) Neutralizzazione con l’anti-Stx1 Ab. (B) Neutralizzazione con l’anti-Stx2 Ab.
le amplificazioni PCR del gene stx1 da OMVs
La reazione a catena della polimerasi del DNA di OMVs di A. hydrophila F-0050 ha rivelato che gli OMV contenevano il gen stx1; quindi, gli OMV potrebbero essere un potenziale veicolo per il trasferimento orizzontale dei geni di virulenza (Figura 4).
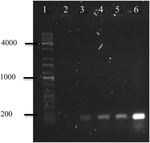
FIGURA 4. Elettroferogramma di amplificazione del gene stx1 dal DNA di OMVs. (1) Scala universale Kapa, (2) Controllo negativo, (3) OMV di A. hydrophila F-0050, (4) A. hydrophila F-0050, (5) OMV di E. coli O157:H7, e (6) E. coli O157: H7.
Discussione
Il genere Aeromonas, come produttore di Stx, potrebbe essere un patogeno emergente che causa diarrea principalmente nei pazienti pediatrici (Hernández-Cortez et al., 2011; Figueras e Baez-Hidalgo, 2014). Tuttavia, questo genere è diventato più rilevante nell’area medica, in quanto è un agente patogeno che causa HUS (San Joaquín e Pickett, 1988; Bogdanović et al., 1991; Robson et al., 1992; Fang et al., 1999; Monforte-Cirac et al., 2010).
In questo studio, abbiamo trovato, mediante amplificazione PCR dei geni stx1 e stx2, che 22/66 (33.3%) dei ceppi aeromonad conteneva il gene stx1, 42/66 (63,6%) ceppi contenevano entrambi i geni, e non c’erano ceppi contenenti stx2 da solo. Questo ci può portare a suggerire che questo gene è ampiamente distribuito nei ceppi di Aeromonas isolati da pazienti pediatrici e che questi pazienti possono essere a rischio di sviluppare HUS.
C’è solo un rapporto precedente in cui i geni stx sono stati studiati in ceppi Aeromonas isolati da diversi ospedali in Spagna; i risultati ottenuti sono stati che 19/80 (23,7%) ceppi avevano il gene stx1 e solo 1/80 (1.25%) il ceppo era positivo per entrambi i geni (Alperi e Figueras, 2010). Questi risultati trasmettono che c’è una maggiore distribuzione del gene nei ceppi messicani; tuttavia, dobbiamo tenere conto che, in Spagna, il genere Aeromonas è un patogeno gastrointestinale noto isolato di routine in pazienti con un profilo gastrointestinale, quindi, viene dato un trattamento adeguato per affrontare questo genere.
In Messico, questo genere non è incluso nella lista dei patogeni che causano la diarrea e non è isolato di routine; di conseguenza, è difficile dare qualsiasi trattamento e buona qualità della vita ai pazienti; inoltre, non ci sono statistiche sulla sua prevalenza. Inoltre, il genere diventa più rilevante a causa delle condizioni socioeconomiche della popolazione messicana, che includono la carenza d’acqua; questo facilita la sua trasmissione e aumenta la possibilità che la HUS diventi un problema di salute pubblica.
È stato eseguito il sequenziamento degli ampliconi ed è stata eseguita una ricerca Blast, che ha mostrato una somiglianza del 79-99% e un valore di aspettativa di 3e-16/2e-97 tra i geni stx1 / stx2 rispetto a E. coli O157:H7.
La LD50 è stata determinata per valutare il danno citotossico nella coltura cellulare di Vero; con questo parametro e sulla base di Marques et al. (1986), i ceppi possono essere raggruppati in bassa (2 × 101 a 6 × 102), moderata (103-104), e alta (105-108) produzione (Figura 1G). Marques et al. (1986) hanno condotto uno studio in cui hanno effettuato un test simile a quello del presente studio, ma con diversi ceppi di STEC, hanno ottenuto che 262/400 (63%) ceppi sono stati raggruppati come produttori di Stx bassi e il 48% ha prodotto danni citotossici; tuttavia, nel 40% di questi ceppi il danno citotossico è stato inibito con anticorpi anti-Stx.
Nel presente studio, il 25,7% dei batteri-gratis preparazioni causato citotossici caratteristica danni di Stx in cellule Vero, suggerendo produzione di active Stx, tutti Aeromonas ceppi sono stati raggruppati come bassa produttori di Stx; tuttavia, nel 50-70% dei cellulari gratis supernatanti, il danno cellulare è stato inibito, e nel 20-30% dei cellulari gratis supernatanti il danno cellulare è stato ridotto (Figura 3). I supernatanti senza cellule in cui il danno cellulare è stato ridotto questo potrebbe essere stato a causa di due varianti nella tossina presente nel surnatante, di conseguenza, poiché sono diversi immunologicamente, lo Stx non poteva essere totalmente inibito e il danno cellulare è rimasto (Marques et al., 1986).
Due studi precedenti (Haque et al., 1996; Alperi e Figueras, 2010) hanno ottenuto rispettivamente il 10,2% e il 10,53% dei ceppi Aeromonas produttori di Stx1. La ragione di una maggiore percentuale di ceppi Aeromonas che producono Stx attivo potrebbe essere dovuta all’elevata disponibilità del batteriofago Stx all’interno della coltura poiché alcuni ceppi STEC portatori del batteriofago Stx sono stati isolati dalle acque reflue urbane degli impianti di trattamento, dalle acque reflue dei macelli e dalle feci di bestiame (García-Aljaro et al., 2004, 2009). Allo stesso modo sono stati condotti studi di trasduzione in vivo con fagi Stx di E. coli, che possono infettare i batteri del microbiota intestinale, dando la caratteristica tossigenica a ceppi che non erano stati infettati prima (Acheson et al., 1998; Schmidt et al., 1999; Gamage et al., 2003). Lo stesso potrebbe accadere con il genere Aeromonas quando si induce un profilo gastrointestinale, seguito dal fatto che il genere Aeromonas ha la capacità di catturare e integrare fattori di virulenza nel suo genoma, e uno di questi è il gene codificante Stx.
Il resto dei ceppi ha causato danni citotonici nella cellula, suggerendo probabilmente la produzione di diverse tossine diverse da Stx, poiché Aeromonas è un genere in grado di produrre due enterotossine citotoniche, termostabili (AST) e termolabili (ALT) (Figueras e Baez-Hidalgo, 2014), che possono causare danni citotonici nelle cellule Vero; un’altra possibilità è che i ceppi Aeromonas abbiano più di un batteriofago inserito nel loro genoma, poiché è stato dimostrato che i ceppi STEC con doppia lisogenesi sono in grado di regolare i batteriofagi per inibire la capacità di induzione del ciclo litico, il che si traduce in una diminuzione della produzione di Stx ma senza perdere il gene (García-Aljaro et al., 2004; Imamovic e Muniesa, 2011). Pertanto, è necessario stabilire quanti batteriofagi contengono i ceppi di Aeromonas, incluso nel presente studio, per essere in grado di correlare la presenza di batteriofagi Stx e la quantità prodotta di Stx.
D’altra parte, la presenza di batteriofagi Stx in Aeromonas inducibili rappresenta un meccanismo di trasferimento orizzontale del gene stx, ma non è l’unico. In studi più recenti, è stato determinato che gli Aeromonas sono in grado di produrre OMV (Guerrero-Mandujano et al., 2015a) e questi OMV sono in grado di trasportare DNA integrato (Guerrero-Mandujano et al., 2015b). Nel presente studio, abbiamo amplificato il gene stx1 dal DNA estratto dagli OMV di un ceppo A. hydrophila di origine clinica, che ha indotto danni citotossici nelle cellule Vero, e questo danno è stato inibito con anticorpi anti-Stx. Ciò indica che il batteriofago Stx è il meccanismo di trasporto del gene stx; inoltre, gli OMV e il sistema di trasporto recentemente chiamato Tipo 0 sono in grado di diffondere il gene stx interspecie o intraspecie. Tuttavia, è necessario confermare se il gene contenuto nell’OMVs è completo. I risultati ottenuti in questo studio supportano l’ipotesi che il gene stx sia un altro fattore di virulenza putativo di Aeromonas che potrebbe essere trasferito attraverso OMV. Potrebbe rappresentare un altro meccanismo per il trasporto orizzontale del gene stx verso altri ceppi di Aeromonas e / o altri generi batterici.
Per questo motivo, questo genere dovrebbe essere incluso come possibile agente causale della gastroenterite quando si cerca di identificare gli agenti causali e dovrebbe essere segnalato come parte delle procedure standard di sorveglianza sanitaria.
Contributi dell’autore
IP-M e AG-M eseguirono gli esperimenti e MR-R eseguirono le analisi bioinformatiche e redassero il manoscritto; CH-C progettò il primer; VB-G e JM-L commentarono e revisionarono criticamente il manoscritto; GC-E concepì lo studio, partecipò alla sua progettazione e coordinamento, valutò i dati e redasse il manoscritto. Tutti gli autori hanno letto e approvato il manoscritto finale. Ringraziamo Sofia Marteli Mulia per aver gentilmente corretto lo stile del manoscritto.
Finanziamenti
GC-E e VB-G hanno ricevuto il sostegno di Estímulos al Desempeño en Investigación e Comisión y Fomento de Actividades Académicas (Instituto Politécnico Nacional) e Sistema Nacional de Investigadores (SNI, CONACyT). Questo studio è stato finanziato dal Segretariato di Ricerca e post-laurea del National Polytechnic Institute (SIP 20160609). Il SIP-IPN non è stato coinvolto nello sviluppo della progettazione dello studio, nella raccolta, nell’analisi e nell’interpretazione dei dati, nella stesura del rapporto né nella decisione di presentare il documento per la pubblicazione. IP – M e AG-M hanno tenuto una borsa di studio da CONACyT e BEIFI.
Dichiarazione sul conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Ringraziamenti
Ringraziamo la Dott. ssa Patricia Arzate Barbosa per aver fornito ceppi isolati e il Dott. Armando Navarro per aver fornito gli anticorpi monoclonali.
Abbreviazioni
Ab, Anticorpo; HUS, Sindrome emolitica uremica; INP, Instituto Nacional de Pediatria Mexico; LD50, Dose letale mediana; OMVs, Outer membrane vesicles; PCR, Polymerase chain reaction; STEC, Shiga toxin-producing E. coli; Stx, Shiga-like toxin; TTP, Thrombotic thrombocytopenic purpura.
Acheson, D. W., Reidl, J., Zhang, X., Keusch, G. T., Mekalanos, J. J., and Waldor, M. K. (1998). In vivo transduction with shiga toxin 1-encoding phage. Infect. Immun. 66, 4496–4498.
Google Scholar
Alperi, A., and Figueras, M. J. (2010). Gli isolati umani di Aeromonas possiedono i geni della tossina di shiga sono (stx1 e stx2) altamente simili alle varianti virulente del gene del ponte di Escherichia coli. Clin. Microbiolo. Infettare. 16, 1563-1567. doi: 10.1111 / j. 1469-0691. 2010.03203.x
CrossRef Full Text/Google Scholar
Bogdanovic, R., Čobeljić, M., Markovic, M., Nikolic, V., Ognjanović, M., Sarjanović, L., et al. (1991). Sindrome emolitico-uremica associata a enterocolite Aeromonas hydrophila. Pediatria. Nephrol. 5, 293-295. doi: 10.1007
CrossRef Testo Completo | Google Scholar
Castro-Escarpulli, G., Aguilera-Arreola, M. G., Giono-Cerezo, S., Hernández-Rodríguez, C. H., Rodriguez-Chacon, M., Soler, F. L., et al. (2002). Il genere Aeromonas > Un importante patogeno in Messico? Enf. Infec. Mike. 22, 206–216.
Google Scholar
Castro-Escarpulli, G., Figueras, M. J., Aguilera-Arreola, M. G., Soler, L., Fernández-Rendón, E., Aparicio, G. O., et al. (2003). Caratterizzazione di Aeromonas spp. isolato dal pesce congelato destinato al consumo umano in Messico. Int. J. Microbiolo alimentare. 84, 41–49. doi: 10.1016/S0168-1605 (02)00393-8
CrossRef Full Text/Google Scholar
Fang, JS, Chen, JB, Chen, WJ, e Hsu, KT (1999). Sindrome emolitico-ureemica in un maschio adulto con enterocolite Aeromonas hydrophila. Nephrol. Quadrante. Trapianto. 14, 439–440. doi: 10.1093/ndt/14.2.439
CrossRef Full Text/Google Scholar
Figueras, MJ e Baez-Hidalgo, R. (2014). “Aeromonas”, in Encyclopedia of Food Microbiology, eds C. A. Batt e M. L. Tortorello (Cambridge, MA: Academic Press), 24-30.
Gamage, S. D., Strasser, J. E., Chalk, C. L., e Weiss, A. A. (2003). L’Escherichia coli non patogeno può contribuire alla produzione di tossina shiga. Infettare. Immun. 71, 3107–3115. doi: 10.1128 / IAI.71.6.3107-3115.2003
CrossRef Full Text/Google Scholar
García-Aljaro, C., Muniesa, M., Jofre, J., and Blanch, A. R. (2004). Prevalenza del gene stx2 in popolazioni coliformi provenienti da ambienti acquatici. Appl. Ambiente. Microbiolo. 70, 3535–3540. doi: 10.1128 / AEM.70.6.3535-3540.2004
CrossRef Full Text/Google Scholar
García-Aljaro, C., Muniesa, M., Jofre, J., and Blanch, A. R. (2009). Diversità genotipica e fenotipica tra batteriofagi indotti portatori di stx2 da ceppi ambientali di Escherichia coli. Appl. Ambiente. Microbiolo. 75, 329–336. doi: 10.1128 / AEM.01367-08
CrossRef Full Text/Google Scholar
Giono-Cerezo, S., Rodríguez-ÁnGeles, M. G., Rodríguez-Cadena, M. J., e Valdespino-Gómez, J. L. (1994). Identificazione di enterotossine e citotossine di Escherichia coli mediante coltura cellulare vero e ibridazione in fase solida (colony blot). Rev. Latinoam. Microbiolo. 36, 231–241.
Google Scholar
Guerrero-Mandujano, A., López-Villegas, E. O., Longa-Briseño, A., e Castro-Escarpulli, G. (2015a). Produzione di vescicole di membrana esterna in un ceppo clinico di Aeromonas hydrophila. Microbiolo CE. 1, 113–117.
Google Scholar
Guerrero-Mandujano, A., Palma-Martínez, I., e Castro-Escarpulli, G. (2015b). Trasportare il DNA dalle vescicole della membrana esterna di Aeromonas hydrophila. Microbiolo CE. 2, 354–357.
Google Scholar
Haque, Q. M., Sugiyama, A., Iwade, Y., Midorikawa, Y., e Yamauchi, T. (1996). Isolati diarroici e ambientali di Aeromonas spp. produrre una tossina simile alla tossina shiga-like 1. Curr. Microbiolo. 32, 239–245. doi: 10.1007/s002849900043
CrossRef Testo Completo | Google Scholar
Hernández-Cortez, C. Aguilera-Arreola, M. G., Chávez-Méndez, Y., Arzate-Barbosa, P., Lara-Hernandez, A., e Castro-Escarpulli, G. (2011). Identificazione genetica di ceppi di Aeromonas spp. isolato presso l’istituto nazionale di pediatria. Rev. Enfer. Infecc. Pediatrico. 24, 157–165.
Google Scholar
Hernández-Cortez, C., Méndez-Tenorio, A., Aguilera-Arreola, M. G., e Castro-Escarpulli, G. (2013). Progettazione e standardizzazione di quattro reazioni a catena della polimerasi multiplex per rilevare i batteri che causano malattie gastrointestinali. Afr. J. Microbiolo. Res. 7, 2673-2681. doi: 10.5897/AJMR2013.5452
CrossRef Full Text/Google Scholar
Imamovic, L., and Muniesa, M. (2011). Quantificazione e valutazione dell’infettività dei batteriofagi codificanti per la tossina shiga nella carne bovina e nell’insalata. Appl. Ambiente. Microbiolo. 77, 3536–3540. doi: 10.1128 / AEM.02703-10
CrossRef Full Text/Google Scholar
Marques, L. R. Nel 1986, dopo la morte del padre, si trasferì a New York, dove rimase fino al 1986. Produzione di tossina simile a shiga da Escherichia coli. J. Infettare. Dis. 154, 338–341. doi: 10.1093/infdis/154.2.338
CrossRef Full Text/Google Scholar
Mauro, SA, e Koudelka, GB (2011). Shiga tossina: espressione, distribuzione, e il suo ruolo nell’ambiente. Tossine 3, 608-625. doi: 10.3390/toxins3060608
CrossRef Full Text/Google Scholar
Monforte-Cirac, M. L., Palacian-Ruiz, M. P., Aisa-Iriarte, M. L., e Espuelas-Monge, M. (2010). Sindrome emolitica uremica di Aeromonas: su un caso clinico. Rev. Clin. Spirito. 210, 206–207. doi: 10.1016 / j.cer.2009.08.002
CrossRef Full Text/Google Scholar
Robson, W. L. M., Leung, A. K. C., e Trevenen, C. L. (1992). Sindrome emolitico-uremica associata a enterocolite Aeromonas hydrophila. Pediatrico. Nephrol. 6, 221–222. doi: 10.1007
CrossRef Testo Completo / Google Scholar
San Joaquin, V. H., e Pickett, D. A. (1988). Gastroenterite associata ad Aeromonas nei bambini. Pediatria. Infettare. Dis. J. 7, 53-57. doi: 10.1097 | 00006454-198801000-00013
CrossRef Full Text/Google Scholar
Schmidt, H., Bielaszewska, M., e Karch, H. (1999). Trasduzione di Escherichia coli enterico isolati con un derivato della tossina shiga 2-codifica batteriofago f3538 isolato da Escherichia coli O157: H7. Appl. Ambiente. Microbiolo. 65, 3855–3861.
Google Scholar