Introduzione
Internazionale politica di conservazione riconosce la biodiversità a tre livelli, ecosistema, la specie e la genetica, e che la gestione dovrebbe mirare a mantenere tutti e tre (Convenzione sulla Diversità Biologica, 2007). Ciò si riflette chiaramente nella Convenzione sulla diversità biologica, Aichi Biodiversity Targets, concordata nel 2010 (https://www.cbd.int/sp/targets/), in cui vi è un riferimento specifico negli obiettivi e negli obiettivi, non solo agli ecosistemi e alle specie, ma anche alla diversità genetica. Tuttavia, gli attuali approcci alla conservazione della biodiversità si basano in gran parte su aree geografiche, ecosistemi, comunità ecologiche e specie, con una minore attenzione alla diversità genetica e al continuum specie-popolazione. In effetti, la maggior parte degli operatori della conservazione si concentra sulle specie come metrica della biodiversità, con una certa considerazione delle unità intraspecifiche all’interno delle specie minacciate gestite intensivamente. In questo contesto, c’è un naturale desiderio di impiegare semplici categorizzazioni: specie denominate e altri taxa denominati (cioè sottospecie) e talvolta unità di conservazione all’interno delle specie. Le specie in particolare forniscono una misura comune della biodiversità che è alla base di molti sforzi scientifici e gestionali (Mace, 2004; Wilson, 2017). Come unità fondamentale di conservazione, le specie sono spesso utilizzate per quantificare il valore della biodiversità attraverso elenchi di specie all’interno di aree protette, identificazione di specie minacciate all’interno di giurisdizioni e come base per le procedure di biosicurezza. Inoltre, le specie sono in genere le entità con cui comunichiamo problemi di conservazione al pubblico.
Questo impulso a identificare semplicemente unità discrete in natura è in contrasto con diverse realtà correlate. Innanzitutto, la speciazione non è istantanea. Piuttosto è tipicamente un processo prolungato (Dynesius e Jansson, 2014; Roux et al., 2016), procedendo da popolazioni isolate, forse soggette a diverse pressioni di selezione, a specie inequivocabili su scale temporali lunghe, con frequenti inversioni causate da cambiamenti ambientali o semplici fusioni genetiche quando gli intervalli si riconnettono (Figura 1; Seehausen et al., 2008; Rosenblum et al., 2012). In secondo luogo, la maggior parte delle specie riconosciute non sono geneticamente uniformi. La divergenza (deriva e isolamento prolungato) e la reticolazione (geneflow, ibridazione, introgressione e ricombinazione) modellano efficacemente la variazione geografica e genomica all’interno delle specie (Cutter e Payseur, 2013; Edwards et al., 2016) e alcune specie possono essere strutturate in lunghe popolazioni isolate, alcune delle quali potrebbero giustificare il riconoscimento come specie complete ma forse morfologicamente criptiche (Bickford et al., 2007; Jorger e Schrödl, 2013). In terzo luogo, come riconosciuto a lungo, le specie nominate non sono equivalenti in termini di quanta eredità evolutiva rappresentino—in parole povere, un ornitorinco (come rappresentante di un lignaggio antico divergente) può essere considerato più di un roditore recentemente divergente (Isaac et al., 2007). In quarto luogo, le designazioni delle specie non sono gettate nella pietra, piuttosto sono ipotesi da testare man mano che nuove prove diventano disponibili (Hey et al., 2003). Ognuno di questi problemi crea complessità e ambiguità nel delimitare le specie in generale e nell’assegnare attenzione alla conservazione a loro o alle loro popolazioni componenti. Inoltre, la delimitazione delle specie è anche soggetta a diversi concetti di specie e modi di diagnosticarle, il che ha causato grande costernazione tra i biologi della conservazione (Mace, 2004; Frankham et al., 2012; Garnett e Christidis, 2017).
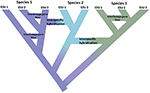
Figura 1. Speciazione come un continuum che progredisce da popolazioni isolate a specie inequivocabili mostrate dallo spostamento di colore con i colori che rappresentano diverse specie. Alcuni lignaggi che sorgono a causa dell’isolamento della popolazione si sviluppano in vere specie con i nodi interni dell’albero che rappresentano l’inizio del processo di speciazione. Altri lignaggi non si sviluppano in vere specie ma divergono nel tempo indicando che le specie non sono geneticamente uniformi. Questi lignaggi sono mostrati come Unità evolutivamente significative (ESU). Il flusso genico inter-lineage è indicato per la specie 1 e la specie 3. In questo caso, l’indipendenza evolutiva può essere transitoria perché due lignaggi appartenenti alla stessa specie non sono isolati riproduttivamente. Mentre rimangono come lignaggi distinti (specie), l’ibridazione interspecifica può verificarsi con uno scambio genico limitato (spostamento di colore) tra le specie.
L’implementazione di efficaci strategie di conservazione che mirano a proteggere sia le specie che la diversità genetica beneficerebbe sia di una visione più coerente di come delimitiamo le specie e le altre unità nel continuum popolazione-specie, sia di come queste unità sono interpretate in diverse legislazioni a livello globale. Le nuove funzionalità della genomica offrono sia opportunità che sfide per raggiungere questi obiettivi. Nel seguito consideriamo: (i) come il nostro genoma-scala di analisi possono essere riconciliati con un approccio conservativo per la delimitazione e la denominazione della specie, ma anche di identificare i principali componenti della diversità genetica all’interno delle specie, (ii) come esistente normativa in materia di conservazione diversi continenti, che definisce “specie” per includere una vasta gamma di infra-categorie specifiche, può essere utilizzato, data l’elevata risoluzione offerta dal genoma schermi, e (iii) come le intuizioni di speciazione e processi di strutturazione di variazione genomica di allineare la legislazione e quindi nella gestione. Suggeriamo quindi alcuni modi per allineare le unità di conservazione agli approcci legislativi al fine di proteggere l’intero spettro della diversità dalle popolazioni alle specie.
Genomica, speciazione e tassonomia
Come molti campi della scienza biologica, la sistematica, la pratica di nominare i taxa e comprendere le loro relazioni e come si formano, viene significativamente avanzata dalla genomica e dai modelli statistici. Sia attraverso il sequenziamento dell’intero genoma (ad esempio, Nater et al., 2017) o campionamento del genoma (SNPs, cattura-sequenziamento del bersaglio; Jones e Good, 2016; Leache e Oaks, 2017), ora abbiamo una risoluzione senza precedenti di modelli di diversità genetica (Funk et al., 2012). Accoppiato con modelli statistici sempre più potenti per inferire storie di divergenza genomica e specie, questi dati stanno fornendo nuove intuizioni nei processi evolutivi che generano specie e variazione genetica.
L’applicazione di questi nuovi strumenti genera due intuizioni opposte. Da un lato, molti studi stanno rivelando popolazioni genetiche altamente divergenti all’interno di specie denominate, le cosiddette specie criptiche (Bickford et al., 2007; Jorger e Schrödl, 2013; Struck et al., 2017). Questo non è nulla di nuovo, in quanto si basa su una lunga storia di genetica delle popolazioni spaziali e filogeografia molecolare che è stato uno sforzo produttivo per tre decenni (Avise et al., 1987). Ciò che la genomica porta a questo sforzo è sia una maggiore risoluzione della struttura della popolazione che dati sufficienti per inferire statisticamente le storie della popolazione e testare modelli alternativi di divergenza (Degnan e Rosenberg, 2009; Sukumaran e Knowles, 2017). D’altra parte, le analisi su scala genomica stanno anche rivelando che (come da tempo riconosciuto per le piante), lo scambio genetico (introgressione) tra le specie animali è più comune di quanto si pensasse in precedenza, sia durante che dopo la speciazione, e può persino guidare nuove radiazioni adattive (Mallet, 2007; Rieseberg e Willis, 2007; Arnold e Kunte, 2017) Figura 1. Messi insieme, queste intuizioni sottolineano ulteriormente che la speciazione è protratta (Roux et al., 2016) e rivelano le forze opposte che operano attraverso il continuum filogeografico-filogenetico (Edwards et al., 2016).
In che modo tutto questo si collega con gli obiettivi di tassonomia e conservazione? C’è una forte e giustificabile preoccupazione che l’aumento della risoluzione offerta dagli schermi del genoma possa portare a una dilagante sovra-suddivisione delle specie (Isaac et al., 2004), potenzialmente limitando la flessibilità di gestione e consegnando piccole popolazioni geneticamente divergenti all’inbreeding e all’eventuale estinzione (Frankham et al., 2012). Al contrario, Gippoliti et al. (2018) sostengono che l’inerzia tassonomica, con conseguente precedente incapacità di riconoscere molte specie di ungulati africani, aveva provocato una depressione di outbreeding dopo la miscelazione e la mancata protezione dei taxa geograficamente limitati. Il loro punto di vista (e vedi anche Morrison et al., 2009) è che non ci sono prove che l’inflazione tassonomica stia avendo effetti negativi sulla conservazione degli ungulati (Gippoliti et al., 2018). Una preoccupazione correlata è che la tassonomia instabile ritarda sia la pratica della conservazione che le percezioni pubbliche (Garnett e Christidis, 2017). Queste preoccupazioni sono in parte guidate dal lungo dibattito sui concetti di specie e sui metodi diagnostici in biologia sistematica, in particolare l’applicazione del Concetto di specie filogenetica che sostiene che le specie sono unità minimamente diagnosticabili (Cracraft, 1983). Tuttavia, la maggior parte dei tassonomisti è intrinsecamente conservatrice, nominando le specie solo quando c’è concordanza su più linee di evidenza (Daylat, 2005; Padial et al., 2010; Schlick-Steiner et al., 2010: Yeates et al., 2011). La nostra opinione è che le specie dovrebbero rappresentare robusti lignaggi in evoluzione indipendente che rimangono in gran parte intatti quando sono in contatto con parenti stretti—in accordo con l’intento del Concetto di specie evolutiva (Wiley, 1978) e del Concetto di specie di Lignaggio generalizzato (de Queiroz, 1998, 2007). In pratica, ciò richiede prove dirette di isolamento riproduttivo o surrogati ragionevoli dello stesso, pur consentendo uno scambio genetico limitato (Singhal et al., 2018).
I dati genomici forniscono una maggiore potenza rispetto a prima per identificare la differenziazione e la divergenza all’interno e tra le specie, richiedendo una maggiore considerazione di come ciò influenzi la delineazione delle unità tassonomiche. Dove le popolazioni sono sia fortemente geneticamente che fenotipicamente divergenti rispetto alle specie già nominate, c’è un forte caso per denominarle come specie separate. Se le popolazioni sono fenotipicamente fortemente divergenti con una scarsa separazione genomica, allora c’è una richiesta di giudizio sul fatto che tali differenze fenotipiche siano (i) ereditabili e (ii) suscettibili di causare un sostanziale isolamento riproduttivo. Le scansioni del genoma possono risolvere tali situazioni testando il flusso genico limitato. Il problema più impegnativo è dove vengono rilevate popolazioni morfologicamente simili, ma geneticamente divergenti. Tali popolazioni possono essere facilmente diagnosticate come lignaggi evolutivi separati applicando metodi di delimitazione statistica ai dati multilocus (Carstens et al., 2013; Rannala, 2015), ma sono popolazioni effimere o specie durevoli (Sukumaran e Knowles, 2017)? È certamente possibile, all’interno delle regole di nomenclatura, riconoscere le specie dalle sole sequenze di DNA, fornendo una diagnostica basata sulla sequenza (Tautz et al., 2003; Cook et al., 2010). Ad esempio, Murphy et al. (2015) delimitano e descrivono sei specie di anfipodi primaverili del deserto australiano sulla base di dati sulla sequenza del DNA nonostante non ci siano chiare differenze morfologiche. Tuttavia, questa è un’area generale di miglioramento in cui i sistematisti devono applicare il termine “specie criptiche” con maggiore coerenza e rigore (Struck et al., 2017).
In situazioni in cui i dati sulla sequenza del DNA forniscono una potenziale caratteristica chiave per il riconoscimento di una specie, suggeriamo che è particolarmente importante applicare altre linee di prova per evitare l’oversplitting (Oliver et al., 2015). Un approccio consiste nel testare un sostanziale isolamento riproduttivo quando tali popolazioni formano zone di contatto naturali. Ad esempio, in un’analisi genomica comparativa delle zone di contatto tra coppie di lignaggi di lucertole Carlia che sono fenotipicamente indistinguibili, gli indici di isolamento genetico erano fortemente correlati al tempo di divergenza (Singhal e Moritz, 2013; Singhal e Bi, 2017) (Figura 2). Le più divergenti di queste coppie di stirpi sono state formalmente riconosciute come specie separate, mentre i lignaggi più strettamente correlati (ma statisticamente diagnosticabili) con evidenza di uno scambio genetico genomicamente esteso tra le zone di contatto non lo erano (Singhal et al., 2018). Data la forte relazione empirica tra divergenza e isolamento riproduttivo in questi taxa, questi autori hanno ulteriormente separato due isolati allopatrici e criptiche (di una specie strettamente correlata) che avevano una divergenza genomica ancora più elevata di quelle delimitate sopra. L’estensione di questo isolamento riproduttivo ha informato la metrica ad altre specie congeneriche con lignaggi filogeografici profondamente divergenti ma morfologicamente simili (Potter et al., 2016, 2018; Afonso Silva et al., 2017a) suggerisce che più specie rimangono da descrivere, specialmente sulle isole, (Figura 2, Carlai amax e C. rufilatus), e in effetti, alcune di queste specie sono state descritte in seguito ad analisi fenotipiche complementari (Afonso Silva et al., 2017b). Altri lignaggi con divergenze più recenti, ma ancora delimitati statisticamente come lignaggi separati utilizzando un ampio insieme di geni nucleari, possono essere rappresentati come Unità evolutivamente significative (ESU) all’interno delle specie.
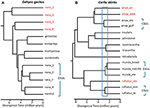
Figura 2. Risoluzione filogenomica dei confini delle specie nelle lucertole dei Tropici monsonici australiani (AMT). In ogni caso le relazioni e i tempi di divergenza relativi sono stimati da 100 esoni utilizzando la coalescenza multispecie implementata in StarBEAST2, ma le scale temporali tra i due esempi non sono necessariamente le stesse. (A) Risultati per il gruppo nana di gechi Gehyra (Moritz et al., 2018). La diversità filogenetica dei lignaggi all’interno di G. nana, come precedentemente riconosciuto, comprendeva altre quattro specie già descritte e morfologicamente distintive. Successive analisi delle sovrapposizioni di gamma e della morfologia hanno portato al riconoscimento di ciascuna delle linee filogeneticamente più divergenti (etichette di taxon rosso) come specie separate (Doughty et al., 2018). I lignaggi (morfologicamente inseparabili) rimasti in G. nana come ora riconosciuti possono essere considerati ESU. (B) Risoluzione in corso dei confini di specie in Carlia scinchi dal AMT. Alcuni lignaggi sono già stati riconosciuti come specie sulla base di una combinazione di delimitazione genetica delle specie e identificazione post hoc delle differenze morfologiche diagnostiche (C. insularis e C. isostriacantha; Afonso Silva et al., 2017b). Per altri complessi (Potter et al., 2016, 2018), revisioni tassonomiche sono in corso. Il rettangolo blu indica l’intervallo di tempi di divergenza in cui è stato osservato un sostanziale isolamento riproduttivo, correlato alla divergenza, nelle zone di contatto tra lignaggi morfologicamente criptici di specie affini (Singhal e Bi, 2017). Conservativamente, e in seguito (Singhal et al., 2018), riconosciamo come specie candidate tre lignaggi genetici con divergenze maggiori di questa soglia empirica. Questo lascerebbe ESU geneticamente distinti all’interno di ciascuno di Carlia amax, Carlia munda e Carlia rufilatus (parentesi blu). Le barre di scala rappresentano stime del tempo di divergenza in milioni di anni.
In casi estremi, i metodi genomici rivelano che le singole specie tassonomiche non solo sono geneticamente eterogenee, ma, in senso filogenetico, contengono anche altre specie nominate. Tale “parafilia” può sorgere anche se recente speciazione da una singola specie geneticamente suddivisa (Patton e Smith, 1994), ma genera anche l’ipotesi che il taxon geneticamente eterogeneo possa rappresentare più specie. Ancora una volta, sono necessarie ulteriori prove. Come esempio, l’analisi filogenomica di un complesso di lucertole Gehyra gecko dell’Australia settentrionale delimitava nove lignaggi evolutivi, con quattro specie già nominate filogeneticamente incorporate tra loro (Moritz et al., 2018; Figura 2). Successive prove di sovrapposizione geografica (pur mantenendo la separazione filogenetica) e sottili differenze morfologiche supportato denominazione dei quattro lignaggi filogeneticamente più divergenti (Doughty et al., 2018). Al contrario, i lignaggi più strettamente correlati (ma statisticamente separabili) avevano distribuzioni adiacenti piuttosto che sovrapposte, ma non erano morfologicamente diagnosticabili e, di conseguenza, sono stati mantenuti come una singola specie (G. nana sensu stricto), anche se con ESU multipli, in attesa di ulteriori indagini (Figura 2). Successive scansioni del genoma di campioni attraverso i confini di questi ESU hanno rivelato che almeno alcuni hanno uno scambio genetico sostanziale, convalidando l’approccio originale e conservativo adottato per riconoscere le specie.
In altri casi, gli studi genomici delle piante, anche attraverso una semplice ordinazione di SNPS, possono rivelare divergenze stark e impreviste tra campioni di popolazione, suggerendo la presenza di taxa criptici che possono quindi essere analizzati per caratteri fenotipici diagnostici. Ad esempio, le prove basate su SNP per la divergenza genomica tra le popolazioni e le differenze ecotipiche nell’eucalipto salubris hanno rivelato due distinti lignaggi molecolari che hanno mantenuto la loro distintività in un’area di sovrapposizione geografica, con l’adattamento ecotipico considerato un fattore importante nel ridurre al minimo il flusso genico tra i lignaggi. (Steane et al., 2015; Figura 3). In un altro caso, nel Pelargonium australiano, l’analisi genomica, combinata con studi morfologici e riproduttivi, ha portato a cambiamenti significativi nella tassonomia, sia spaccando che ammassando i taxa, specialmente dove i tratti morfologici precedentemente usati sono trovati per essere labili e discordanti con la filogenesi (Nicotra et al., 2016).
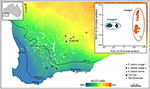
Figura 3. L’analisi genomica utilizzando i dati SNP ha identificato lignaggi criptici nel diffuso Eucalyptus salubris. La posizione dei campioni popolazioni di ogni lignaggio sono mostrati attraverso un gradiente indice di aridità nel sud ovest Australia occidentale. Il PCoA basato su distanze genetiche binarie derivate da 16.122 marcatori di DArTseq, mostra due lignaggi distinti. Adattato da Steane et al. (2015).
I casi sopra illustrano l’approccio progressivo di test di ipotesi che è stato utilizzato nella tassonomia integrativa (Padial et al., 2010; Yeates et al., 2011). Inoltre, questi casi utilizzano una qualche forma di sequenziamento della riduzione del genoma per ottenere dati di alta qualità da 1000 di geni indipendenti, dando molta più fiducia nei risultati rispetto al caso utilizzando solo loci plastid o alcuni geni nucleari. Un esempio di come l’applicazione della tassonomia integrativa possa alterare drasticamente le percezioni della diversità proviene da un’indagine genetica, acustica e morfologica combinata delle rane del Madagascar che ha quasi raddoppiato la diversità delle specie conosciute (Vieites et al., 2009). Questi autori hanno proposto una serie di termini che riflettono lo stato di risoluzione tassonomica e congruenza: Specie candidate confermate, per taxa geneticamente e fenotipicamente divergenti, ma non ancora nominati; Specie candidate non confermate per taxa geneticamente divergenti in cui la morfologia e l’acustica non erano ancora state esaminate; e Lignaggi profondamente conspecifici per entità note per essere geneticamente divergenti ma fenotipicamente criptiche. Le analisi genomiche devono ancora essere applicate a questi taxa, ma sicuramente informerebbero le decisioni sui lignaggi conspecifici rispetto alle specie candidate.
Gli approcci genomici, anche se applicati in modo conservativo, porteranno senza dubbio a un’ulteriore suddivisione dei taxa morfologicamente conservatori. Ma, al contrario, ci sono chiari esempi in cui gli stessi approcci rivelano specie sovra-divise o diagnosticate in modo errato (Nicotra et al., 2016). Naturalmente ci sono già molti studi molecolari su complessi di specie vegetali e animali basati su set di dati su scala più piccola (ad es., microsatellite, mtDNA e cpDNA) che hanno identificato dove le specie sono state diagnosticate erroneamente, o dove più specie sono state circoscritte tassonomicamente ma sono ora considerate come una singola specie. Ad esempio, Rabosky et al. (2014) ha usato le filogenie mitocondriali e nucleari del gene per rivedere il complesso scincide Ctenotus inornatus, con conseguente riduzione netta delle specie e riallineamento dei confini delle specie; i tratti morfologici applicati in precedenza, mentre convenienti per i biologi sul campo, erano altamente labili e in gran parte non corrispondevano ai confini delle specie diagnosticati geneticamente. Nelle piante alcuni degli esempi più noti si trovano nelle orchidee, come il genere europeo di orchidee Ophrys e il genere australiano di orchidee Caladenia, dove gli studi molecolari indicano significativamente meno entità che sarebbero riconosciute come specie, rispetto a quelle basate su tratti morfologici e sindromi da impollinazione pseudocopulatoria (vedi Devey et al., 2008; Swarts et al., 2014).
I dati su scala genomica, se interpretati in modo conservativo e idealmente con analisi parallele dei fenotipi, continueranno a portare a cambiamenti tassonomici, specialmente nei taxa morfologicamente conservatori (Fišer et al., 2018). Ma, in contrasto con le preoccupazioni espresse da alcuni (Garnett e Christidis, 2017), riteniamo che questa sia la corretta ricerca della sistematica evolutiva, anche se i risultati sono temporaneamente scomodi.
Genomica e unità di conservazione intraspecifica
Sebbene meno controversa della delimitazione tassonomica e del dibattito a livello di specie, ma per molti versi altrettanto complessa, è stata la discussione significativa sulla definizione e la delimitazione delle unità di conservazione come elementi importanti della diversità intraspecifica che devono essere presi in considerazione nelle azioni di conservazione (Ryder, 1986; Waples, 1991; Dizon et al., 1992; Moritz, 1994; Vogler e de Salle, 1994; Crandall et al., 2000; Fraser e Bernatchez, 2001; Avise, 2004; Funk et al., 2012) (Figura 4). Dato l’approccio cauto al riconoscimento delle specie dalle prove genomiche che sosteniamo sopra (e vedi anche Oliver et al., 2015; Singhal et al., 2018), è inevitabile che alcune specie tassonomiche conterranno ancora popolazioni isolate a lungo al loro interno. Concetti come Unità evolutivamente significative (ESU) o unità designabili (DUS) sono stati proposti per integrare la tassonomia esistente (Ryder, 1986; Waples, 1991; Moritz, 1994; Mee et al., 2015). Come per le specie, le interpretazioni e i criteri variano, ma l’intento comune è quello di riconoscere i principali elementi di diversità intraspecifica per le azioni di conservazione (Funk et al., 2012). Moritz (1994, 2002) ha sostenuto un approccio molecolare semplicistico ma pratico per identificare segmenti in evoluzione indipendente all’interno delle specie, l’asse “storico” della diversità; mentre altri hanno posto maggiore enfasi sull’asse “adattivo” per includere la diversità funzionale (Ryder, 1986; Crandall et al., 2000; Fraser e Bernatchez, 2001; Waples et al., 2001; Mee et al., 2015) (Figura 4). Qualunque siano i meriti relativi di queste alternative, è chiaro che gli strumenti tecnici e analitici della genomica della popolazione possono ora identificare in modo robusto entrambe le componenti della diversità intraspecifica (Funk et al., 2012; Hoban et al. Nel 2013, Whitlock, 2014, Yang e Rannala, 2014).
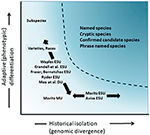
Figura 4. Separazione della diversità genetica in due componenti: variazione adattiva che nasce attraverso la selezione naturale, e analizzato attraverso l’analisi dei fenotipi e l’isolamento storico o divergenza neutra valutata attraverso la divergenza genomica. L’area ombreggiata più scura sopra la curva indica condizioni in cui è probabile che le popolazioni siano considerate come specie separate nella maggior parte dei concetti. Vari concetti di unità di conservazione e categorie tassonomiche infra-specifiche sono mostrati relativi ai due assi della diversità genetica (dopo Moritz, 2002).
Questo porta alla domanda: perché non nominare semplicemente ESU come sottospecie? Le sottospecie sono state tradizionalmente definite e viste sia dai botanici che dagli zoologi come insiemi fenotipicamente distinti e allopatrici di popolazioni che possono intergrarsi l’una nell’altra ai confini geografici (Mayr, 1942; Grant, 1981). In quanto tali riflettono potenzialmente divergenza adattiva senza necessariamente essere stati storicamente isolati. Le designazioni delle sottospecie sono ampiamente utilizzate nelle piante, con differenze geografiche ed ecologiche il mezzo principale con cui vengono distinte (Hamilton e Reichard, 1992). Nella misura in cui le sottospecie manifestano un adattamento locale, con o senza flusso genico in corso, suggeriamo che le sottospecie definite fenotipicamente debbano continuare ad essere riconosciute nella conservazione e non confuse con ESU, almeno non ESU come definito per rappresentare popolazioni storicamente isolate.
Allo stesso modo in cui la genomica della popolazione fornisce una precisione molto maggiore nell’identificazione di ESU utilizzando loci neutri e adattivi, fornisce anche un mezzo per utilizzare un gran numero di loci neutri per delimitare più facilmente le unità di gestione (MUS) come unità demograficamente indipendenti caratterizzate da un flusso genico limitato (Funk et al., 2012). Mentre la maggior parte della nostra attenzione qui è sulle unità ESU e analoghe come DUS che possono essere riconosciute come le principali unità intra-specifiche per la conservazione e sono state accettate per la protezione in alcune giurisdizioni, le MUS sono state anche identificate rappresentando gruppi di popolazioni demograficamente indipendenti (vedi Palsbøll et al., 2007) che può essere gestito per mantenere l’ESU più grande (Moritz, 1994; Funk et al., 2012). Le unità di gestione possono essere importanti nel monitoraggio e nella gestione delle popolazioni di specie e sono state anche denominate “stock”, particolarmente rilevanti per la gestione della pesca (Avise, 2004; Laikre et al., 2005). La definizione di MUs di Moritz (1994) come “popolazioni con significativa divergenza di frequenze alleliche a loci nucleari o mitocondriali, indipendentemente dal carattere distintivo filogenetico degli alleli” è generalmente ampiamente accettata, anche se, come sottolineato da Palsbøll et al. (2007), la connettività demografica è il fattore chiave non il livello del flusso genico storico.
Delimitare le unità di conservazione come elementi importanti della diversità intraspecifica ha anche implicazioni significative per il salvataggio genetico di piccole popolazioni inbred attraverso il ripristino del flusso genico o l’aumento e sta diventando sempre più chiaro che il salvataggio genetico deve essere considerato in modo più ampio se si vuole evitare l’estinzione della popolazione aumentata (Love Stowell et al., 2017; Ralls et al., 2018). È stato proposto da Moritz (1999) che mescolare MUS ma non ESUS potrebbe essere considerato una strategia appropriata per il salvataggio genetico. Come sottolineato da Frankham et al. (2012) la miscelazione di specie tassonomicamente riconosciute non sarebbe generalmente considerata accettabile dai manager, nel qual caso l’oversplitting tassonomico potrebbe limitare le opportunità di salvare piccole popolazioni inbred. Suggeriamo che mentre un approccio più rigoroso per delimitare le unità intraspecifiche come MUS potrebbe aiutare ad alleviare questo problema, è anche chiaro che la stima genomica dei tassi di migrazione e della divergenza adattativa ha il potenziale per fornire una guida molto più solida non solo nel definire i confini delle specie e la variazione intraspecifica, ma nella progettazione di strategie di salvataggio genetico.
Filogenomica e conservazione
La genomica combinata con metodi filogenetici può facilmente risolvere le relazioni e la distanza evolutiva tra i taxa, oltre a delimitare le specie. Ciò è meglio ottenuto utilizzando metodi di albero di specie, piuttosto che concatenazione, come il primo conto per l’eterogeneità albero gene ed evitare l’inflazione eccessiva di lunghezze di punta a causa di polimorfismo ancestrale (Edwards et al., 2016; Ogilvie et al., 2016). Mentre un numero massiccio di loci spesso non è essenziale, tra cui più loci spesso migliora la risoluzione di entrambe le relazioni e le lunghezze dei rami (ad esempio, Blom et al., 2016). L’importanza per la conservazione è duplice. In primo luogo, le valutazioni filogenetiche consentono di considerare il carattere distintivo delle specie o degli ESU, non solo se sono descritti. Quindi, il programma EDGE (Isaac et al., 2007) sostiene la ponderazione delle specie per la conservazione in base alla quantità di storia evolutiva unica che rappresentano, oltre ai soliti criteri IUCN. In secondo luogo, le lunghezze dei rami stessi possono rappresentare valori di biodiversità, indipendentemente dalla tassonomia (Faith, 1992), che ha dimostrato valore nella priorità delle aree per la conservazione (Laity et al., 2015; Rosauer et al., 2016, 2018). In questo senso, l’adozione di metriche filogenetiche che utilizzano misure come la diversità filogenetica e l’endemismo filogenetico consentono di evidenziare aree di elevata diversità evolutiva che possono essere mirate alla protezione e ricevere un’alta priorità in qualsiasi processo di pianificazione per una migliore conservazione della biodiversità. Questo approccio può migliorare la protezione della diversità genetica e dei processi evolutivi per diversi gruppi di organismi su una gamma di scale geografiche (Laici et al., 2015) prendere decisioni sulle aree per dare la priorità più robusta ai capricci di come le prove genomiche vengono tradotte in riconoscimento di specie o unità intra specifiche.
Il mondo reale—Definizioni legislative
Le politiche e i quadri legislativi per la conservazione della biodiversità vanno dalle convenzioni e strategie internazionali, passando per strategie e legislazione nazionali, fino alla legislazione e alle strategie statali o regionali all’interno dei paesi. I principali strumenti internazionali biologici di conservazione comprendono la Convenzione sulla Diversità Biologica, attuato attraverso la Convenzione delle Parti (COP), la Convenzione del Patrimonio Mondiale, Globale Strategia per la Biodiversità, a Montreal il Processo per la Conservazione delle Foreste, la Convenzione di Ramsar sulle zone Umide, la Convenzione sulle Specie Migratorie, la Convenzione sul Commercio Internazionale delle Specie Minacciate di estinzione (CITES), l’Unione Internazionale per la Conservazione della Natura (IUCN) lista rossa sulle specie minacciate, e la convenzione Quadro delle Nazioni Unite sui cambiamenti climatici. Tutte queste carte incarnano il principio della conservazione delle specie e della diversità genetica come obiettivo fondamentale della conservazione della biodiversità, con le specie che sono l’unità ampiamente definita per la protezione. Tuttavia, le definizioni di specie previste dalla legislazione e dalle direttive internazionali sono estremamente variabili. Incorporano il riconoscimento tassonomico di specie e taxa intraspecifici, ma possono anche incorporare concetti genetici / evolutivi che sono spesso coperti dalla definizione legale di una” specie ” (Tabella 1).

Tabella 1. Legislazione mondiale e direttive che definiscono “specie” per l’elenco e la protezione.
Data l’ampia gamma di livelli infra-specifici che possono essere protetti (Tabella 1), la legislazione e i relativi processi di inserimento nell’elenco dei paesi dovrebbero facilitare la conservazione della diversità genetica. Ad esempio, la definizione di “specie”, se si considera la legislazione in tutto il mondo (Tabella 1), può includere: specie, sottospecie, varietà e popolazioni geograficamente e / o geneticamente distinte, mentre secondo la legislazione sudafricana può includere anche cultivar, razze geografiche, ceppi e ibridi. Pertanto, secondo la maggior parte della legislazione globale, una” specie ” include entità che riflettono vari livelli infra-specifici che fanno tutti parte di una più ampia distribuzione delle specie e riflettono in varia misura il continuum popolazione-specie. Ciò è parallelo all’inevitabile difficoltà che i tassonomisti, i biologi evoluzionisti e i genetisti della conservazione devono affrontare nel trovare un modo più coerente e uniforme per specificare le unità all’interno di questo continuum.
Alcuni sostengono che le preoccupazioni riguardanti le implicazioni politiche delle specie e l’incertezza infra-specifica sono fuori luogo (Hey et al., 2003), ma siamo ancora del parere che mentre la legislazione può fornire un meccanismo per proteggere la diversità genetica a una serie di livelli, l’onere è sui biologi di fornire maggiore chiarezza e coerenza su come tali livelli dovrebbero essere delimitati dalle specie fino alle singole popolazioni. Attualmente i manager e i legislatori sono lasciati con una serie sconcertante di concetti di specie e unità infra-specifiche fornite dagli scienziati quando delimitano le unità attraverso il continuum popolazione-specie. Ad esempio, tutte le giurisdizioni considerano le specie riconosciute dai tassonomisti, come unità chiave per la conservazione (Tabella 1), tuttavia c’è ancora un ampio dibattito tra i tassonomisti sui concetti di specie e su come le specie vengono diagnosticate nella pratica varia all’interno e tra i gruppi. Ciò ha portato a una recente richiesta di un approccio più regolamentato alla tassonomia (Garnett e Christidis, 2017), a cui altri hanno contrastato che la regolamentazione della tassonomia non è la risposta (Thomson et al., 2018). Anche se siamo d’accordo con alcuni dei punti fatti da Thomson et al. (2018) suggeriamo che sia piuttosto ingenuo proporre che la legislazione stessa debba cambiare per fare riferimento esplicitamente al concetto specifico di taxon implicito da un nome. Questi e precedenti dibattiti di questo tipo evidenziano la necessità per i biologi evoluzionisti e i tassonomisti di prendere l’iniziativa nel garantire chiarezza (trasparenza) nel modo in cui traducono prove genomiche e di altro tipo alla delimitazione di specie o unità intraspecifiche e in che modo questi taxa si riferiscono alla legislazione pertinente per la conservazione e la protezione.
Traduzione in gestione
Come discusso sopra, tutte le normative e le direttive riconoscono il livello di specie come unità fondamentale di conservazione, ma prevedono anche il riconoscimento di varie unità infra-specifiche. Molti hanno anche un requisito implicito per la denominazione formale e la descrizione tassonomica delle specie se considerati per la conservazione priorità e protezione formale. Ad esempio, secondo le linee guida Red Listing IUCN l’elenco delle specie senza nome è scoraggiato e c’è un requisito che la nuova descrizione delle specie sia pubblicata entro quattro anni dall’inclusione della specie nella lista rossa IUCN o sarebbe stata rimossa. Ciò evidenzia l’importanza della denominazione tassonomica e della descrizione delle entità, sia a livello di specie che a livello infra-specifico, le limitate informazioni tassonomiche per molti gruppi di piante e animali (Mace, 2004; Oliver e Lee, 2010; Wilson, 2017) e la necessità di accelerare lo sforzo tassonomico, in particolare nelle aree ricche di biodiversità (Riedel et al., 2013; Wege et al., 2015; Wilson, 2017).
Le specie tassonomicamente denominate e le categorie tassonomiche intraspecifiche, come sottospecie e varietà, forniscono generalmente le unità di base per l’elenco formale, la priorità di conservazione e la protezione, ma in alcune giurisdizioni altre categorie di unità di conservazione intraspecifiche sono formalmente riconosciute. Questi sono generalmente identificati utilizzando dati genomici e fenotipici combinati. Ad esempio, lo USA Endangered Species Act riconosce segmenti di popolazione distinti (DPSS) che sono ampiamente basati su ESU (Waples, 1991), mentre il Canadian Species at Risk Act riconosce DUS. Un’interpretazione operativa di “popolazioni distinte” ai sensi del Canadian Species at Risk Act, DUs mira a identificare insiemi discreti di popolazioni che rappresentano componenti importanti dell’eredità evolutiva delle specie e che difficilmente possono essere sostituiti attraverso la dispersione naturale (Mee et al., 2015).
Sia DUs che DPSs si sovrappongono concettualmente con ESU (Waples et al., 2013) e sono stati applicati più estesamente ai pesci nordamericani, compresi i salmonidi nord-occidentali degli Stati Uniti (Waples, 1995) e i coregonidi canadesi (Mee et al., 2015) È interessante notare che il DPSS negli Stati Uniti si applica solo ai vertebrati mentre in Canada il DUS può essere applicato a qualsiasi organismo che può essere elencato sotto il suo Atto (vedi Waples et al., 2013), con DUS attualmente elencati per artropodi, molluschi e piante. In Australia, come in Canada, popolazioni distinte possono essere elencate per qualsiasi organismo riconosciuto ai sensi dell’Environment Protection and Biodiversity Conservation Act 1999 (EPBC Act) anche se non sono una categoria specificamente nominata, come DUSs e DPSs. Tuttavia, ad oggi sono stati riconosciuti solo per i vertebrati. Negli Stati Uniti ci sono 127 DPSS vertebrati elencati per la conservazione (https://ecos.fws.gov/ecp/); in Canada 70 DU sono stati elencati per tutti gli organismi (http://www.registrelep-sararegistry.gc.ca/species/schedules_e.cfm?id=1); e in Australia le popolazioni di 7 specie di vertebrati che coprono mammiferi, pesci e uccelli sono state riconosciute come aventi uno stato di conservazione specifico ai sensi dell’EPBC Act (http://www.environment.gov.au/cgi-bin/sprat/public/publicthreatenedlist.pl). Mentre le valutazioni di queste popolazioni negli Stati Uniti e in Canada generalmente utilizzano informazioni genetiche (Waples et al., 2013), in Australia sono di solito basati sull’identificazione geografica e giurisdizionale, anche se in alcuni di questi casi sono stati supportati dall’analisi della differenziazione genetica per i mammiferi (Firestone et al., 1999; Armstrong, 2006) e fish (Colgan e Paxton, 1997; Ahonen et al., 2009).
L’elenco delle unità di conservazione infra-specifiche per piante e invertebrati in Canada (DUs) ma non negli Stati Uniti o in Australia evidenzia un altro problema non specificamente correlato alla delimitazione delle unità di conservazione ma più in generale a significative incongruenze nella priorità di conservazione della biodiversità; vertebrati iconici come uccelli, mammiferi e pesci sono al centro di molte giurisdizioni mentre altri organismi non lo sono. Questo elenco preferenziale delle popolazioni di determinati organismi riflette una serie di questioni che hanno meno a che fare con la scienza della delimitazione delle unità di conservazione e più a che fare con l’elenco delle popolazioni basato sulla consapevolezza pubblica, sull’opportunità politica e sulla natura carismatica dell’organismo da elencare (vedi Waples et al., 2013).
Mentre in Australia alcune popolazioni di noto mammiferi, uccelli e pesci sono protetti ai sensi delle EPBC Atto, ora c’è una notevole quantità di informazioni basate su studi molecolari in rettili, invertebrati e piante dove la popolazione può essere riconosciuto come Esù o MUs, e molti di questi sono di conservazione significato data la loro stretta gamma geografica, le piccole dimensioni della popolazione, tasso di declino, e la predisposizione a mettere in pericolo i processi. Ad esempio, esempi eclatanti di popolazioni altamente strutturate e lignaggi profondamente divergenti all’interno di specie che si adattano facilmente al requisito di una ESU possono essere trovati in una gamma di vertebrati a bassa dispersione dai tropici (Figura 2) e altrove. Allo stesso modo negli invertebrati australiani, come anfipodi e millepiedi, una struttura genetica significativa e modelli filogeografici supportano la delimitazione di gruppi di popolazione discreti come ESU geograficamente disparati, con intervalli geografici ristretti, limitati a habitat specifici e rappresentati da solo poche popolazioni (Finston et al., 2007; Murphy et al., 2013; Nistelberger et al., 2014a). Spesso, questi endemici a corto raggio sono taxa di preoccupazione per la conservazione (Harvey et al., 2011) e sono co-localizzati in hotspot endemismo (ad esempio, Oliver et al., 2017), che sono a loro volta alte priorità per l’attenzione alla conservazione multi-taxon (Rosauer et al., 2018), come la regione di Kimberley e la Stirling Range nel sud ovest dell’Australia occidentale.
piante Australiane, ci sono anche un numero significativo di genetica di popolazione e phylogeographic studi individuazione di distinti gruppi di popolazione all’interno di una gamma di specie di generi diversi, che riflettono disgiunti e storicamente popolazione isolata sistemi geologici e edafiche complessità, e situato a contrasto habitat in termini di vegetazione e clima (vedi Broadhurst et al., 2017). Popolazioni geneticamente distinte sono particolarmente evidenti nelle specie del sud-ovest dell’Australia occidentale (Coates, 2000; Byrne et al., 2014) con una serie di studi che evidenziano una struttura genetica significativa e lignaggi altamente divergenti in una gamma di generi e famiglie di piante tra cui: Acacia (Mimosaceae; Coates, 1988), Atriplex (Amaranthaceae; Shepherd et al., 2015), Banksia (Proteaceae; Coates et al., 2015), Lambertia (Proteaceae; Byrne et al., 2002), Hakea (Proteaceae; Sampson et al., 2014), Eremophila (Scrophulariaceae; Llorens et al., 2015), Pultanaea (Fabaceae; Millar e Byrne, 2013), e Calothamnus (Myrtaceae; Nistelberger et al., 2014b). In molti casi queste popolazioni sono riconosciute nella pianificazione della conservazione dalle agenzie governative statali e prese in considerazione nelle valutazioni di impatto ambientale (vedi Shepherd et al., 2015) anche se non sono elencati per la protezione ai sensi della legislazione. Tuttavia, riconosciamo che il riconoscimento formale di queste unità migliorerebbe la loro conservazione a lungo termine.
Questi esempi australiani evidenziano la crescente necessità di riconoscere questo strato di diversità genetica al di sotto del livello di specie. La sfida consiste nel garantire che popolazioni altamente strutturate e lignaggi profondamente divergenti che sono di interesse per la conservazione siano protetti di fronte a richieste concorrenti di attenzione alla conservazione. Al di là delle specie iconiche, questa sfida è evidente anche a livello globale con l’eccezione del Canada. La mancata protezione di unità di conservazione chiaramente delineate aumenterà probabilmente il rischio di estinzione criptica e perdita di una significativa diversità genetica, che in molti casi può coinvolgere lignaggi e popolazioni storicamente isolate che sono persistite all’interno delle specie per milioni di anni (Moritz, 2002).
Conclusioni e raccomandazioni
L’elenco delle specie e di varie categorie infra-specifiche sotto la legislazione sulle specie minacciate o sotto organizzazioni internazionali come la IUCN indica un impegno a proteggere la diversità genetica attraverso il continuum della popolazione di specie. Tuttavia, gli attuali approcci alla conservazione della biodiversità si concentrano in gran parte sulla fine delle specie di questo continuum con una minore attenzione alla diversità genetica e alla variazione infra-specifica. Questa attenzione più limitata alla variazione genetica a livello di intra-specie suggerisce che mentre i responsabili della conservazione riconoscono sempre più il valore di considerare esplicitamente le informazioni genetiche nelle azioni di gestione e nella definizione delle priorità, spesso mancano di informazioni attuabili dagli scienziati della biodiversità (Waples et al., 2008; Laikre et al., 2010; Cook e Sgrò, 2017; Taylor et al., 2017). Questo divario tra scienza e pratica di conservazione impedisce un approccio globale più efficace alla conservazione e alla gestione della diversità genetica.
Suggeriamo che la mancanza di chiarezza da parte degli scienziati della biodiversità sulle unità di conservazione si basa in gran parte su incongruenze a due livelli. Il primo riguarda i concetti di specie multiple e i vari metodi diagnostici utilizzati per delimitare le specie che creano incoerenza tra i gruppi tassonomici. Il secondo riguarda la mancanza di coerenza e accordo da parte di tassonomisti, biologi evoluzionisti e biologi della conservazione su come trattare i taxa infra-specifici e altre entità come ESU, DUS e MUS.
Mentre riteniamo che la legislazione sia spesso già sufficientemente flessibile in termini di come vengono definiti i taxa (o “specie”), sosteniamo che sia necessario affrontare sistematicamente il continuum delle specie di popolazione e definire più chiaramente le unità per la conservazione con un maggiore riconoscimento dato a concetti come ESU e come questi si riferiscono alla legislazione pertinente. Sottolineiamo che questo problema diventerà senza dubbio più critico dato il potere della genomica di rilevare la differenziazione a scale progressivamente più fini e il suo crescente uso per delimitare specie ed entità infra-specifiche.
Dall’analisi precedente, offriamo le seguenti raccomandazioni:
(i) I dati genomici dovrebbero essere interpretati in modo conservativo quando si delimitano le specie, utilizzando un approccio tassonomico integrativo che in genere include prove al di là dei soli dati genetici.
(ii) in Cui i genetisti identificare lignaggi più o distinte popolazioni all’interno di una specie, e questi non garantisce l’elevazione a livello di specie e non sono tassonomicamente riconosciuto che non ci sia più forte impulso e un approccio più uniforme per identificare chiaramente giustificato intraspecifica unità per una concreta valutazione dello stato di conservazione o di valutazioni d’impatto, e che queste siano esplicitamente riconosciuto come unità di misura per la conservazione nel contesto della legislazione pertinente a tale giurisdizione.
(iii) La IUCN, attraverso il suo Conservation Genetics Specialist Group e i suoi sottogruppi regionali, ha preso in considerazione lo sviluppo di linee guida per il riconoscimento e l’etichettatura di unità intraspecifiche allineate alle migliori pratiche scientifiche.
(iv) La visione più sfumata che emerge dalla genomica evolutiva delle specie e dal continuum dalle popolazioni alle specie dovrebbe essere comunicata in modo più efficace dagli scienziati ai responsabili della conservazione e ai responsabili politici.
Più in generale facciamo eco alle raccomandazioni fatte di recente in una serie di recensioni (Frankham, 2010; Santamaría e Méndez, 2012; Hoban et al., 2013; Moritz e Potter, 2013; Laici et al., 2015; Pierson et al., 2016; Cook e Sgrò, 2017; Taylor et al., 2017) che evidenziano la necessità per gli scienziati di migliorare la comunicazione e aumentare la consapevolezza con i responsabili politici, le agenzie governative, le organizzazioni non governative e i professionisti che i risultati della sistematica molecolare, della tassonomia molecolare, della filogeografia e della ricerca genetica della popolazione possono fornire informazioni critiche sulla conservazione della diversità genetica e sulla gestione dei processi evolutivi.
Contributi dell’autore
DC, MB e CM hanno contribuito alla preparazione e alla revisione del manoscritto.
Dichiarazione sul conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Riconoscimenti
Questo progetto è stato sostenuto dall’Australian Research Council (ARC Linkage Grant n. LP120200063), l’Australian National University e il Western Australian Department of Biodiversity, Conservation, and Attractions.
Afonso Silva, A. C., Bragg, J. G., Potter, S., Fernandes, C., Coelho, M. M., e Moritz, C. (2017a). Tropical specialist versus climate generalist: diversificazione e storia demografica delle specie sorelle di Carlia skinks dall’Australia nord-occidentale. Mol. Ecol. 26:4045–4058. doi: 10.1111 / mec.14185
CrossRef Full Text/Google Scholar
Afonso Silva, A. C., Santos, N., Ogilvie, H. A., e Moritz, C. (2017b). Validazione e descrizione di due nuovi scinchi Arcobaleno dell’Australia nord-occidentale con metodi e morfologia coalescenti multispecie. PeerJ 5: e3724. doi: 10.7717 / peerj.3724
PubMed Abstract / CrossRef Full Text/Google Scholar
Ahonen, H., Harcourt, R. G., e Stow, A. J. (2009). Il DNA nucleare e mitocondriale rivela l’isolamento di popolazioni di squali nutrici grigi in pericolo (Carcharias taurus). Mol. Ecol. 18, 4409–4421. doi: 10.1111/j.1365-294X. 2009. 04377.x
PubMed Abstract | CrossRef Full Text/Google Scholar
Armstrong, KN (2006). Struttura filogeografica in Rhinonicteris aurantia (Chiroptera: Hipposideridae): implicazioni per la conservazione. Acta Chiropterologica 8, 63-81. doi: 10.3161/1733-5329(2006)82.0.COil sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione., e Kunte, K. (2017). Scambio genetico adattivo: una storia aggrovigliata di mescolanza e innovazione evolutiva. Tendenze Ecol. Evol. 32, 601–611. doi: 10.1016 / j.albero.2017.05.007
PubMed Abstract | CrossRef Full Text/Google Scholar
Avise, JC (2004). Marcatori molecolari, Storia naturale ed evoluzione. 2 ° Edn. Sunderland, MA; Oxford: Oxford University Press.il nostro sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. (1987). Filogeografia intraspecifica: il ponte del DNA mitocondriale tra genetica di popolazione e sistematica. Ann. Rev. Ecol. Sys. 18, 489–522. doi: 10.1146/annurev.es.18.110187.002421
CrossRef Full Text/Google Scholar
Bickford, D., Lohman, DJ, Sodhi, NS, Ng, PK, Meier, R., Winker, K., et al. (2007). Specie criptiche come finestra sulla diversità e la conservazione. Tendenze Ecol. Evol. 22, 148–155. doi: 10.1016 / j.albero.2006.11.004
PubMed Abstract | CrossRef Full Text/Google Scholar
Blom, MPK, Bragg, JG, Potter, S. e Moritz, C. (2016). Contabilizzazione dell’incertezza nella stima dell’albero genetico: riassunto – inferenza dell’albero delle specie coalescenti in una radiazione impegnativa di lucertole australiane. Syst. Biol. 66, 352–366. doi: 10.1093/sysbio/syw089
PubMed Abstract / CrossRef Full Text/Google Scholar
Broadhurst, L., Breed, M., Lowe, A., Bragg, J., Catullo, R., Coates, DJ, et al. (2017). Diversità genetica e struttura della flora australiana. Divers. Distrib. 23, 41–52. doi: 10.1111/ddi.12505
CrossRef Full Text/Google Scholar
Byrne, M., Coates, D. J., Forest, F., Hopper, S. D., Krauss, S. L., Sniderman, J. M. K., et al. (2014). “A diverse flora-specie e relazioni genetiche”, in Plant Life on the Sandplains in Southwest Australia, a Global Biodiversity Hotspot, editors H. Lambers (Crawley, WA: University of Western Australia Publishing), 81-99.
PubMed Abstract
Byrne, M., Macdonald, B., e Coates, DJ (2002). Le piante di Acacia acuminata (Leguminosae:Mimosoideae) sono un gruppo di piante spermatofite dicotiledoni appartenenti alla famiglia delle Mimosoideae. complesso in Australia occidentale. J. Evol. Biol. 15, 576–587. doi: 10.1046 / j. 1420-9101. 2002.00429.il sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. Come fallire nella delimitazione delle specie. Mol. Ecol. 22, 4369–4383. doi: 10.1111 / mec.12413
PubMed Abstract | CrossRef Full Text/Google Scholar
Coates, D. J. (1988). Diversità genetica e struttura genica della popolazione nel raro Chittering grass wattle, Acacia anomala Court. Aust. J. Bot. 36:273–286. doi: 10.1071/BT9880273
CrossRef Full Text/Google Scholar
Coates, DJ (2000). Definizione di unità di conservazione in una flora ricca e frammentata: implicazioni per la gestione delle risorse genetiche e dei processi evolutivi nelle piante del sud-ovest dell’Australia. Aust. J. Bot. 48, 329–339. doi: 10.1071/BT99018
CrossRef Full Text/Google Scholar
Coates, DJ, McArthur, SL, e Byrne, M. (2015). Significativa perdita di diversità genetica in seguito all’estinzione della popolazione causata da patogeni nella rara endemica Banksia brownii (Proteaceae). Biol. Conserv. 192, 353–360. doi: 10.1016 / j.biocon.2015.10.013
CrossRef Full Text/Google Scholar
Colgan, D. J., e Paxton, J. R. (1997). Genetica biochimica e riconoscimento di uno stock occidentale del pesce gemma comune, Rexea solandri (Scombroidea: Gempylidae)., in Australia. Guastare. Freshw. Res. 48, 103-118. doi: 10.1071/MF96048
CrossRef Full Text/Google Scholar
Convention on Biological Diversity (2007). È disponibile online all’indirizzo:https://www.cbd.int/doc/meetings/cop-bureau/cop-bur-2007/cop-bur-2007-10-14-en.pdf
Cook, C. N., e Sgrò, C. M. (2017). Allineare scienza e politica per ottenere una conservazione evolutivamente illuminata. Conserv. Biol. 31, 501–512. doi: 10.1111 / cobi.12863
PubMed Abstract | CrossRef Full Text/Google Scholar
Cook, LG, Edwards, RD, Crisp, MD e Hardy, NB (2010). Hai bisogno di morfologia sempre essere richiesto per nuove descrizioni di specie? Invertire. Sys. 24, 322–332. doi: 10.1071/IS10011
CrossRef Full Text / Google Scholar
Cracraft, J. (1983). Concetti di specie e analisi di speciazione. Curr. Ornitolo. 1, 159–187. doi: 10.1007/978-1-4615-6781-3_6il sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. Considerando i processi evolutivi nella biologia della conservazione. Tendenze Ecol. Evol. 15, 290–295. doi: 10.1016/S0169-5347(00)01876-0
PubMed Abstract | CrossRef Full Text/Google Scholar
Cutter, A. D., and Payseur, B. A. (2013). Firme genomiche di selezione nei siti collegati: unificare la disparità tra le specie. NAT. Rev. Genet. 14, 262–274. doi: 10.1038/nrg3425
PubMed Abstract | CrossRef Full Text/Google Scholar
Dayrat, B. (2005). Verso la tassonomia integrativa. Biol. J. Linn. Soc. 85, 407–415. doi: 10.1111 / j. 1095-8312. 2005.00503.x5
CrossRef Full Text/Google Scholar
de Queiroz, K. (1998). ” The general lineage concept of species, species criteria, and the process of speciation, ” in Forms: Species and Speciation, D. J. Howard, r S. H. Berlocher (New York, NY: Oxford University Press), 57-75.
Google Scholar
de Queiroz, K. (2007). Concetti di specie e delimitazione di specie. Syst. Biol. 56, 879–886. doi: 10.1080 | 10635150701701083
PubMed Abstract / CrossRef Full Text / Google Scholar
Degnan, JH, and Rosenberg, N. A. (2009). Discordanza degli alberi genetici, inferenza filogenetica e coalescenza multispecie. Tendenze Ecol. Evol. 24, 332–340. doi: 10.1016 / j.albero.2009.01.009
PubMed Abstract | CrossRef Full Text/Google Scholar
Devey, DS, Bateman, R. M., Fay, M. F., e Hawkins, J. A. (2008). Amici o parenti? Filogenetica e delimitazione delle specie nel controverso genere europeo di orchidee Ophrys. Ann. Bot. 101, 385–402. doi: 10.1093/aob | mcm299
PubMed Abstract/CrossRef Full Text/Google Scholar
Dizon, A. E., Lockyer, C, Perrin, W. F., Demaster, D. P., e Sisson, J. (1992). Ripensare il concetto di stock-un approccio filogeografico. Conserv. Biol. 6, 24–36. doi: 10.1046 / j. 1523-1739. 1992. 610024.x
CrossRef Full Text/Google Scholar
Doughty, P., Bourke, G., Tedeschi, L., Pratt, R., Oliver, P., Palmer, R., et al. (2018). Delimitazione delle specie nella Gehyra nana (Squamata: Gekkonidae). complesso: evoluzione morfologica criptica e divergente nei Tropici monsonici australiani, con la descrizione di quattro nuove specie. Zootaxa, 4403: 201-244. doi: 10.11646 / zootaxa.4403.2.1
PubMed Abstract | CrossRef Full Text/Google Scholar
Dynesius, M., and Jansson, R. (2014). Persistenza dei lignaggi all’interno delle specie: un controllo trascurato dei tassi di speciazione. Evoluzione 68, 923-934. doi: 10.1111 / evo.12316
PubMed Abstract / CrossRef Full Text / Google Scholar
Edwards, S. V., Potter, S., Schmitt, C. J., Bragg, J. G., e Moritz, C. (2016). Reticulation, divergence, and the phylogeography-phylogenetics continuumc. Proc. Natl. Acad. Sic. U. S. A. 113, 8025-8032. doi: 10.1073 / pnas.1601066113
CrossRef Full Text/Google Scholar
Faith, D. P. (1992). Valutazione della conservazione e diversità filogenetica. Biol. Svantaggio. 61, 1–10. doi: 10.1016/0006-3207(92)91201-3il sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. La speciazione criptica in due generi di anfipodi sotterranei diffusi riflette i modelli di drenaggio storici in un paesaggio antico. Mol. Ecol. 16, 355–365. doi: 10.1111/j.1365-294X. 2006. 03123.per questo motivo, il progetto è stato pubblicato nel 1999, con l’aggiunta di una versione aggiornata del testo originale. La struttura filogeografica della popolazione di Dasyurus maculatus (Dasyuridae: Marsupalia), un marsupiale carnivoro in via di estinzione. Mol. Ecol. 8, 1613–1625. doi: 10.1046 / j. 1365-294x. 1999. 00745.il sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. Specie criptiche come finestra sul cambiamento di paradigma del concetto di specie. Mol. Ecol. 27, 613–635. doi: 10.1111 / mec.14486
PubMed Abstract | CrossRef Full Text/Google Scholar
Frankham, R. (2010). Sfide e opportunità degli approcci genetici alla conservazione biologica. Biol. Conserv. 143,1919–1927. doi: 10.1016 / j.biocon.2010.05.011
CrossRef Full Text/Google Scholar
Frankham, R., Ballou, JD, Dudash, M. R., Eldridge, MD, Fenster, CB, Lacy, RC, et al. (2012). Implicazioni dei diversi concetti di specie per la conservazione della biodiversità. Biol. Conserv. 153, 25–31. doi: 10.1016 / j.biocon.2012.04.034
CrossRef Full Text/Google Scholar
Fraser, DJ, e Bernatchez, L. (2001). Conservazione evolutiva adattiva: verso un concetto unificato per la definizione delle unità di conservazione. Mol. Ecol. 10, 2741–2752. doi: 10.1046 / j. 1365-294X. 2001.t01-1-01411.il sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione., e Allendorf, F. W. (2012). Sfruttare la genomica per delineare le unità di conservazione. Tendenze Ecol. Evol. 27, 489–496. doi: 10.1016 / j.albero.2012.05.012
PubMed Abstract | CrossRef Full Text/Google Scholar
Garnett, S. T., and Christidis, L. (2017). Tassonomia anarchia ostacola la conservazione. Natura 546, 25-27. doi: 10.1038/546025a
PubMed Abstract | CrossRef Full Text/Google Scholar
Gippoliti, S., Cotterill, F. P. D., Zinner, D., and Groves, CP (2018). Impatti dell’inerzia tassonomica per la conservazione della diversità degli ungulati africani: panoramica. Biol. Apoc 93, 115-130. doi: 10.1111 / brv.12335
PubMed Abstract | CrossRef Full Text/Google Scholar
Grant, V. (1981). Speciazione vegetale. New York, NY: Columbia University Press.
PubMed Abstract/Google Scholar
Hamilton, C. W., e Reichard, S. H. (1992). Pratica corrente nell’uso di sottospecie, varietà e forma nella classificazione delle piante selvatiche. Taxon 41, 485-498. il nostro sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione., Johnson, MS, Teale, RJ, et al. (2011). Proteggere gli innocenti: studiare taxa endemici a corto raggio migliora i risultati di conservazione. Invertire. Syst. 25, 1–10. il nostro sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione e per migliorare la tua esperienza di navigazione. Comprendere e affrontare l’incertezza delle specie in biologia e conservazione. Tendenze Ecol. Evol. 18, 597–603. doi: 10.1016 / j.albero.2003.08.014
CrossRef Full Text/Google Scholar
Hoban, S. M., Hauffe, H. C., Perez-Espona, S., Arntzen, J. W., Bertorelle, G., Bryja, J., et al. (2013). Portare la diversità genetica in prima linea nella politica e nella gestione della conservazione. Conserv. Genet. Resour. 5, 593–598. doi: 10.1007 / s12686-013-9859-y
CrossRef Full Text/Google Scholar
Isaac, NJ, Mallet, J., and Mace, GM (2004). Inflazione tassonomica: la sua influenza sulla macroecologia e la conservazione. Tendenze Ecol. Evol. 19, 464–469. doi: 10.1016 / j.albero.2004.06.004
PubMed Abstract | CrossRef Full Text/Google Scholar
Isaac, NJ, Turvey, S. T., Collen, B., Waterman, C., e Baillie, J. E. (2007). Mammiferi sul BORDO: priorità di conservazione basate su minaccia e filogenesi. PLoS UNO 2:e296. doi: 10.1371 / giornale.pone.0000296
PubMed Abstract | CrossRef Full Text/Google Scholar
Jones, M. R., and Good, JM (2016). Cattura mirata nella genomica evolutiva ed ecologica. Mol. Ecol. 25, 185–202. doi: 10.1111 / mec.13304
PubMed Abstract | CrossRef Full Text/Google Scholar
Jorger, KM, and Schrödl, M. (2013). Come descrivere una specie criptica? Sfide pratiche della tassonomia molecolare. Anteriore. Zool. 10:59. doi: 10.1186/1742-9994-10-59
PubMed Abstract / CrossRef Full Text/Google Scholar
Laikre, L., Allendorf, F., Aroner, L. C., Baker, S., Gregovich, D. P., Hansen, M. M., et al. (2010). Trascuratezza della diversità genetica nell’attuazione della convenzione sulla diversità biologica. Conserv. Biol. 24, 86–88. doi: 10.1111 / j. 1523-1739. 2009.01425.il sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. Struttura genetica della popolazione dei pesci: implicazioni per la gestione delle zone costiere. Ambio 34.111–119. doi: 10.1579/0044-7447-34.2.111
PubMed Abstract | CrossRef Full Text/Google Scholar
Laici, T., Laffan, SW, González-Orozco, CE, Faith, D. P., Rosauer, D. F., Byrne, M., et al. (2015). Phylodiversity per informare la politica di conservazione: un esempio australiano. Sic. Ambiente totale. 534, 131–143. doi: 10.1016 / j. scitotenv.2015.04.113
PubMed Abstract | CrossRef Full Text/Google Scholar
Leache, A. D., and Oaks, J. R. (2017). L’utilità del polimorfismo a singolo nucleotide (SNP). dati in filogenetica. Ann. Rev. Ecol. Syst. 48, 69–84. doi: 10.1146/annurev-ecolsys-110316-022645
CrossRef Full Text | Google Scholar
Llorens, T. M., Macdonald, B., McArthur, S., Coates, D. J., and Byrne, M. (2015). Disjunct, highly divergent genetic lineages within two rare Eremophila (Scrophulariaceae: Myoporeae). species in a biodiversity hotspot: implications for taxonomy and conservation. Biol. J. Linn. Soc. 177, 96–111. doi: 10.1111/boj.12228
CrossRef Full Text | Google Scholar
Love Stowell, S. M., Pinzone, C. A., and Martin, A. P. (2017). Superare le barriere agli interventi attivi per la diversità genetica. Biodivers. Conserv. 26, 1753–1765. doi: 10.1007 / s10531-017-1330-z
CrossRef Full Text/Google Scholar
Mace, GM (2004). Il ruolo della tassonomia nella conservazione delle specie. Philo. Trans. R. Soc. B: Biol. Sic. 359, 711–719. doi: 10.1098 / rstb.2003.1454
PubMed Abstract | CrossRef Full Text/Google Scholar
Mallet, J. (2007). Speciazione ibrida. Natura 446, 279-283. doi: 10.1038/nature05706
PubMed Abstract | CrossRef Full Text/Google Scholar
Mayr, E. (1942). Sistematica e origine delle specie. New York, NY: Columbia University Press.
Google Scholar
Mee, J. A., Bernatchez, L., Reist, J. D., Rogers, S. M., e Taylor, E. B. (2015). Identificazione di unità designabili per la conservazione intraspecifica priorità: un approccio gerarchico applicato al complesso di specie di coregone lacustre (Coregonus spp.). Evol. Appl. 8, 423–441. doi: 10.1111 / eva.12247
PubMed Abstract | CrossRef Full Text | Google Scholar
Millar, M. A., and Byrne, M. (2013). Cryptic divergent lineages of Pultenaea pauciflora M.B. Scott (Fabaceae: Mirbelieae) exhibit different evolutionary history. Biol. J. Linn. Soc. 108, 871–881. doi: 10.1111/bij.12002
CrossRef Full Text | Google Scholar
Moritz, C. (1994). Defining evolutionarily significant units for conservation. Trends Ecol. Evol. 9, 373–375. doi: 10.1016/0169-5347(94)90057-4
CrossRef Full Text | PubMed Abstract
Moritz, C. (1999). Unità di conservazione e traslocazioni: strategie per la conservazione dei processi evolutivi. Hereditas 130, 217-228. doi: 10.1111 / j. 1601-5223. 1999.00217.x
CrossRef Full Text/Google Scholar
Moritz, C. (2002). Strategie per proteggere la diversità biologica e i processi evolutivi che la sostengono. Syst. Biol. 51, 238–254. doi: 10.1080 | 10635150252899752
PubMed Abstract / CrossRef Full Text/Google Scholar
Moritz, C., and Potter, S. (2013). L’importanza di una prospettiva evolutiva nella pianificazione della politica di conservazione. Mol. Ecol. 22, 5969–5971. doi: 10.1111 / mec.12565
PubMed Abstract / CrossRef Full Text/Google Scholar
Moritz, C., Pratt, R. C., Bank, S., Bourke, G., Bragg, J. G., Doughty, P., et al. (2018). Diversità di lignaggio criptico, divergenza delle dimensioni del corpo e simpatia in un complesso di specie di lucertole australiane (Gehyra). Evoluzione 27, 54-66. doi: 10.1111 / evo.13380
CrossRef Full Text/Google Scholar
Morrison, WR, Lohr, JL, Duchen, P., Wilches, R., Trujillo, D., Mair, M., et al. (2009). L’impatto del cambiamento tassonomico sulla conservazione: Uccide, può salvare o è solo irrilevante? Biol. Svantaggio. 142, 3201–3206. doi: 10.1016 / j.biocon.2009.07.019
CrossRef Full Text/Google Scholar
Murphy, NP, Adams, M., Guzik, MT e Austin, A. D. (2013). Straordinario micro-endemismo negli anfipodi primaverili del deserto australiano. Mol. Filogeneta. Evol. 66, 645–653. doi: 10.1016 / j. ympev.2012.10.013
PubMed Abstract | CrossRef Full Text/Google Scholar
Murphy, NP, King, RA e Delean, S. (2015). Specie, ESU o popolazioni? Delimitando e descrivendo la diversità morfologicamente criptica negli anfipodi primaverili del deserto australiano. Invertire. Syst. 29, 457–467. doi: 10.1071/IS14036
CrossRef Full Text/Google Scholar
Nater, A., Mattle-Greminger, MP, Nurcahyo, A., Nowak, MG, de Manual, M., Desai, T., et al. (2017). Prove morfometriche, comportamentali e genomiche per una nuova specie di orango. Curr. Biol cellulare. 27, 3487–3498. doi: 10.1016 / j.cub.2017.09.047
CrossRef Full Text / Google Scholar
Nicotra, AB, Chong, C., Bragg, JG, Ong, CR, Aitken, NC, Chuah, A., et al. (2016). Popolazione e decomposizione filogenomica tramite genotipizzazione-by-sequenziamento in Pelargonium australiano. Mol. Ecol. 25, 2000–2014. doi: 10.1111 / mec.13584
PubMed Abstract | CrossRef Full Text/Google Scholar
Nistelberger, H., Byrne, M., Coates, D., and Roberts, JD (2014a). Forte struttura filogeografica in un millepiedi indica vicarianza pleistocene tra le popolazioni su formazioni di ferro fasciato in Australia semi-arida. PLoS UNO 9:e0093038. doi: 10.1371 / giornale.pone.0093038
PubMed Abstract | CrossRef Full Text/Google Scholar
Nistelberger, H., Gibson, N., Macdonald, B., Tapper, S.-L., e Byrne, M. (2014b). Prove filogeografiche per due rifugi mesici in un hotspot di biodiversità. Eredità 113, 454-463. doi: 10.1038 / hdy.2014.46
PubMed Abstract | CrossRef Full Text/Google Scholar
Ogilvie, H. A., Heled, J., Xie, D., and Drummond, A. J. (2016). Prestazioni computazionali e accuratezza statistica di beast e confronti con altri metodi. Syst. Biol. 65, 381–396. doi: 10.1093 / sysbio / syv118
PubMed Abstract | CrossRef Full Text/Google Scholar
Oliver, PM, Keogh, JS, and Moritx, C. (2015). Nuovi approcci alla catalogazione e alla comprensione della diversità evolutiva: una prospettiva dall’erpetologia australiana. Aust. J. Zool. 62, 417–430. il sito utilizza cookie tecnici, di terze parti, per migliorare la tua esperienza di navigazione e per migliorare la tua esperienza di navigazione. Un nuovo hotspot di endemismo vertebrato e un refugium evolutivo in Australia tropicale. Subacqueo. Distrib. 23, 53–66. doi: 10.1111/ddi.12506
CrossRef Full Text/Google Scholar
Oliver, PM, and Lee, MSY (2010). I codici botanici e zoologici impediscono la ricerca sulla biodiversità scoraggiando la pubblicazione di nuove specie senza nome. Taxon 59, 1201-1205. doi: 10.2307/20773990
CrossRef Full Text/Google Scholar
Padial, J., Miralles, A., De la Riva, I., and Vences, M. (2010). Il futuro integrativo della tassonomia. Anteriore. Zool. 7:16. doi: 10.1186/1742-9994-7-16
PubMed Abstract | CrossRef Full Text/Google Scholar
Palsbøll, PJ, Bérubé, M., and Allendorf, FW (2007). Identificazione delle unità di gestione utilizzando i dati genetici della popolazione. Tendenze Ecol. Evol. 22, 11–16. doi: 10.1016 / j.albero.2006.09.003
PubMed Abstract | CrossRef Full Text/Google Scholar
Patton, JL, and Smith, MF (1994). Parafilia, polifilia, e la natura dei confini di specie in Gopher tasca (Genere Thomomys). Syst. Biol. 43, 11–26. doi: 10.1093 / sysbio / 43.1.11
CrossRef Full Text/Google Scholar
Pierson, J. C., Coates, D. J., Oostermeijer, J. G. B., Beissinger, SR, Bragg, J. G., Sunnucks, P., et al. (2016). Fattori genetici nei piani di recupero delle specie minacciate in tre continenti. Anteriore. Ecol. Ambiente. 14, 433–440. doi: 10.1002 / tassa.1323
CrossRef Full Text/Google Scholar
Potter, S., Bragg, JG, Peter, BM, Bi, K. e Moritz, C. (2016). Filogenomica alle punte: inferire lignaggi e la loro storia demografica in una lucertola tropicale, Carlia amax. Mol. Ecol. 25, 1367–1380. doi: 10.1111 / mec.13546
PubMed Abstract | CrossRef Full Text/Google Scholar
Potter, S., Xue, A. T., Bragg, J. G., Rosauer, D. F., Roycroft, E. J., and Moritz, C. (2018). Pleistocene cambiamenti climatici guidare la diversificazione attraverso una savana tropicale. Mol. Ecol. 27, 520–532. doi: 10.1111 / mec.14441 / full
PubMed Abstract | CrossRef Full Text/Google Scholar
Rabosky, D. L., Hutchinson, M. N., Donnellan, S. C., Talaba, A. L., e Lovette, I. J. (2014). Disassemblaggio filogenetico dei confini delle specie in un gruppo diffuso di scinchi australiani (Scincidae: Ctenotus). Mol. Filogeneta. Evol. 77, 71–82. doi: 10.1016 / j. ympev.2014.03.026
PubMed Abstract | CrossRef Full Text/Google Scholar
Ralls, K., Ballou, J. D., Dudash, M. R., Eldridge, MDB, Fenster, C. B., Lacy, R. C., et al. (2018). Chiedere un cambiamento di paradigma nella gestione genetica delle popolazioni frammentate. Conserv. Lett. 11, 1–6. doi: 10.1111 / conl.124126
CrossRef Full Text/Google Scholar
Rannala, B. (2015). L’arte e la scienza della delimitazione delle specie. Curr. Zool. 61, 846–853. in questo modo, il sistema di gestione delle risorse idriche e la gestione delle risorse idriche sono in grado di migliorare la qualità delle risorse idriche e di migliorare la qualità delle risorse idriche. Tassonomia integrativa in rapida evoluzione-verso una maggiore sostenibilità nella ricerca sulla biodiversità. Anteriore. Zool. 10:15. doi: 10.1186 | 1742-9994-10-15
PubMed Abstract/CrossRef Full Text/Google Scholar
Rieseberg, L. H., and Willis, JH (2007). Speciazione vegetale. Scienza 317, 910-914. doi: 10.1126 / scienza.1137729
PubMed Abstract | CrossRef Full Text/Google Scholar
Rosauer, D. F., Blom, M. P. K., Bourke, G., Catalano, S., Donnellan, S., Mulder, E., et al. (2016). Filogeografia, hotspot e priorità di conservazione: un esempio dall’estremità superiore dell’Australia. Biol. Svantaggio. 204, 83–93. doi: 10.1016 / j.biocon.2016.002
CrossRef Full Text/Google Scholar
Rosauer, D. F., Byrne, M., Blom, M. P. K., Coates, DJ, Donnellan, S., Doughty, P., et al. (2018). La pianificazione della conservazione del mondo reale per la diversità evolutiva nel Kimberley, in Australia, evita la tassonomia incerta Conserv. Lett. 11: e12438. doi: 10.1111 / conl.12438
CrossRef Full Text/Google Scholar
Rosenblum, E. B., Sarver, B. A. J., Brown, J. W., Des Roches, S., Hardwick, K. M., Hether, T. D., et al. (2012). Riccioli d’oro incontra Santa Rosalia: un modello di speciazione effimera spiega i modelli di diversificazione attraverso scale temporali. Evol. Biol. 39, 255–261. doi: 10.1007 / s11692-012-9171-per questo motivo, il progetto è stato pubblicato nel 2016, in collaborazione con il Dipartimento di Scienze della formazione e della formazione professionale. Facendo luce sulla zona grigia della speciazione lungo un continuum di divergenza genomica. PLoS Biol. 14: e2000234. doi: 10.1371 / giornale.pbio.2000234
PubMed Abstract | CrossRef Full Text/Google Scholar
Ryder, O. A. (1986). Conservazione delle specie e sistematica-il dilemma delle sottospecie. Tendenze Ecol. Evol. 1, 9–10. doi: 10.1016/0169-5347 (86)90059-5
CrossRef Full Text | Google Scholar
Sampson, J. F., Byrne, M., Yates, C. J., Gibson, N., Thavornkanlapachai, R., Stankowski, S., et al. (2014). L’immigrazione genetica contemporanea mediata dal polline riflette l’isolamento storico di un raro arbusto in un paesaggio frammentato. Eredità 112, 172-181. doi: 10.1038 / hdy.2013.89
PubMed Abstract | CrossRef Full Text/Google Scholar
Santamaría, L., and Méndez, P. F. (2012). Evoluzione della politica sulla biodiversità: lacune attuali e bisogni futuri. Evol. Appl. 5, 202–218. doi: 10.1111 / j.1752-4571.2011.00229.per questo motivo, il progetto è stato pubblicato nel 2010 con il titolo di studio: “Il libro è stato pubblicato nel 2010 e pubblicato nel 2010. Tassonomia integrativa: un approccio multisource per esplorare la biodiversità. Annu. Rev. Entomolo. 55, 421–438. doi: 10.1146/annurev-en-112408-085432
PubMed Abstract | CrossRef Full Text/Google Scholar
Seehausen, O, Takimoto, G., Roy, D., e Jokela, J. (2008). Inversione di speciazione e dinamiche di biodiversità con ibridazione in ambienti mutevoli. Mol Ecol. 17, 30–44. doi: 10.1111/j.1365-294X. 2007. 03529.il sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. Una rara e nuova specie di Atriplex (Chenopodiaceae) comprendente due popolazioni geneticamente distinte ma morfologicamente criptiche nell’arida Australia occidentale: implicazioni per la tassonomia e la conservazione. Aust. Syst. Bot. 28, 234–245. doi: 10.1071 / SB15029
CrossRef Full Text/Google Scholar
Singhal, S., and Bi, K. (2017). La storia pulisce i pasticci: l’impatto del tempo nel guidare la divergenza e l’introgressione in una zona di sutura tropicale. Evoluzione 71, 1888-1899. doi: 10.1111 / evo.13278
PubMed Abstract | CrossRef Full Text/Google Scholar
Singhal, S., Hoskin, C., Couper, P., Potter, S., e Moritz, C. (2018). Quadro per la risoluzione delle specie criptiche: un caso di studio dalle lucertole dei tropici umidi australiani. Syst. Biol. . doi: 10.1093 / sysbio / syy
PubMed Abstract | CrossRef Full Text/Google Scholar
Singhal, S., and Moritz, C. (2013). Isolamento riproduttivo tra linee filogeografiche scale con divergenza. Proc. Roy. Soc B 280:20132246. doi: 10.1098 / rspb.2013.2246026
PubMed Abstract | CrossRef Full Text/Google Scholar
Steane, D. A., Potts, B. M., McLean, E., Collins, L., Prober, S. M., Stock, W. D., et al. (2015). Le scansioni a livello del genoma rivelano una struttura di popolazione criptica in un eucalipto adattato a secco. Albero Genet. Genomi 11: 33. doi: 10.1007 / s11295-015-0864-z
CrossRef Full Text/Google Scholar
Struck, T. H., Feder, J. L., Bendiksby, M., Birkeland, S., Cerca, J., Gusarov, V. I., et al. (2017). Trovare processi evolutivi nascosti in specie criptiche. Tendenze Ecol. Evol. 33, 153–163. doi: 10.1016 / j.albero.2017.11.007
PubMed Abstract | CrossRef Full Text/Google Scholar
Sukumaran, J., and Knowles, LL (2017). La multispecie coalescente delimita la struttura non specie. Proc. NAT. Aca. Sic. U. S. A. 114, 1607-1612. doi: 10.1073 / pnas.1607921114
PubMed Abstract | CrossRef Full Text/Google Scholar
Swarts, ND, Clements, MA, Bower, CC e Miller, JT (2014). Definizione di unità di conservazione in un complesso di orchidee morfologicamente simili, sessualmente ingannevoli e altamente minacciate. Biol. Svantaggio. 174, 55–64. doi: 10.1016 / j.biocon.2014.03.017
CrossRef Full Text/Google Scholar
Tautz, D., Arctander, P., Minelli, A., Thomas, R. H. e Vogler, A. P. (2003). Un appello per la tassonomia del DNA. Tendenze Ecol. Evol. 18, 70–74. doi: 10.1016/S0169-5347(02)00041-1
CrossRef Full Text/Google Scholar
Taylor, HR, Dussex, N., e van Heezik, Y. (2017). Colmare il divario di genetica della conservazione identificando gli ostacoli all’implementazione per gli operatori della conservazione. Glob. Ecol. Conserv. 10, 231–242. doi: 10.1016 / j.gecco.2017.04.001
CrossRef Full Text/Google Scholar
Thomson, S.A., Pyle, R. L., Ahyong, S. T., Alonso-Zarazaga, M., Ammirati, J., Araya, J. F., et al. (2018). La tassonomia basata sulla scienza è necessaria per la conservazione globale. PLoS Biol. 16: e2005075. doi: 10.1371 / rivista.pbio.2005075
PubMed Abstract | CrossRef Full Text/Google Scholar
Vieites, DR, Wollenberg, KC, Andreonec, F., Köhler, J., Glaw, F. e Vences, M. (2009). Vasta sottovalutazione della biodiversità del Madagascar evidenziata da un inventario integrativo di anfibi. Proc. Natl. Acad. Sic. USA 106, 8267-8272. doi: 10.1073 / pnas.0810821106
CrossRef Full Text
Vogler, A. P., e de Salle, R. (1994). Diagnosi delle unità di Gestione della conservazione. Svantaggio. Biol. 8, 354–363. doi: 10.1046 / j. 1523-1739. 1994.08020354.x
CrossRef Full Text/Google Scholar
Waples, RS (1991). Salmone del Pacifico, Oncorhynchus spp., e la definizione di” specie ” sotto l’Endangered Species Act. Guastare. Pesce. Apoc 53, 11-22.
Google Scholar
Waples, RS (1995). “Evolutionarily significant units and the conservation of biological diversity under the Endangered Species Act,” in Evolution and the Aquatic Ecosystem: Defining Unique Units in Population Conservation, eds J. L. Nielsen (Bethesda, MD: American Fisheries Society), 8-27.il nostro sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. (2001). Caratterizzare la diversità nel salmone dal nord-ovest del Pacifico. J. Pesce Biol. 59, 1–41. doi: 10.1111 / j. 1095-8649. 2001.tb01376.il sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. Un racconto di due atti: specie in via di estinzione che elencano le pratiche in Canada e negli Stati Uniti. BioScience 63, 723-734. doi: 10.1093 / bioscience / 63.9.723
CrossRef Full Text/Google Scholar
Waples, RS, Punt, AE e Cope, JM (2008). Integrare i dati genetici nella gestione delle risorse marine: come possiamo farlo meglio? Pesce 9, 423-449. doi: 10.1111 / j. 1467-2979. 2008.00303.il nostro sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. Tassonomia strategica in un paesaggio biodiverse: un nuovo approccio per massimizzare i risultati di conservazione per la flora rara e poco conosciuta. Biodivers. Conserv. 24, 17–32. doi: 10.1007 / s10531-014-0785-4
CrossRef Full Text/Google Scholar
Whitlock, R. (2014). Relazioni tra diversità genetica adattativa e neutra e struttura e funzionamento ecologico: una meta-analisi. J. Ecol. 102, 857–872. doi: 10.1111 | 1365-2745.12240
PubMed Abstract/CrossRef Full Text/Google Scholar
Wiley, E. O. (1978). Il concetto di specie evolutiva riconsiderato. Syst. Biol. 27, 17–26. doi: 10.2307/2412809
CrossRef Full Text/Google Scholar
Wilson, E. O. (2017). La ricerca sulla biodiversità richiede più stivali sul terreno. NAT. Ecol. Evol. 1, 1590–1591. doi: 10.1038 / s41559-017-0360-y
PubMed Abstract | CrossRef Full Text/Google Scholar
Yang, ZH e Rannala, B. (2014). Delimitazione delle specie non guidate utilizzando i dati della sequenza del DNA da loci multipli. Mol. Biol. Evol. 31, 3125–3135. doi: 10.1093/molbev/msu279
PubMed Abstract | CrossRef Full Text/Google Scholar
Yeates, D. K., Seago, A., Nelson, L., Cameron, SL, Joseph, L. e Trueman, WH (2011). Tassonomia integrativa o tassonomia iterativa? Sys. Ent. 36, 209–217. doi: 10.1111/j.1365-3113.2010.00558.x
CrossRef Full Text | Google Scholar